
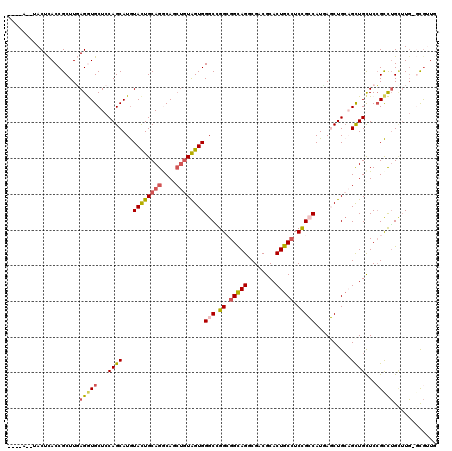
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 5,462,351 – 5,462,489 |
| Length | 138 |
| Max. P | 0.820009 |

| Location | 5,462,351 – 5,462,462 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.85 |
| Mean single sequence MFE | -50.35 |
| Consensus MFE | -30.82 |
| Energy contribution | -31.02 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.729048 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5462351 111 + 22224390 --------ACUCACCGCUUCAGGUGCUCCAGCAUGUACUGCAGACAGCUGUGGUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUGAGCUGGCGCUGCUCGGCCUGCUUG-CCGGUG --------...((((((..(((((((.(((((...(((..(((....)))..)))((((((.(((((((...)).)))))))))))....))))).))......)))))...)-.))))) ( -54.80) >DroVir_CAF1 2216 119 + 1 GCAAUAGGACUCACCGCUUGAGAUGCUCCAGCAUAUACUGCAGGCAACUGUAGUGGGCCGGCGGCAGGCGACGCACUGCGUCCGCCAUUAGCUGCAGCUGCUCCGUUGGCAUU-GCAUUG (((((........(((((.(((...))).)))....(((((((....)))))))))((((((((..((((((((...)))).))))..((((....))))..)))))))))))-)).... ( -53.30) >DroPse_CAF1 210 115 + 1 ----AACUACUCACCGCUUGAGGUGCUCCAGCAUGUACUGCAGGCAGCUGUAGUGGGCCGGAGGCAGACGACGCACUGCCUCUGACAUCAUCUGCAGCUGCUCCGCCUGUUUG-GUGUUC ----.......((((((...(.((((....)))).)...)).((((((((((((((..(((((((((........)))))))))...))).)))))))))))..........)-)))... ( -50.60) >DroGri_CAF1 483 117 + 1 GCGCU--AACUCACCGCUUGAGAUGCUCCAGCAUGUAUUGCAGGCAACUAUAGUGGGCAGGCGGUAGUCGACGCACUGCCUCCGCCAUGAGCUGCAGCUGUUCCGCUUGCUUG-GCGUUG (((((--((((((..(((((..((((........))))..))))).......(..((((((((........))).)))))..)....))))..(((((......)).))))))-)))).. ( -43.00) >DroEre_CAF1 1179 113 + 1 ----A--CACUUACCGCUUCAGGUGCUCCAGCAUGUACUGCAGACACGAGUGGUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUGAGCUGCCGUUGCUCCGCCUGAUUG-CUGGUG ----.--....((((((.(((((((....((((...((.((((.(((.....)))((((((.(((((((...)).))))))))))).....)))).)))))).)))))))..)-).)))) ( -49.60) >DroPer_CAF1 210 116 + 1 ----AACUACUCACCGCUUGAGGUGCUCCAGCAUGUACUGCAGGCAGCUGUAGUGGGCCGGAGGCAGACGACGCACUGCCUCUGACAUCAUCUGCAGCUGCUCCGCCUGUUUGAGUGUUC ----...((((((..((...(.((((....)))).)...)).((((((((((((((..(((((((((........)))))))))...))).))))))))))).........))))))... ( -50.80) >consensus ____A__UACUCACCGCUUGAGGUGCUCCAGCAUGUACUGCAGGCAGCUGUAGUGGGCCGGCGGCAGGCGACGCACUGCCUCCGCCAUGAGCUGCAGCUGCUCCGCCUGCUUG_GCGUUG ....................(((((...((((...((((((((....))))))))(((.((.(((((........))))).)))))..........))))...)))))............ (-30.82 = -31.02 + 0.20)


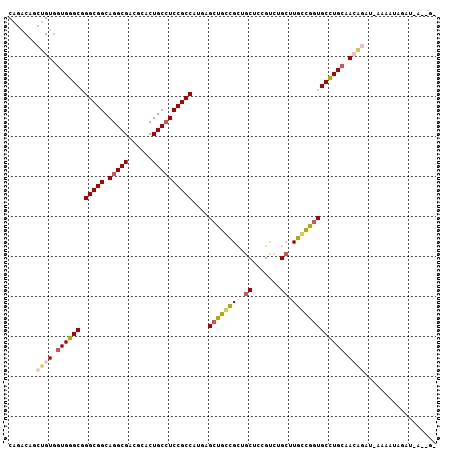
| Location | 5,462,383 – 5,462,489 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 80.90 |
| Mean single sequence MFE | -43.73 |
| Consensus MFE | -35.12 |
| Energy contribution | -35.65 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.820009 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5462383 106 + 22224390 CAGACAGCUGUGGUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUGAGCUGGCGCUGCUCGGCCUGCUUGCCGGUGCCUGCAACAGAUCAAAAUAGAU-AGAGA ..((...((((.(..(((.(((((.(((((......))))))))))....(((((((..((.......)).))))))))))..).)))).)).........-..... ( -45.50) >DroVir_CAF1 2256 103 + 1 CAGGCAACUGUAGUGGGCCGGCGGCAGGCGACGCACUGCGUCCGCCAUUAGCUGCAGCUGCUCCGUUGGCAUUGCAUUGCCUGCAAAGAA----AUCAAAUUAGCGU (((((((.((((((..((((((((..((((((((...)))).))))..((((....))))..))))))))))))))))))))).......----............. ( -43.30) >DroSec_CAF1 2200 102 + 1 CAGACAGCUGUGGUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUGAGCUGGCGCUGCUCCGUCUGCUUGCCGGUGCCUGCAACAGAUAAAAAUAGAU-A---- .......((((.(..(((.(((((.(((((......))))))))))....(((((((..((.......)).))))))))))..).))))............-.---- ( -44.80) >DroSim_CAF1 1941 102 + 1 CAGACAGCUGUGGUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUAAGCUGGCGCUGCUCCGUCUGCUUGCCGGUGCUUGCAACAGAUAAAAAUAAAU-A---- .......((((.(..(((.(((((.(((((......))))))))))....(((((((..((.......)).))))))))))..).))))............-.---- ( -43.10) >DroEre_CAF1 1213 100 + 1 CAGACACGAGUGGUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUGAGCUGCCGUUGCUCCGCCUGAUUGCUGGUGCCUGCAACAGAUGACA---GAU----GA (((.(((.((..((((((((((((.(((((......))))))))))..((((.......))))))))).))..)).))).)))............---...----.. ( -45.10) >DroYak_CAF1 1271 93 + 1 CAGACACCUGUGAUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUCAGCUGCCGCUGCUCCGUCUGCUUGGCGGUGCCUGCAAGAGA---------AU----U- (((.((((..((((((.((((.(((((((...)).)))))))))))))))...((((..((.......)).)))))))).))).......---------..----.- ( -40.60) >consensus CAGACAGCUGUGGUGGGCGGGCGGCAGGCGACGCACUGCCUCCGCCAUGAGCUGCCGCUGCUCCGUCUGCUUGCCGGUGCCUGCAACAGAU_AAAAUAGAU_A__G_ .......((((.((((((.(((((.(((((......))))))))))....(((((((..((.......)).))))))))))))).)))).................. (-35.12 = -35.65 + 0.53)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:23:38 2006