
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 5,337,436 – 5,337,556 |
| Length | 120 |
| Max. P | 0.590058 |

| Location | 5,337,436 – 5,337,556 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.67 |
| Mean single sequence MFE | -41.92 |
| Consensus MFE | -31.47 |
| Energy contribution | -32.52 |
| Covariance contribution | 1.05 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.538108 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5337436 120 + 22224390 CAAAAUACUCGAUCGUUAAUGGCGCCCGCACCUGCACCAACACCAACGAGACGGUCCUCUAGUAAUUGGUGAUGUUGGUGCCGCGGCGGGGCAGCAGCGACCAUCGUCGCAGCUCCACUA .....................((.(((((..(.(((((((((((((..(((......))).....)))))...)))))))).)..)))))))(((.(((((....))))).)))...... ( -43.50) >DroVir_CAF1 78429 120 + 1 CCAAGUACUCUAUUGUGAAUGGCCCCCGCACCUGCACCAACACCAACGAGACUAUCCUCUAGUAAUUGGUGAUGUUGGUGCCGCGGCGCGGCAGCAGCGACCAGCGUCGUGGCACCGCCG .....(((......)))...(((....((....))..((.((((((....((((.....))))..)))))).))..((((((((((((((((......).)).)))))))))))))))). ( -45.60) >DroGri_CAF1 70619 105 + 1 CCAAGUACUCGAUCGUGAAUGGCCCACGCACCUGCACCAACACCAAUGAGACUGUCCUCUAGUAAUUGGUGAUGUUGGUGCCGCGGCGUGGCAGUA---------------GCACUGCCG (((..(((......)))..)))...((((..(.((((((((((((((...((((.....)))).))))))...)))))))).)..))))(((((..---------------...))))). ( -41.30) >DroWil_CAF1 11156 120 + 1 CGAAAUACUCAAUUGUCAAUGGACCACGCACAUGCACCAACACCAAUGAGACGGUCCUCUAGUAAUUGGUAAUGCUGGUGCCACGGCGCGGAAGCAGUGAUCAUCAUCGGAGCACCACCA .....(((.((((((..(..(((((..((....)).....((....))....)))))..)..))))))))).((.((((((..((..((....)).(((.....)))))..)))))).)) ( -35.40) >DroMoj_CAF1 101141 120 + 1 CGAAGUACUCGAUCGUGAAUGGCCCACGCACCUGCACCAACACCAACGAGACUGUCCUCUAGUAAUUGGUGAUGUUGGUGCCGCGGCGUGGCAGCACCGAUCAGCAUCGCAGCGCAGCGG ..........(((((((....(((.((((..(.(((((((((.((.(((.((((.....))))..))).)).))))))))).)..)))))))..)).))))).....(((......))). ( -44.20) >DroAna_CAF1 68180 117 + 1 CCAAGUACUCGAUCGUCAACGGCGCCCGCACCUGCACCAACACCAACGAGACGGUCCUCUAGUAAUUGGUGAUGUUGGUGCCGCGGCGGGGCAGCAGCGACCACCGUCGCA---CCACCU ....((.......((....))((.(((((..(.(((((((((((((..(((......))).....)))))...)))))))).)..))))))).)).(((((....))))).---...... ( -41.50) >consensus CCAAGUACUCGAUCGUGAAUGGCCCACGCACCUGCACCAACACCAACGAGACGGUCCUCUAGUAAUUGGUGAUGUUGGUGCCGCGGCGCGGCAGCAGCGACCAGCGUCGCAGCACCACCG .....................((.(((((..(.(((((((((((((.(((......)))......)))))...)))))))).)..))))))).((.(((((....))))).))....... (-31.47 = -32.52 + 1.05)



| Location | 5,337,436 – 5,337,556 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.67 |
| Mean single sequence MFE | -48.15 |
| Consensus MFE | -33.47 |
| Energy contribution | -33.28 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.590058 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 5337436 120 - 22224390 UAGUGGAGCUGCGACGAUGGUCGCUGCUGCCCCGCCGCGGCACCAACAUCACCAAUUACUAGAGGACCGUCUCGUUGGUGUUGGUGCAGGUGCGGGCGCCAUUAACGAUCGAGUAUUUUG (((((((((.(((((....))))).)))((((((((...((((((((((((..........((((....))))..)))))))))))).)))).)))).))))))................ ( -54.40) >DroVir_CAF1 78429 120 - 1 CGGCGGUGCCACGACGCUGGUCGCUGCUGCCGCGCCGCGGCACCAACAUCACCAAUUACUAGAGGAUAGUCUCGUUGGUGUUGGUGCAGGUGCGGGGGCCAUUCACAAUAGAGUACUUGG (((((((((((......))).))))))))(((((((...((((((((((((.(....((((.....))))...).)))))))))))).)))))))(((..((((......)))).))).. ( -52.90) >DroGri_CAF1 70619 105 - 1 CGGCAGUGC---------------UACUGCCACGCCGCGGCACCAACAUCACCAAUUACUAGAGGACAGUCUCAUUGGUGUUGGUGCAGGUGCGUGGGCCAUUCACGAUCGAGUACUUGG .(((((...---------------..))))).((((...((((((((...((((((.....((((....)))))))))))))))))).))))(((((.....)))))............. ( -39.00) >DroWil_CAF1 11156 120 - 1 UGGUGGUGCUCCGAUGAUGAUCACUGCUUCCGCGCCGUGGCACCAGCAUUACCAAUUACUAGAGGACCGUCUCAUUGGUGUUGGUGCAUGUGCGUGGUCCAUUGACAAUUGAGUAUUUCG ..(..((((((....(((..(((.((...((((((((((.(((((((...((((((.....((((....))))))))))))))))))))).))))))..)).)))..)))))))))..). ( -43.00) >DroMoj_CAF1 101141 120 - 1 CCGCUGCGCUGCGAUGCUGAUCGGUGCUGCCACGCCGCGGCACCAACAUCACCAAUUACUAGAGGACAGUCUCGUUGGUGUUGGUGCAGGUGCGUGGGCCAUUCACGAUCGAGUACUUCG .(((......))).(((((((((.....(((.((((((.((((((((((((..........((((....))))..))))))))))))..))).))))))......))))).))))..... ( -45.70) >DroAna_CAF1 68180 117 - 1 AGGUGG---UGCGACGGUGGUCGCUGCUGCCCCGCCGCGGCACCAACAUCACCAAUUACUAGAGGACCGUCUCGUUGGUGUUGGUGCAGGUGCGGGCGCCGUUGACGAUCGAGUACUUGG ....((---(((..((((.(((((.((.((((((((...((((((((((((..........((((....))))..)))))))))))).)))).)))))).).)))).)))).)))))... ( -53.90) >consensus CGGCGGUGCUGCGACGAUGGUCGCUGCUGCCACGCCGCGGCACCAACAUCACCAAUUACUAGAGGACAGUCUCGUUGGUGUUGGUGCAGGUGCGGGGGCCAUUCACGAUCGAGUACUUGG ..((((((...((((....)))).))))))(((((....((((((((...((((((.....((((....))))))))))))))))))....)))))........................ (-33.47 = -33.28 + -0.19)

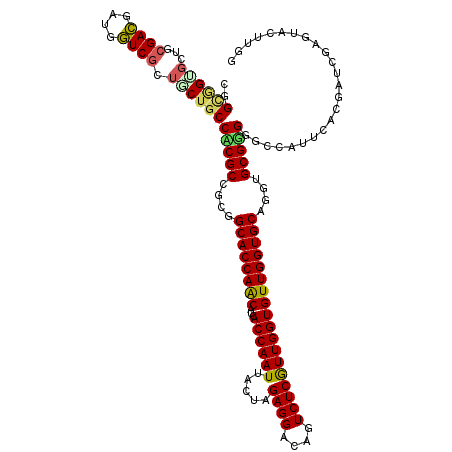
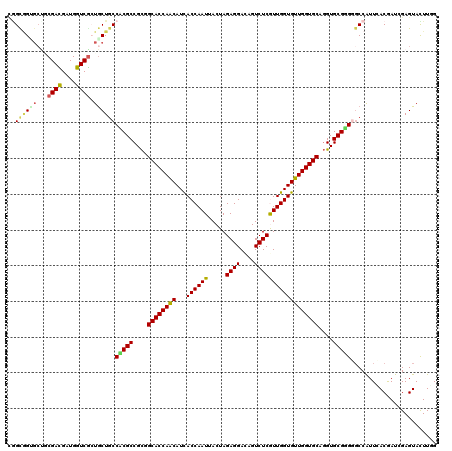
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:22:37 2006