
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,880,547 – 4,880,658 |
| Length | 111 |
| Max. P | 0.994580 |

| Location | 4,880,547 – 4,880,658 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 90.06 |
| Mean single sequence MFE | -25.58 |
| Consensus MFE | -20.17 |
| Energy contribution | -19.65 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.43 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.552495 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4880547 111 + 22224390 AUUAAUGGCUGCAUUUAUAUCGUGUUUUUAGAUCAGCCAACUAACAUUUUCGAACUGUAGCUCAUCAAUAUAAUUUAAACGUCCAGCUCGGGGCGAUCCCCGAAUUUAAAA (((.(((((((((.((.....(((((....(......)....))))).....)).)))))).))).)))..................((((((....))))))........ ( -21.10) >DroSec_CAF1 31836 106 + 1 ---AAUGGCUGCAUUUAUAUCGUGUUUUUAGAUCAGCCAACCAACAUUUUCGGGCUGUAGCUCAUCAAUAUAAUUUAAACGGCCAGCUCGGGGCGAUCCCCGAAUUUA--A ---..((((((..((((.((..((((...((..(((((..............)))))...))....))))..)).))))))))))..((((((....)))))).....--. ( -28.64) >DroSim_CAF1 33038 109 + 1 AUUAAUGGCUGCAUUUAUAUCGUGUUUUUAGAUCAGCCAACCAACAUUUUCGGGCUGUAGCUCAUCAAUAUAAUUUAAACGGCCAGCUCGGGGCGAUCCCCGAAUUUA--A .....((((((..((((.((..((((...((..(((((..............)))))...))....))))..)).))))))))))..((((((....)))))).....--. ( -28.64) >DroEre_CAF1 34750 108 + 1 AUUAAUGGCUGCAUUUAUAUCGUGAUUUUAGAUCAGCCAG-CAACAUUUUUGGGUUGUAGCUCAUCAAUAUAAUUUAAACAGCCAGCUCGGGGCGAUCCCCAAGUUUA--A .....(((((((((.......)))......(((.(((..(-((((........))))).))).))).............))))))(((.((((....)))).)))...--. ( -26.90) >DroYak_CAF1 32559 108 + 1 AUUAAUAGCUGCAUUUAUAUUGUGAUUUUAGAUCAGCCAG-CAACAUUUUUGGGUUGUAGCUCAUCAAUGUAAUUUAAACGACGAGCUCGGGGCGAUACCCAAGUUAA--A .......((((.(((((...........)))))))))...-..(((((..(((((....)))))..))))).............((((.(((......))).))))..--. ( -22.60) >consensus AUUAAUGGCUGCAUUUAUAUCGUGUUUUUAGAUCAGCCAACCAACAUUUUCGGGCUGUAGCUCAUCAAUAUAAUUUAAACGGCCAGCUCGGGGCGAUCCCCGAAUUUA__A .....((((((.(((((...........))))))))))).........(((((((((..........................))))))(((......))))))....... (-20.17 = -19.65 + -0.52)

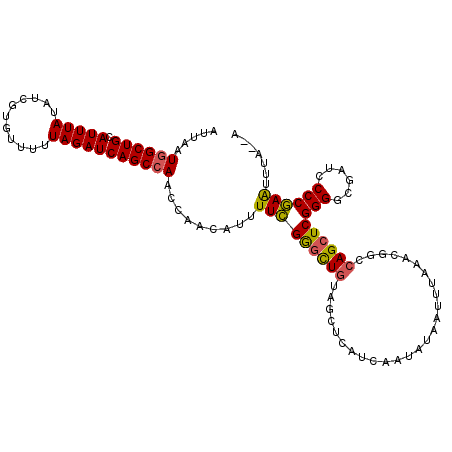
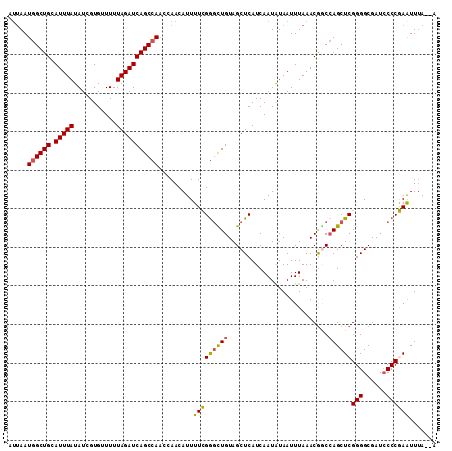
| Location | 4,880,547 – 4,880,658 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 90.06 |
| Mean single sequence MFE | -29.08 |
| Consensus MFE | -24.50 |
| Energy contribution | -23.98 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.84 |
| SVM decision value | 2.49 |
| SVM RNA-class probability | 0.994580 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4880547 111 - 22224390 UUUUAAAUUCGGGGAUCGCCCCGAGCUGGACGUUUAAAUUAUAUUGAUGAGCUACAGUUCGAAAAUGUUAGUUGGCUGAUCUAAAAACACGAUAUAAAUGCAGCCAUUAAU .......(((((((....))))((((((...(((((...........)))))..)))))))))...((((((.(((((......................))))))))))) ( -26.95) >DroSec_CAF1 31836 106 - 1 U--UAAAUUCGGGGAUCGCCCCGAGCUGGCCGUUUAAAUUAUAUUGAUGAGCUACAGCCCGAAAAUGUUGGUUGGCUGAUCUAAAAACACGAUAUAAAUGCAGCCAUU--- .--....(((((((....))))))).((((.((.....((((((((((.(((((((((........))))..))))).))).........)))))))..)).))))..--- ( -29.30) >DroSim_CAF1 33038 109 - 1 U--UAAAUUCGGGGAUCGCCCCGAGCUGGCCGUUUAAAUUAUAUUGAUGAGCUACAGCCCGAAAAUGUUGGUUGGCUGAUCUAAAAACACGAUAUAAAUGCAGCCAUUAAU .--....(((((((....))))))).((((.((.....((((((((((.(((((((((........))))..))))).))).........)))))))..)).))))..... ( -29.30) >DroEre_CAF1 34750 108 - 1 U--UAAACUUGGGGAUCGCCCCGAGCUGGCUGUUUAAAUUAUAUUGAUGAGCUACAACCCAAAAAUGUUG-CUGGCUGAUCUAAAAUCACGAUAUAAAUGCAGCCAUUAAU .--....(((((((....))))))).(((((((.....((((((((...(((((((((........))))-.)))))(((.....))).))))))))..)))))))..... ( -35.80) >DroYak_CAF1 32559 108 - 1 U--UUAACUUGGGUAUCGCCCCGAGCUCGUCGUUUAAAUUACAUUGAUGAGCUACAACCCAAAAAUGUUG-CUGGCUGAUCUAAAAUCACAAUAUAAAUGCAGCUAUUAAU .--.....((((((.(((...)))((((((((............))))))))....))))))....((((-.((((((......................)))))).)))) ( -24.05) >consensus U__UAAAUUCGGGGAUCGCCCCGAGCUGGCCGUUUAAAUUAUAUUGAUGAGCUACAGCCCGAAAAUGUUGGUUGGCUGAUCUAAAAACACGAUAUAAAUGCAGCCAUUAAU .......(((((((....))))))).((((((((((.(((.....(((.(((((((((........))))..))))).))).........))).))))))..))))..... (-24.50 = -23.98 + -0.52)

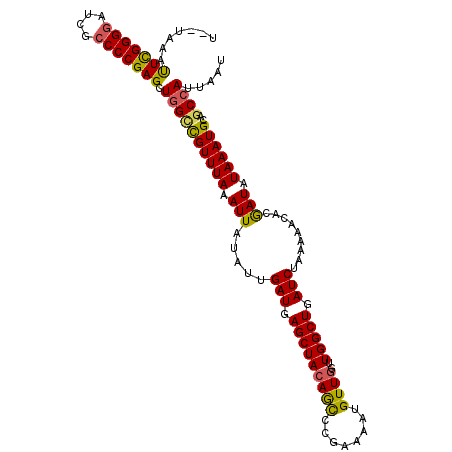
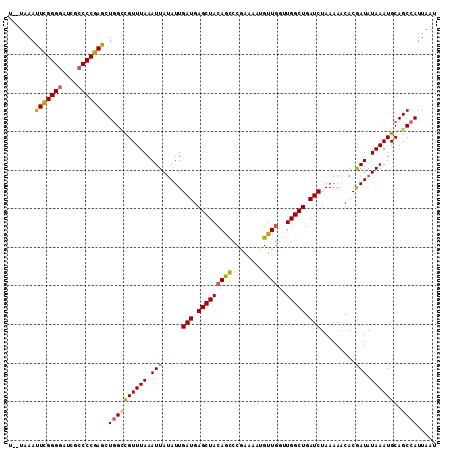
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:18:22 2006