
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,440,622 – 4,440,934 |
| Length | 312 |
| Max. P | 0.999843 |

| Location | 4,440,622 – 4,440,742 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -47.20 |
| Consensus MFE | -44.16 |
| Energy contribution | -44.48 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.02 |
| Mean z-score | -3.70 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.926290 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4440622 120 + 22224390 CAUCUUGCAGCCAUUUCUUCGGACUCCGCUACAGAAAGACAGACAGUCGGUUUGAGUUGAGUCGGGUUGCUCCACCCGCUCCGCUUCCGUUGUGGCUCGAAUUUAAAUGGCAGCAAGAAA ..((((((.(((((((.(((((((((.(((.((((..(((.....)))..))))))).)))))((((......))))...((((.......))))..))))...))))))).)))))).. ( -47.00) >DroSec_CAF1 42308 120 + 1 CAUCUUGCAGCCAUUUCUUCGGACUCCGCUACAGAAAGACAGACAGUCGGUUUGAGUUGAGUCGGGUUGCUCCACCCGCUCCGCUUCCGUUGUGGCUCGAAUUUAAAUGGCAGCAAGAAA ..((((((.(((((((.(((((((((.(((.((((..(((.....)))..))))))).)))))((((......))))...((((.......))))..))))...))))))).)))))).. ( -47.00) >DroEre_CAF1 44144 120 + 1 CAUCUUGCAGCCAUUUCUUCGGACUCCGCAACAGAAAGACAGACAGUCGGUUUGAGUUGAGUCGGGUUGCUCCACCCGCUUCGCUUCCGUUGUGGCUCGAAUUUAAAUGGCAGCAAGAAA ..((((((.(((((((.(((((...(((((((.....(((.....)))((...((((.(((.(((((......)))))))).))))))))))))).)))))...))))))).)))))).. ( -48.70) >DroYak_CAF1 42997 120 + 1 CAUCUUGCAGCCAUUUCUUCGGACUCAGCUACAGAAAGACAGACAGUCGGUUUGAGUUGAGUCGGGUUGCUCCACCCGCUCCGCUUCCGUUGUGGCUCGAAUUUAAAUGGCAGCAAAAAA ....((((.(((((((.(((((((((((((.((((..(((.....)))..)))))))))))))((((......))))...((((.......))))..))))...))))))).)))).... ( -46.10) >consensus CAUCUUGCAGCCAUUUCUUCGGACUCCGCUACAGAAAGACAGACAGUCGGUUUGAGUUGAGUCGGGUUGCUCCACCCGCUCCGCUUCCGUUGUGGCUCGAAUUUAAAUGGCAGCAAGAAA ..((((((.(((((((.(((((.....(((((((...(((.....)))((...((((.(((.(((((......)))))))).)))))).))))))))))))...))))))).)))))).. (-44.16 = -44.48 + 0.31)



| Location | 4,440,622 – 4,440,742 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -47.68 |
| Consensus MFE | -44.29 |
| Energy contribution | -44.60 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.69 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.917162 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4440622 120 - 22224390 UUUCUUGCUGCCAUUUAAAUUCGAGCCACAACGGAAGCGGAGCGGGUGGAGCAACCCGACUCAACUCAAACCGACUGUCUGUCUUUCUGUAGCGGAGUCCGAAGAAAUGGCUGCAAGAUG ..((((((.(((((((...((((..((.(.(((((((..((((((((......))))).)))..).......(((.....))).)))))).).))....)))).))))))).)))))).. ( -47.80) >DroSec_CAF1 42308 120 - 1 UUUCUUGCUGCCAUUUAAAUUCGAGCCACAACGGAAGCGGAGCGGGUGGAGCAACCCGACUCAACUCAAACCGACUGUCUGUCUUUCUGUAGCGGAGUCCGAAGAAAUGGCUGCAAGAUG ..((((((.(((((((...((((..((.(.(((((((..((((((((......))))).)))..).......(((.....))).)))))).).))....)))).))))))).)))))).. ( -47.80) >DroEre_CAF1 44144 120 - 1 UUUCUUGCUGCCAUUUAAAUUCGAGCCACAACGGAAGCGAAGCGGGUGGAGCAACCCGACUCAACUCAAACCGACUGUCUGUCUUUCUGUUGCGGAGUCCGAAGAAAUGGCUGCAAGAUG ..((((((.(((((((...((((..((.(((((((((.((..(((((......)))))..))..........(((.....)))))))))))).))....)))).))))))).)))))).. ( -49.70) >DroYak_CAF1 42997 120 - 1 UUUUUUGCUGCCAUUUAAAUUCGAGCCACAACGGAAGCGGAGCGGGUGGAGCAACCCGACUCAACUCAAACCGACUGUCUGUCUUUCUGUAGCUGAGUCCGAAGAAAUGGCUGCAAGAUG ..((((((.(((((((...((((..((((.(((((((..((((((((......))))).)))..).......(((.....))).)))))).).)).)..)))).))))))).)))))).. ( -45.40) >consensus UUUCUUGCUGCCAUUUAAAUUCGAGCCACAACGGAAGCGGAGCGGGUGGAGCAACCCGACUCAACUCAAACCGACUGUCUGUCUUUCUGUAGCGGAGUCCGAAGAAAUGGCUGCAAGAUG ..((((((.(((((((...((((..((.(.(((((((..((((((((......))))).)))..........(((.....)))))))))).).))....)))).))))))).)))))).. (-44.29 = -44.60 + 0.31)

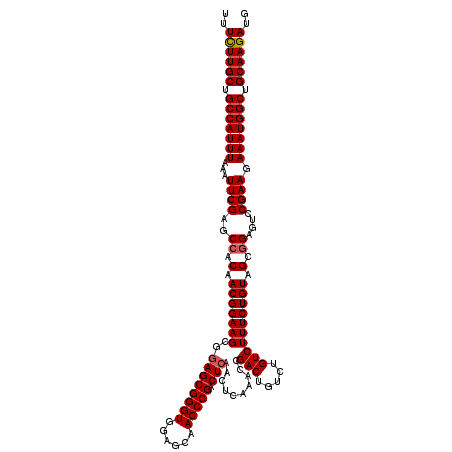
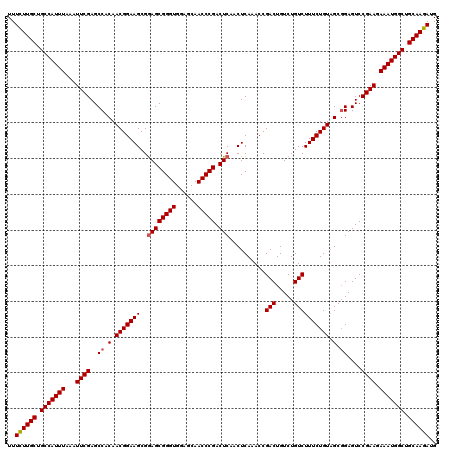
| Location | 4,440,742 – 4,440,854 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.68 |
| Mean single sequence MFE | -33.50 |
| Consensus MFE | -26.32 |
| Energy contribution | -26.60 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.76 |
| Structure conservation index | 0.79 |
| SVM decision value | 3.12 |
| SVM RNA-class probability | 0.998490 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4440742 112 + 22224390 AUACAGCCACAUGGAUAUACAUAUGUAUGUAUGUAUCUACACUAUAGA--GAGAUGAAGAUUCAGAUAGGUAUCUGUAUCUGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGA ....(((((((..((((((.((((.(((...((.((((.((((.....--.)).)).)))).)).))).)))).))))))))))------)))(((((((.....)))))))........ ( -35.40) >DroSec_CAF1 42428 106 + 1 AUACA--CACAUGGAUAUACAUAUGUA----UGUGUCUACACUAUAGA--GAGAUGAAGAUUCAGAUAGGUAUCUGUAUCUGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGA .....--..((((((((((.((((.((----(.(((((.((((.....--.)).)).)))..)).))).)))).))))))))))------..((((((((.....))))))))....... ( -30.80) >DroSim_CAF1 43615 108 + 1 AUACAGCCACAUGGAUAUACAUAUGUA----UGUGUCUACACUAUAGA--GAGAUGAAGAUUCAGAUAGGUAUCUGUAUCUGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGA ....(((((((..((((((.((((.((----(.(((((.((((.....--.)).)).)))..)).))).)))).))))))))))------)))(((((((.....)))))))........ ( -35.50) >DroEre_CAF1 44264 110 + 1 AUACAGCCACAAAGAUAUAUGUAUGUAUGUACGUAUCUACACUAUAG----AGAUGAAGAUUCAGAUAGGUAUCUGUAUCUGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGA ....((((((..((((((..((((....)))))))))).........----......((((.(((((....))))).)))))))------)))(((((((.....)))))))........ ( -34.40) >DroYak_CAF1 43117 112 + 1 AUACAUUCACAUAGAUAUAUGUAUGU--------AUCUACACUAUAGAGAGAGAUGAAGAUUCAGAUAGGUAUCUGUAUCUGUGCCUGUGCCUGGUGCAUUUUGAAUGCACCAUUAAUGA ...(((...((((((((((.((((.(--------((((...((......)).((.......))))))).)))).))))))))))...)))..((((((((.....))))))))....... ( -31.40) >consensus AUACAGCCACAUGGAUAUACAUAUGUA____UGUAUCUACACUAUAGA__GAGAUGAAGAUUCAGAUAGGUAUCUGUAUCUGUG______GCUGGUGCAUUUUGAAUGCACCAUUAAUGA .........((((((((((.((((.((....((.((((.((((........)).)).)))).))..)).)))).))))))))))........((((((((.....))))))))....... (-26.32 = -26.60 + 0.28)


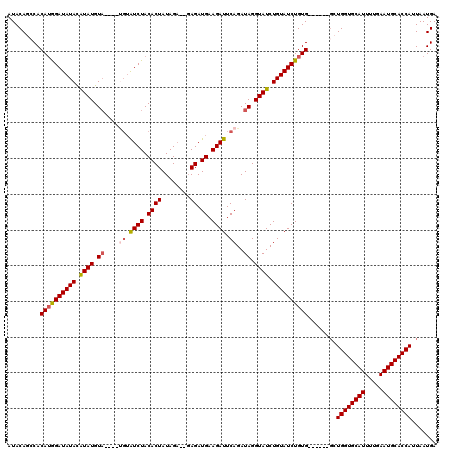
| Location | 4,440,742 – 4,440,854 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.68 |
| Mean single sequence MFE | -31.36 |
| Consensus MFE | -24.02 |
| Energy contribution | -23.94 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.04 |
| Mean z-score | -4.98 |
| Structure conservation index | 0.77 |
| SVM decision value | 2.96 |
| SVM RNA-class probability | 0.997906 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4440742 112 - 22224390 UCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC--UCUAUAGUGUAGAUACAUACAUACAUAUGUAUAUCCAUGUGGCUGUAU ........(((((((.....)))))))(((------(((((((.(((((....))))).)))).......--......(((...(((((((......)))))))...))))))))).... ( -34.50) >DroSec_CAF1 42428 106 - 1 UCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC--UCUAUAGUGUAGACACA----UACAUAUGUAUAUCCAUGUG--UGUAU .......((((((((.....))))))))..------...((((.(((((....))))).)))).......--......(((....)))(----((((((((......))))))--))).. ( -29.50) >DroSim_CAF1 43615 108 - 1 UCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC--UCUAUAGUGUAGACACA----UACAUAUGUAUAUCCAUGUGGCUGUAU ........(((((((.....)))))))(((------(((((((.(((((....))))).)))........--..(((((((((......----))))).))))......))))))).... ( -32.10) >DroEre_CAF1 44264 110 - 1 UCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCU----CUAUAGUGUAGAUACGUACAUACAUACAUAUAUCUUUGUGGCUGUAU ........(((((((.....)))))))(((------(((((((.(((((....))))).))).......----.....(((((......)))))...............))))))).... ( -30.90) >DroYak_CAF1 43117 112 - 1 UCAUUAAUGGUGCAUUCAAAAUGCACCAGGCACAGGCACAGAUACAGAUACCUAUCUGAAUCUUCAUCUCUCUCUAUAGUGUAGAU--------ACAUACAUAUAUCUAUGUGAAUGUAU .......((((((((.....))))))))...(((..(((((((.(((((....))))).)))).................((((((--------(........))))))))))..))).. ( -29.80) >consensus UCAUUAAUGGUGCAUUCAAAAUGCACCAGC______CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC__UCUAUAGUGUAGAUACA____UACAUAUGUAUAUCCAUGUGGCUGUAU .(((((.((((((((.....))))))))...........((((.(((((....))))).)))).............)))))..............((((((......))))))....... (-24.02 = -23.94 + -0.08)

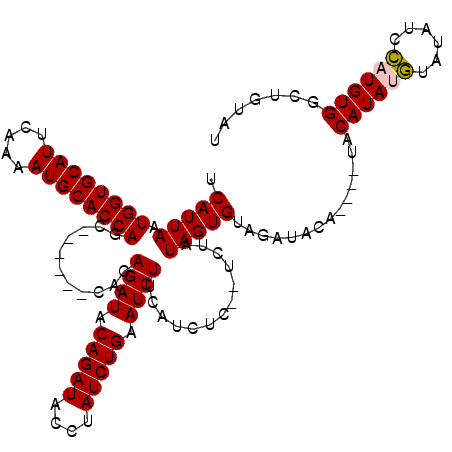
| Location | 4,440,782 – 4,440,894 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.83 |
| Mean single sequence MFE | -31.02 |
| Consensus MFE | -28.60 |
| Energy contribution | -28.60 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.92 |
| SVM decision value | 4.23 |
| SVM RNA-class probability | 0.999843 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4440782 112 - 22224390 CCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC--UCUAUAGU .....((((.....((((.(((..(.....)..)))..)))).....((((((((.....))))))))..------...((((.(((((....))))).))))...))))--........ ( -29.50) >DroSec_CAF1 42462 112 - 1 CCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC--UCUAUAGU .....((((.....((((.(((..(.....)..)))..)))).....((((((((.....))))))))..------...((((.(((((....))))).))))...))))--........ ( -29.50) >DroSim_CAF1 43651 112 - 1 CCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC--UCUAUAGU .....((((.....((((.(((..(.....)..)))..)))).....((((((((.....))))))))..------...((((.(((((....))))).))))...))))--........ ( -29.50) >DroEre_CAF1 44304 110 - 1 CCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACAGAUACAGAUACCUAUCUGAAUCUUCAUCU----CUAUAGU .....((((.....((((.(((..(.....)..)))..)))).....((((((((.....))))))))..------...((((.(((((....))))).))))...)))----)...... ( -29.50) >DroYak_CAF1 43149 120 - 1 CCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCUGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGGCACAGGCACAGAUACAGAUACCUAUCUGAAUCUUCAUCUCUCUCUAUAGU .....((((....((((..(((....))).))))(((((........((((((((.....))))))))....)))))..((((.(((((....))))).))))...)))).......... ( -37.10) >consensus CCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC______CACAGAUACAGAUACCUAUCUGAAUCUUCAUCUC__UCUAUAGU ..((..((......(((..(((....))).)))..))..))......((((((((.....))))))))...........((((.(((((....))))).))))................. (-28.60 = -28.60 + -0.00)


| Location | 4,440,820 – 4,440,934 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.44 |
| Mean single sequence MFE | -41.06 |
| Consensus MFE | -33.56 |
| Energy contribution | -33.76 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.66 |
| SVM RNA-class probability | 0.970371 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4440820 114 + 22224390 UGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGAUCAGGCGGGCAGCGAGUACGCAGACCUUGAUUCCCAAUGGUCGAGACUGGCGAGUAAUUGCCAGAGGAUCGCUGAGAUCU ....------..((((((((.....)))))))).....((((.((((..(.(((....))).(((((((.....))).))))....(((((((....)))))))..)..))))..)))). ( -44.40) >DroSec_CAF1 42500 114 + 1 UGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGAUCAGGCGGGCAGCGAGUACGCAGACCUUGAUUCCCAAUGGUCGAGACUGGCGAGUAAUUGCCAGAGGAUCGCUGAGAUCU ....------..((((((((.....)))))))).....((((.((((..(.(((....))).(((((((.....))).))))....(((((((....)))))))..)..))))..)))). ( -44.40) >DroSim_CAF1 43689 114 + 1 UGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGAUCAGGCGGGCAGCGAGUACGCAGACCUUGAUUCCCAAUGGUCGAGACUGGCGAAUAAUUGCCAGAGGAUCGCUGAGAUCU ....------..((((((((.....)))))))).....((((.((((..(.(((....))).(((((((.....))).))))....(((((((....)))))))..)..))))..)))). ( -42.10) >DroEre_CAF1 44340 107 + 1 UGUG------GCUGGUGCAUUUUGAAUGCACCAUUAAUGAUCAGGCGGGCAGCGAGUACGCAGACCUUGAUUCCCAAUGGUCGAG-------AGUAAUUGCCAGAGGAUCGCUGAAACCU ...(------((((((((((.....)))))))).....((((...(.(((((((....))).(((((((.....))).))))...-------......)))).)..)))))))....... ( -36.00) >DroYak_CAF1 43189 120 + 1 UGUGCCUGUGCCUGGUGCAUUUUGAAUGCACCAUUAAUGAUCAGGCAGGCAGCGAGUACGCAGACCUUGAUUCCCAAUGGUCGAGAGUGGAGAGUAAUUGCCAGAGGAUCGCAGAGACCC .....((.(((.((((((((.....)))))))).....((((...(.(((((((....))).(((((((.....))).))))................)))).)..))))))).)).... ( -38.40) >consensus UGUG______GCUGGUGCAUUUUGAAUGCACCAUUAAUGAUCAGGCGGGCAGCGAGUACGCAGACCUUGAUUCCCAAUGGUCGAGACUGGCGAGUAAUUGCCAGAGGAUCGCUGAGAUCU ............((((((((.....))))))))(((.(((((...(.(((((((....))).(((((((.....))).))))................)))).)..))))).)))..... (-33.56 = -33.76 + 0.20)


| Location | 4,440,820 – 4,440,934 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.44 |
| Mean single sequence MFE | -36.94 |
| Consensus MFE | -32.43 |
| Energy contribution | -31.87 |
| Covariance contribution | -0.56 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.88 |
| SVM decision value | 3.85 |
| SVM RNA-class probability | 0.999661 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4440820 114 - 22224390 AGAUCUCAGCGAUCCUCUGGCAAUUACUCGCCAGUCUCGACCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACA .((((...(((.....(((((........)))))((((((...)))))).....(((..(((....))).))))))..)))).....((((((((.....))))))))..------.... ( -36.90) >DroSec_CAF1 42500 114 - 1 AGAUCUCAGCGAUCCUCUGGCAAUUACUCGCCAGUCUCGACCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACA .((((...(((.....(((((........)))))((((((...)))))).....(((..(((....))).))))))..)))).....((((((((.....))))))))..------.... ( -36.90) >DroSim_CAF1 43689 114 - 1 AGAUCUCAGCGAUCCUCUGGCAAUUAUUCGCCAGUCUCGACCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACA .((((...(((.....(((((........)))))((((((...)))))).....(((..(((....))).))))))..)))).....((((((((.....))))))))..------.... ( -36.90) >DroEre_CAF1 44340 107 - 1 AGGUUUCAGCGAUCCUCUGGCAAUUACU-------CUCGACCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC------CACA .(((......((((..(.((((......-------...((((.(((.....))))))).(((....))))))).)...)))).....((((((((.....))))))))))------)... ( -33.60) >DroYak_CAF1 43189 120 - 1 GGGUCUCUGCGAUCCUCUGGCAAUUACUCUCCACUCUCGACCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCUGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGGCACAGGCACA (((((.....)))))...((((................((((.(((.....))))))).(((....)))))))((((((........((((((((.....))))))))....)))))).. ( -40.40) >consensus AGAUCUCAGCGAUCCUCUGGCAAUUACUCGCCAGUCUCGACCAUUGGGAAUCAAGGUCUGCGUACUCGCUGCCCGCCUGAUCAUUAAUGGUGCAUUCAAAAUGCACCAGC______CACA .((((...(((.......((((................((((.(((.....))))))).(((....))))))))))..)))).....((((((((.....))))))))............ (-32.43 = -31.87 + -0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:12:18 2006