
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,432,530 – 4,432,685 |
| Length | 155 |
| Max. P | 0.997831 |

| Location | 4,432,530 – 4,432,645 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.08 |
| Mean single sequence MFE | -37.30 |
| Consensus MFE | -26.76 |
| Energy contribution | -28.04 |
| Covariance contribution | 1.28 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.748837 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4432530 115 + 22224390 UGCGUGUUUAUCUUUAUUGGACUCGGUUGGAAAACCAUGUCACGUGCCCGAUUAAAAAUCCACCUUAUAU-----AAAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUU .........(((..(((..((.((((((....)))).((((..(((.(((((((.....(((((((....-----.)))))))..))))))).)))..))))...))))..)))..))). ( -38.90) >DroSec_CAF1 34879 115 + 1 UGCGUGUUUAUCUUUAUUGGUCUCCGUUGGAAAACCAUGUCACGUGCCCGAUUAUAAAUCCACCUUAUAU-----AAAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUU ...............((((((..(((((....)))..((((..(((.(((((((((...(((((((....-----.)))))))))))))))).)))..))))........)).)))))). ( -37.30) >DroSim_CAF1 36237 115 + 1 UGCGUGUUUAUCUUUAUUGGUCUCCGUUGGAAAACCAUGUCACGUGCCCGAUUAUAAAUCCACCUUAUAU-----AAAGUGGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUU ...............((((((..(((((....)))..((((..(((.((((((((..((((((.((....-----)).)))))))))))))).)))..))))........)).)))))). ( -32.30) >DroEre_CAF1 35824 120 + 1 UGCGUGUUUAUCUUUAUUGGCCUCGGCUGGUAAACCAUGUCACGUGCCCGAUUAUAAAUCCACCUUAUAUAUUAUAUUGGCGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUU .(((((......(((((..((....))..)))))......)))))..(((((((((...((.((.((((...))))..)).)))))))))))..((..(((....).))..))....... ( -33.50) >DroYak_CAF1 35027 115 + 1 UGCGUGUUUAUCUUUAUUGGGCUCGGUUGGAAAACCAUGCCACGUGUCCGAUUAUAAAUCCACCUUAUAU-----AGAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGUUACCGAUU ....(((((.(((......(((..((((....))))..)))..(((((((((((((...(((((((....-----.)))))))))))))))))))).))).))))).............. ( -44.50) >consensus UGCGUGUUUAUCUUUAUUGGUCUCGGUUGGAAAACCAUGUCACGUGCCCGAUUAUAAAUCCACCUUAUAU_____AAAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUU ...............(((((.......(((....)))((((..(((.(((((((((...(((((((..........)))))))))))))))).)))..))))............))))). (-26.76 = -28.04 + 1.28)



| Location | 4,432,530 – 4,432,645 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.08 |
| Mean single sequence MFE | -34.77 |
| Consensus MFE | -28.56 |
| Energy contribution | -30.00 |
| Covariance contribution | 1.44 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.94 |
| Structure conservation index | 0.82 |
| SVM decision value | 2.94 |
| SVM RNA-class probability | 0.997831 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4432530 115 - 22224390 AAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUUU-----AUAUAAGGUGGAUUUUUAAUCGGGCACGUGACAUGGUUUUCCAACCGAGUCCAAUAAAGAUAAACACGCA ...............(((((((..(.((((((.((..(((((((.-----....))))))).....)).)))))).)(.(((.(((((....))))).))))......)))))))..... ( -33.90) >DroSec_CAF1 34879 115 - 1 AAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUUU-----AUAUAAGGUGGAUUUAUAAUCGGGCACGUGACAUGGUUUUCCAACGGAGACCAAUAAAGAUAAACACGCA ...............(((((((..(.((((((.(((((((((((.-----....)))))))...)))).)))))).)......((((((((....)))))))).....)))))))..... ( -37.20) >DroSim_CAF1 36237 115 - 1 AAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCCACUUU-----AUAUAAGGUGGAUUUAUAAUCGGGCACGUGACAUGGUUUUCCAACGGAGACCAAUAAAGAUAAACACGCA ...............(((((((..(.((((((.((((.(((((((-----....)))))))...)))).)))))).)......((((((((....)))))))).....)))))))..... ( -34.50) >DroEre_CAF1 35824 120 - 1 AAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCGCCAAUAUAAUAUAUAAGGUGGAUUUAUAAUCGGGCACGUGACAUGGUUUACCAGCCGAGGCCAAUAAAGAUAAACACGCA ..(((((...........((((..(.((((((.(((((((((..............)))))...)))).)))))).)..))))(((....))))))))...................... ( -31.04) >DroYak_CAF1 35027 115 - 1 AAUCGGUAACAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUCU-----AUAUAAGGUGGAUUUAUAAUCGGACACGUGGCAUGGUUUUCCAACCGAGCCCAAUAAAGAUAAACACGCA ...............(((((((..(.((((((.((((((((((..-----.....))))))...)))).)))))).)(.(((.(((((....))))).))))......)))))))..... ( -37.20) >consensus AAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUUU_____AUAUAAGGUGGAUUUAUAAUCGGGCACGUGACAUGGUUUUCCAACCGAGACCAAUAAAGAUAAACACGCA ...............(((((((..(.((((((.(((((((((((..........)))))))...)))).)))))).)......(((((((......))))))).....)))))))..... (-28.56 = -30.00 + 1.44)

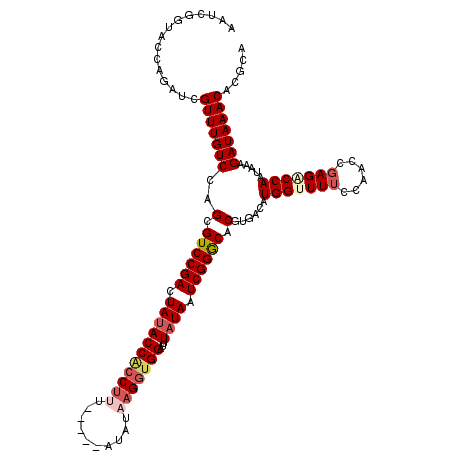
| Location | 4,432,570 – 4,432,685 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.70 |
| Mean single sequence MFE | -34.16 |
| Consensus MFE | -29.74 |
| Energy contribution | -30.66 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.907318 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4432570 115 + 22224390 CACGUGCCCGAUUAAAAAUCCACCUUAUAU-----AAAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUUGCACCUUUUAUGGAGCCGUAUACUCGGCGAUUAUCUCUGG ...(((((((((((.....(((((((....-----.)))))))..)))))))..((..(((....).))..)).......))))..........((((......))))............ ( -33.90) >DroSec_CAF1 34919 115 + 1 CACGUGCCCGAUUAUAAAUCCACCUUAUAU-----AAAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUUGCACCUUUUAUGGAGCCGUAUACUCGGCGAUUAUCUCUGG ...(((((((((((((...(((((((....-----.))))))))))))))))..((..(((....).))..)).......))))..........((((......))))............ ( -36.30) >DroSim_CAF1 36277 115 + 1 CACGUGCCCGAUUAUAAAUCCACCUUAUAU-----AAAGUGGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUUGCACCUUUUAUGGAGCCGUAUACUCGGCGAUUAUCUCUGG ...((((((((((((..((((((.((....-----)).))))))))))))))..((..(((....).))..)).......))))..........((((......))))............ ( -31.30) >DroEre_CAF1 35864 120 + 1 CACGUGCCCGAUUAUAAAUCCACCUUAUAUAUUAUAUUGGCGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUUGCACCUUUUAUGGAGCCGUAUACUCGGCGAUUAUCUCUGG ...(((((((((((((...((.((.((((...))))..)).)))))))))))..((..(((....).))..)).......))))..........((((......))))............ ( -30.00) >DroYak_CAF1 35067 115 + 1 CACGUGUCCGAUUAUAAAUCCACCUUAUAU-----AGAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGUUACCGAUUGCACCUUUUAUGGAGCCGUAUACUUGGCGAUUAUCUCUGU ((.(((((((((((((...(((((((....-----.))))))))))))))))))))))......(((((.......)))))..((......)).((((......))))............ ( -39.30) >consensus CACGUGCCCGAUUAUAAAUCCACCUUAUAU_____AAAGGUGGUAUAGUCGGACGCUGGACAAACGAUCUGGUACCGAUUGCACCUUUUAUGGAGCCGUAUACUCGGCGAUUAUCUCUGG ((.(((.(((((((((...(((((((..........)))))))))))))))).)))))......(((((.(....))))))..((......)).((((......))))............ (-29.74 = -30.66 + 0.92)



| Location | 4,432,570 – 4,432,685 |
|---|---|
| Length | 115 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.70 |
| Mean single sequence MFE | -35.31 |
| Consensus MFE | -31.22 |
| Energy contribution | -32.54 |
| Covariance contribution | 1.32 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.64 |
| SVM RNA-class probability | 0.996030 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4432570 115 - 22224390 CCAGAGAUAAUCGCCGAGUAUACGGCUCCAUAAAAGGUGCAAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUUU-----AUAUAAGGUGGAUUUUUAAUCGGGCACGUG ...(.(((((.(((((......)))).........(((((.....))))).....).)))))).(.((((((.((..(((((((.-----....))))))).....)).)))))).)... ( -35.90) >DroSec_CAF1 34919 115 - 1 CCAGAGAUAAUCGCCGAGUAUACGGCUCCAUAAAAGGUGCAAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUUU-----AUAUAAGGUGGAUUUAUAAUCGGGCACGUG ...(.(((((.(((((......)))).........(((((.....))))).....).)))))).(.((((((.(((((((((((.-----....)))))))...)))).)))))).)... ( -38.30) >DroSim_CAF1 36277 115 - 1 CCAGAGAUAAUCGCCGAGUAUACGGCUCCAUAAAAGGUGCAAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCCACUUU-----AUAUAAGGUGGAUUUAUAAUCGGGCACGUG ...(.(((((.(((((......)))).........(((((.....))))).....).)))))).(.((((((.((((.(((((((-----....)))))))...)))).)))))).)... ( -35.60) >DroEre_CAF1 35864 120 - 1 CCAGAGAUAAUCGCCGAGUAUACGGCUCCAUAAAAGGUGCAAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCGCCAAUAUAAUAUAUAAGGUGGAUUUAUAAUCGGGCACGUG ...(.(((((.(((((......)))).........(((((.....))))).....).)))))).(.((((((.(((((((((..............)))))...)))).)))))).)... ( -34.34) >DroYak_CAF1 35067 115 - 1 ACAGAGAUAAUCGCCAAGUAUACGGCUCCAUAAAAGGUGCAAUCGGUAACAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUCU-----AUAUAAGGUGGAUUUAUAAUCGGACACGUG ............(((........))).........((.((((.((((.....)))).)))))).(.((((((.((((((((((..-----.....))))))...)))).)))))).)... ( -32.40) >consensus CCAGAGAUAAUCGCCGAGUAUACGGCUCCAUAAAAGGUGCAAUCGGUACCAGAUCGUUUGUCCAGCGUCCGACUAUACCACCUUU_____AUAUAAGGUGGAUUUAUAAUCGGGCACGUG ...(.(((((.(((((......)))).........(((((.....))))).....).)))))).(.((((((.(((((((((((..........)))))))...)))).)))))).)... (-31.22 = -32.54 + 1.32)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:47 2006