
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,428,534 – 4,428,773 |
| Length | 239 |
| Max. P | 0.999837 |

| Location | 4,428,534 – 4,428,653 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.96 |
| Mean single sequence MFE | -25.46 |
| Consensus MFE | -20.00 |
| Energy contribution | -20.20 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.601203 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428534 119 + 22224390 UACUCGUAAGAUACAAACAUAUAUUUGUCUCUUGAUU-UGCGUAUAGAGUAUGAGAGAAUAAAAGAAAUUUAUGGACAAACUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUG ......(((((.(((((......))))).)))))...-.((((((...(((((........(((....)))..........(((((((((((....)))))))))))))))).)))))). ( -25.90) >DroSec_CAF1 30911 118 + 1 UACUCGUAAGAUACAAACAAAUUGUUGUCUCUCGAUU-UGCGUAUAGAGUAUG-GAGAAUAAAAGAAAUUUAUGGACAAACUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUG ...(((..((((((((.....))).)))))..)))..-.((((((...(((((-.......(((....)))..........(((((((((((....)))))))))))))))).)))))). ( -27.40) >DroSim_CAF1 32213 119 + 1 UACUCGUAAGAUACAAACAAAUUGUUGUCUCUCGAUU-UGCGUAUAGAGUAUGAGAGAAUAAAAGAAAUUUAUGGACAAACUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUG ...(((..((((((((.....))).)))))..)))..-.((((((...(((((........(((....)))..........(((((((((((....)))))))))))))))).)))))). ( -27.20) >DroEre_CAF1 31887 119 + 1 UACUCGUCAGAUAUAAACAAAUU-UUGUCUGUUGAUAGAGAGUAUAGAGUACGAGAGAAUAAAAGAAAUUUAUGGACAAACUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUG ..(((((....((((......((-(((((....)))))))..))))....)))))...............((((.((....(((((((((((....)))))))))))......)).)))) ( -21.80) >DroYak_CAF1 31249 118 + 1 UACUCGCAAGAUACAAACAAAUU-UUGUCUCUCGAUU-UGCGUGUAGAGUACGAUAGAAUAAAAGAAAUUUAUGGACAAACUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUG ..(.(((((((.(((((.....)-)))).)))....(-((((((((..((.(.(((((..........))))).))).......((((((((....)))))))).))))))))))))).) ( -25.00) >consensus UACUCGUAAGAUACAAACAAAUU_UUGUCUCUCGAUU_UGCGUAUAGAGUAUGAGAGAAUAAAAGAAAUUUAUGGACAAACUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUG ...(((..(((((............)))))..)))....((((((...(((((.....(((((.....)))))........(((((((((((....)))))))))))))))).)))))). (-20.00 = -20.20 + 0.20)



| Location | 4,428,613 – 4,428,733 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 99.33 |
| Mean single sequence MFE | -36.44 |
| Consensus MFE | -36.44 |
| Energy contribution | -36.64 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.56 |
| Structure conservation index | 1.00 |
| SVM decision value | 4.21 |
| SVM RNA-class probability | 0.999837 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428613 120 + 22224390 CUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUGGCUCGUUUGAGAUUUGUUUAAUAAUCAAAAUUCAUUAAGCGAGUGCCGAGUGUAUCGGUGAAAUUAAGCACCCAUAGAUA .(((((((((((....))))))))))).(((.(((((...((((((((((((((((.........))))..)).))))))))))(((((.....)))))........))))))))..... ( -37.60) >DroSec_CAF1 30989 120 + 1 CUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUGGCUCGUUUGAGAUUUGUUUAAUAAUCAAAAUUCAUUAAGCGAGUGCCGAGUGUAUCGGUGAAAUUAAGCACCCAUAGAUA .(((((((((((....))))))))))).(((.(((((...((((((((((((((((.........))))..)).))))))))))(((((.....)))))........))))))))..... ( -37.60) >DroSim_CAF1 32292 120 + 1 CUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUGGCUCGUUUGAGAUUUGUUUAAUAAUCAAAAUUCAUUAAGCGAGUGCCGAGUGUAUCGGUGAAAUUAAGCACCCAUAGAUA .(((((((((((....))))))))))).(((.(((((...((((((((((((((((.........))))..)).))))))))))(((((.....)))))........))))))))..... ( -37.60) >DroEre_CAF1 31966 120 + 1 CUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUGGCUCGUUUGAGAUUUGUUUAAUAAUCAAAAUUCAUUAAGCGAGUGCCGAGUGUAUCGGUGAAAUUAAGCACCCAUAGAUA .(((((((((((....))))))))))).(((.(((((...((((((((((((((((.........))))..)).))))))))))(((((.....)))))........))))))))..... ( -37.60) >DroYak_CAF1 31327 120 + 1 CUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUGGCUCGUUUGAGAUUUGUUUAAUAAUCAAAAUUCAUUAAGCGAGUGCCCAGUGAAUCGGUGAAAUUAAGCACCCAUAGAUA .(((((((((((....))))))))))).(((.(((((...((((((((((((((((.........))))..)).))))))))))(((.........)))........))))))))..... ( -31.80) >consensus CUAUGCAUUAUUAAAAAAUGAUGUGUACAUGCGGUGCGUGGCUCGUUUGAGAUUUGUUUAAUAAUCAAAAUUCAUUAAGCGAGUGCCGAGUGUAUCGGUGAAAUUAAGCACCCAUAGAUA .(((((((((((....))))))))))).(((.(((((...((((((((((((((((.........))))..)).))))))))))(((((.....)))))........))))))))..... (-36.44 = -36.64 + 0.20)

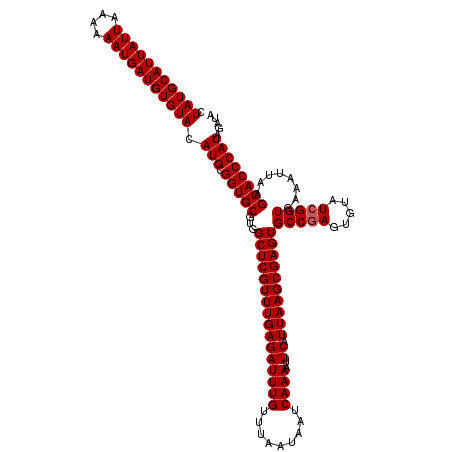

| Location | 4,428,613 – 4,428,733 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 99.33 |
| Mean single sequence MFE | -23.42 |
| Consensus MFE | -21.64 |
| Energy contribution | -21.84 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.924192 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428613 120 - 22224390 UAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGCCACGCACCGCAUGUACACAUCAUUUUUUAAUAAUGCAUAG ...((((((((((.........((((.....)))).....((((..(((..((((.........))))..)))...))))...)))))(.(((....))))..............))))) ( -22.80) >DroSec_CAF1 30989 120 - 1 UAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGCCACGCACCGCAUGUACACAUCAUUUUUUAAUAAUGCAUAG ...((((((((((.........((((.....)))).....((((..(((..((((.........))))..)))...))))...)))))(.(((....))))..............))))) ( -22.80) >DroSim_CAF1 32292 120 - 1 UAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGCCACGCACCGCAUGUACACAUCAUUUUUUAAUAAUGCAUAG ...((((((((((.........((((.....)))).....((((..(((..((((.........))))..)))...))))...)))))(.(((....))))..............))))) ( -22.80) >DroEre_CAF1 31966 120 - 1 UAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGCCACGCACCGCAUGUACACAUCAUUUUUUAAUAAUGCAUAG ...((((((((((.........((((.....)))).....((((..(((..((((.........))))..)))...))))...)))))(.(((....))))..............))))) ( -22.80) >DroYak_CAF1 31327 120 - 1 UAUCUAUGGGUGCUUAAUUUCACCGAUUCACUGGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGCCACGCACCGCAUGUACACAUCAUUUUUUAAUAAUGCAUAG ...(((((((((((((((.(((..((((((.(((((....))))).)))))).)))..)))))......(((....)))....)))))(.(((....))))..............))))) ( -25.90) >consensus UAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGCCACGCACCGCAUGUACACAUCAUUUUUUAAUAAUGCAUAG ...((((((((((.........((((.....)))).....((((..(((..((((.........))))..)))...))))...)))))(.(((....))))..............))))) (-21.64 = -21.84 + 0.20)

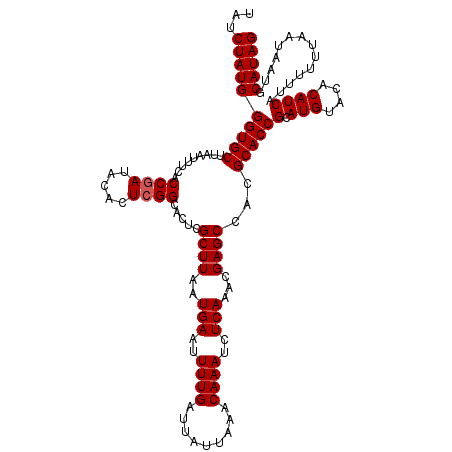
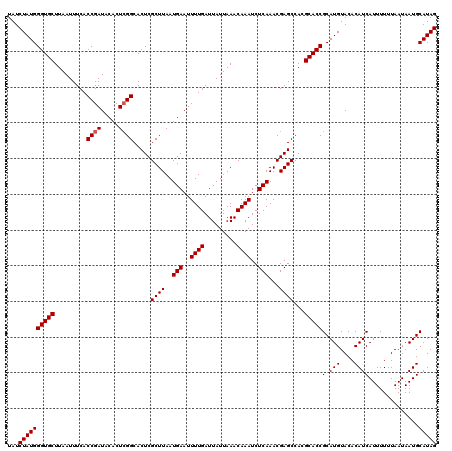
| Location | 4,428,653 – 4,428,773 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 99.00 |
| Mean single sequence MFE | -24.43 |
| Consensus MFE | -23.43 |
| Energy contribution | -23.63 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.27 |
| SVM RNA-class probability | 0.937911 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428653 120 - 22224390 AAUUCAGUUCGUUGACCAUCUCUGCGAUAUAUUUAUAUUGUAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGC ......(((((((..........((((((((.......))))))...(((((((...................)))))))))....(((..((((.........))))..)))))))))) ( -23.21) >DroSec_CAF1 31029 120 - 1 AAUUCAGUUCGUUGACCAUCUCUGCGAUAUAUUUAUAAUGUAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGC ......(((((((..........(((((((((.....)))))))...(((((((...................)))))))))....(((..((((.........))))..)))))))))) ( -23.81) >DroSim_CAF1 32332 120 - 1 AAUUCAGUUCGUUGACCAUCUCUGCGAUAUAUUUAUAAUGUAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGC ......(((((((..........(((((((((.....)))))))...(((((((...................)))))))))....(((..((((.........))))..)))))))))) ( -23.81) >DroEre_CAF1 32006 120 - 1 AAUUCAGUUCGUUGACCAUCUCUGCGAUAUAUUUAUAAUGUAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGC ......(((((((..........(((((((((.....)))))))...(((((((...................)))))))))....(((..((((.........))))..)))))))))) ( -23.81) >DroYak_CAF1 31367 120 - 1 AAUUCAGUUCGUUGACCAUCUCUGCGAUAUAUUUAUAAUGUAUCUAUGGGUGCUUAAUUUCACCGAUUCACUGGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGC ......(((((((((.(((((.((.(((((((.....))))))))).))))).(((((.(((..((((((.(((((....))))).)))))).)))..))))).......)).))))))) ( -27.50) >consensus AAUUCAGUUCGUUGACCAUCUCUGCGAUAUAUUUAUAAUGUAUCUAUGGGUGCUUAAUUUCACCGAUACACUCGGCACUCGCUUAAUGAAUUUUGAUUAUUAAACAAAUCUCAAACGAGC ......(((((((..........(((((((((.....)))))))...(((((((...................)))))))))....(((..((((.........))))..)))))))))) (-23.43 = -23.63 + 0.20)


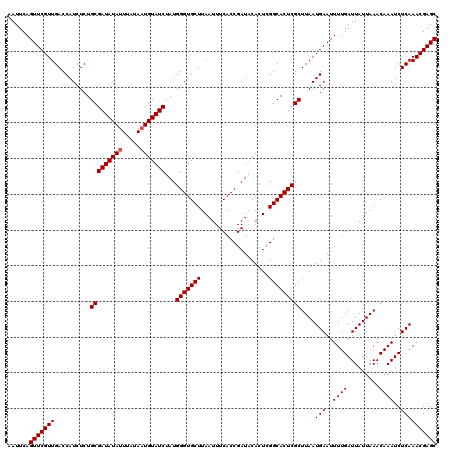
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:30 2006