
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,428,010 – 4,428,203 |
| Length | 193 |
| Max. P | 0.999810 |

| Location | 4,428,010 – 4,428,130 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.83 |
| Mean single sequence MFE | -36.56 |
| Consensus MFE | -35.74 |
| Energy contribution | -35.62 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.44 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.48 |
| SVM RNA-class probability | 0.957769 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428010 120 - 22224390 UUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGUUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCUUCUCCCACUUUCAGACUUCAAAUGCCGCGCCAAAUACGAA ....((.(((.((((((....(((((((((((((.....)))))).)))))))((((((((....))))))))............))))))..))).))..................... ( -37.20) >DroSec_CAF1 30399 120 - 1 UUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCUUCUCCUACUUUCAGACUUCAAAUGCCGCGCCAAAUACGAA ....((.(((.((((((....(((((((((((((.....)))))).)))))))((((((((....))))))))............))))))..))).))..................... ( -35.40) >DroSim_CAF1 31705 120 - 1 UUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCUUCUCCCACUUUCAGACUUCAAAUGCCGCGCCAAAUACGAA ....((.(((.((((((....(((((((((((((.....)))))).)))))))((((((((....))))))))............))))))..))).))..................... ( -37.20) >DroEre_CAF1 31380 120 - 1 UUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGACUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCUUCUCCCACUUUCAGGCUUCAAAUACCGCGCCAAAAACAAA .............(((((..((.(.((((((((((((....)...))))))).((((((((....)))))))).................)))).).))..))))).............. ( -35.80) >DroYak_CAF1 30733 120 - 1 UUCCGAUGUCCAAGGUGUCUGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCUUCUCCCACUUUCAGACUUCAAAUGCCGCGCCAAAAACGAC ....((.(((.((((((....(((((((((((((.....)))))).)))))))((((((((....))))))))............))))))..))).))..................... ( -37.20) >consensus UUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCUUCUCCCACUUUCAGACUUCAAAUGCCGCGCCAAAUACGAA ....((.(((.((((((....(((((((((((((.....)))))).)))))))((((((((....))))))))............))))))..))).))..................... (-35.74 = -35.62 + -0.12)


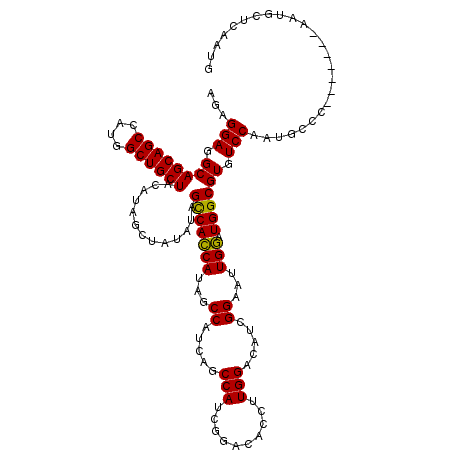
| Location | 4,428,050 – 4,428,163 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.37 |
| Mean single sequence MFE | -36.26 |
| Consensus MFE | -33.76 |
| Energy contribution | -33.28 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.90 |
| SVM RNA-class probability | 0.981769 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428050 113 + 22224390 AGAGGAGGCAGCAGCCAUGGCUGCUACAUAGCUAUAUAGCCAACAUAGCCAUCAGCCAUCGGACACCUUGGACAUCGGAAUUGUAUGGCGUGUCCAAUGCCC-------AAUGCUCAAUG .((((((((((((((....)))))).....(((((.........))))).....))).))((((((((...(((.......)))..)).)))))).......-------....))).... ( -36.80) >DroSec_CAF1 30439 108 + 1 AGAGGAGGCAGCAGCCAUGGCUGCUACAUAGCUAUAUAGCCACCAUAGCCAUCAGCCAUCGGACACCUUGGACAUCGGAAUUGGAUGGCGUGUCCAAUGCCC-------AAUGCU----- ...(((.((((((((....)))))).............(((((((...((.....(((..(....)..))).....))...))).)))))).))).......-------......----- ( -35.40) >DroSim_CAF1 31745 108 + 1 AGAGGAGGCAGCAGCCAUGGCUGCUACAUAGCUAUAUAGCCACCAUAGCCAUCAGCCAUCGGACACCUUGGACAUCGGAAUUGGAUGGCGUGUCCAAUGCCC-------AAUGCU----- ...(((.((((((((....)))))).............(((((((...((.....(((..(....)..))).....))...))).)))))).))).......-------......----- ( -35.40) >DroEre_CAF1 31420 120 + 1 AGAGGAGGCAGCAGCCAUGGCUGCUACAUAGCUAUAUAGUCACCAUAGCCAUCAGCCAUCGGACACCUUGGACAUCGGAAUUGGAUGGCGUGUCCAAUGCCCAAUACCCAAUGCCCAAUG ....(((((((((((....)))))).....(((((.........))))).....))).))((.((..((((..((.((.(((((((.....)))))))..)).))..)))))).)).... ( -36.00) >DroYak_CAF1 30773 113 + 1 AGAGGAGGCAGCAGCCAUGGCUGCUACAUAGCUAUAUAGCCACCAUAGCCAUCAGCCAUCAGACACCUUGGACAUCGGAAUUGGUUGGCGUGUCCAAUGCCC-------AAUGUCCAAUG ....(((((((((((....)))))).....(((((.........))))).....))).)).......((((((((.((.(((((.........)))))..))-------.)))))))).. ( -37.70) >consensus AGAGGAGGCAGCAGCCAUGGCUGCUACAUAGCUAUAUAGCCACCAUAGCCAUCAGCCAUCGGACACCUUGGACAUCGGAAUUGGAUGGCGUGUCCAAUGCCC_______AAUGCUCAAUG ...(((.((((((((....)))))).............(((((((...((.....(((..........))).....))...))).)))))).)))......................... (-33.76 = -33.28 + -0.48)


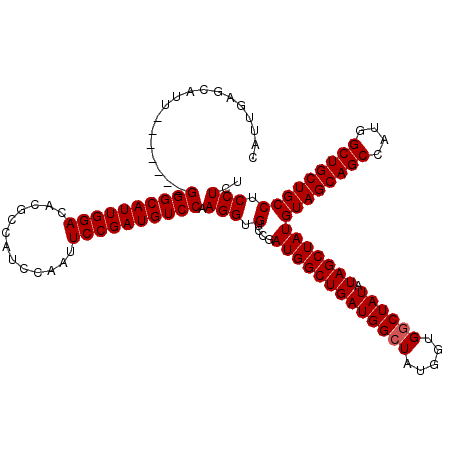
| Location | 4,428,050 – 4,428,163 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.37 |
| Mean single sequence MFE | -49.75 |
| Consensus MFE | -42.72 |
| Energy contribution | -42.92 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -4.34 |
| Structure conservation index | 0.86 |
| SVM decision value | 4.14 |
| SVM RNA-class probability | 0.999810 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428050 113 - 22224390 CAUUGAGCAUU-------GGGCAUUGGACACGCCAUACAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGUUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCU (((((..((((-------((((((((((.............)))))))))))..)))..)))))....((((((((((.....))))))))))((((((((....))))))))....... ( -48.32) >DroSec_CAF1 30439 108 - 1 -----AGCAUU-------GGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCU -----(((((.-------.(.(((((((((((.((((.......)))).)....)))))))))).)..))(((((((((.(((((((....))))))).))))))))))))......... ( -46.80) >DroSim_CAF1 31745 108 - 1 -----AGCAUU-------GGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCU -----(((((.-------.(.(((((((((((.((((.......)))).)....)))))))))).)..))(((((((((.(((((((....))))))).))))))))))))......... ( -46.80) >DroEre_CAF1 31420 120 - 1 CAUUGGGCAUUGGGUAUUGGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGACUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCU (((((((((((((..((((((.((((((.......))))))))))))..)))..))))))))))....(((((((((....)...))))))))((((((((....))))))))....... ( -55.40) >DroYak_CAF1 30773 113 - 1 CAUUGGACAUU-------GGGCAUUGGACACGCCAACCAAUUCCGAUGUCCAAGGUGUCUGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCU .((..((((((-------((((((((((.............)))))))))))..)))))..))(((.(.((((((((((.(((((((....))))))).))))))))))).)))...... ( -51.42) >consensus CAUUGAGCAUU_______GGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUGGCUAUAUAGCUAUGUAGCAGCCAUGGCUGCUGCCUCCUCU ..................((((((((((.............)))))))))).(((.(....(((((((((((((.....)))))).)))))))((((((((....))))))))).))).. (-42.72 = -42.92 + 0.20)


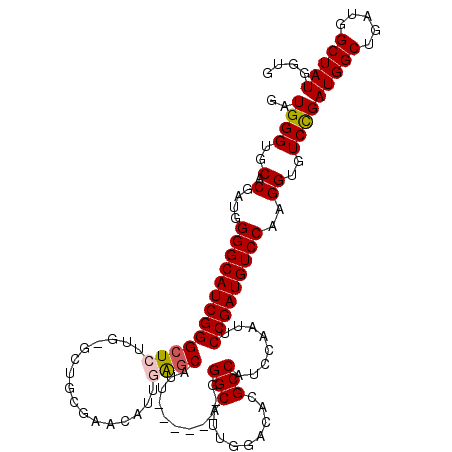
| Location | 4,428,090 – 4,428,203 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.76 |
| Mean single sequence MFE | -43.60 |
| Consensus MFE | -32.89 |
| Energy contribution | -33.09 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.825842 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4428090 113 - 22224390 GAUGGGUGCACGAUGGGGCAUCGGGCUCUUGAGCUGCGAACAUUGAGCAUU-------GGGCAUUGGACACGCCAUACAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGUUG ((((..(((.(((((...)))))((((....)))))))..)))).(((((.-------.(.((((((((((.....(((.......))).....)))))))))).)..)).)))...... ( -40.30) >DroSec_CAF1 30479 99 - 1 GAUGGGUGCACGAUGGGGCAUCGGGCUCUUG--------------AGCAUU-------GGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUG .......((.(.((.((.(((((((((((((--------------..((((-------(((.((((((.......)))))))))))))..))))).)))))))).)).)))))....... ( -42.50) >DroSim_CAF1 31785 99 - 1 GAUGGGUGCACGAUGGGGCAUCGGGCUCUUG--------------AGCAUU-------GGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUG .......((.(.((.((.(((((((((((((--------------..((((-------(((.((((((.......)))))))))))))..))))).)))))))).)).)))))....... ( -42.50) >DroEre_CAF1 31460 120 - 1 GAUGGGUGCACGAUGGGGCAUCGGGCUCUUGGGCUGCGAACAUUGGGCAUUGGGUAUUGGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUG ........(((.(((.(.(((((((((....)))......(((((((((((((..((((((.((((((.......))))))))))))..)))..)))))))))).)))))).)))).))) ( -49.20) >DroYak_CAF1 30813 113 - 1 GAUGGGUGCACGAUGGGGCAUCGGGCUCUUGAGCUGCGAACAUUGGACAUU-------GGGCAUUGGACACGCCAACCAAUUCCGAUGUCCAAGGUGUCUGAUGGCUGAUGGCUAUGGUG ........(((.(((.(.(((((((((....)))......(((..((((((-------((((((((((.............)))))))))))..)))))..))).)))))).)))).))) ( -43.52) >consensus GAUGGGUGCACGAUGGGGCAUCGGGCUCUUG_GCUGCGAACAUUGAGCAUU_______GGGCAUUGGACACGCCAUCCAAUUCCGAUGUCCAAGGUGUCCGAUGGCUGAUGGCUAUGGUG ..((((..(.(....(((((((((((((................))))...........(((.........)))........)))))))))..))..))))(((((.....))))).... (-32.89 = -33.09 + 0.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:26 2006