
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,378,332 – 4,378,439 |
| Length | 107 |
| Max. P | 0.512545 |

| Location | 4,378,332 – 4,378,439 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 77.87 |
| Mean single sequence MFE | -34.08 |
| Consensus MFE | -23.09 |
| Energy contribution | -23.53 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.08 |
| Structure conservation index | 0.68 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.512545 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4378332 107 + 22224390 --ACUACAAUCAUUAUU---AU-CU---UUCAGAUUUAUUUUCGAGGCUCGUGAAUGGCCCAAUGGUCACUAUGUGCACACCCUGUAUGGCAAAAAGGAGGAGCCUCGAGGGUGAG --...............---..-..---.......((((((((((((((((((..(((((....))))).......)))..(((...........)))..))))))))))))))). ( -31.40) >DroPse_CAF1 209892 104 + 1 --AU------UGCUGUU---CG-AUUGACACAGAUUCAUCUUCGAGGCGCGCGAAUGGCCCAAUGGCCACUACGUGCACACCCUGUAUGGCAAGAAGGAGGAGCCGCGAGGGUGAG --..------..(((((---(.-...)).))))..(((((((((.((((((((..(((((....)))))...))))).(..(((...........)))..).))).))))))))). ( -41.00) >DroEre_CAF1 174408 103 + 1 ------CUAUGACUAAU---AU-CU---UCCAGAUUCAUUUUCGAGGCGCGUGAAUGGCCCAAUGGUCACUAUGUGCACACCCUGUAUGGCAAAAAGGAGGAGCCUCGAGGGUGAG ------...........---..-..---.......((((((((((((((((((..(((((....))))).)))))...(..(((...........)))..).))))))))))))). ( -31.70) >DroMoj_CAF1 269023 113 + 1 UCUCUAAACUCUCUAUUGUUCCCUC---UGCAGAUUCAUUUUUGAGGCGCGCGAGUGGCCUAAUGGGCAUUAUGUGCACACGCUCUAUGGCAAGAACGAGGAGCCGCGAGGGUAAG ....................(((((---.((....(((....)))((((((((((((.((....)).)))).)))))...((.(((......))).))....)))))))))).... ( -34.20) >DroAna_CAF1 196291 106 + 1 --AU-AUCCUUUCUAUU---CC-AU---UUCAGAUUCAUUUUCGAGGCUCGUGAAUGGCCGAAUGGACACUACGUGCACACCCUAUACGGCAAGAAGGAGGAGCCGCGAGGGUUUG --..-((((((.(..((---((-.(---(((.....(((((.((.....)).)))))((((.((((................)))).))))..))))..))))..).))))))... ( -25.19) >DroPer_CAF1 232644 104 + 1 --AU------UGCUGUU---CG-AUUGACACAGAUUCAUCUUCGAGGCGCGCGAAUGGCCCAAUGGCCACUACGUGCACACCCUGUAUGGCAAGAAGGAGGAGCCGCGAGGGUGAG --..------..(((((---(.-...)).))))..(((((((((.((((((((..(((((....)))))...))))).(..(((...........)))..).))).))))))))). ( -41.00) >consensus __AU_____UUACUAUU___CC_AU___UACAGAUUCAUUUUCGAGGCGCGCGAAUGGCCCAAUGGCCACUACGUGCACACCCUGUAUGGCAAGAAGGAGGAGCCGCGAGGGUGAG ...................................(((((((((.((((((((..(((((....)))))...))))).(..(((...........)))..).))).))))))))). (-23.09 = -23.53 + 0.45)

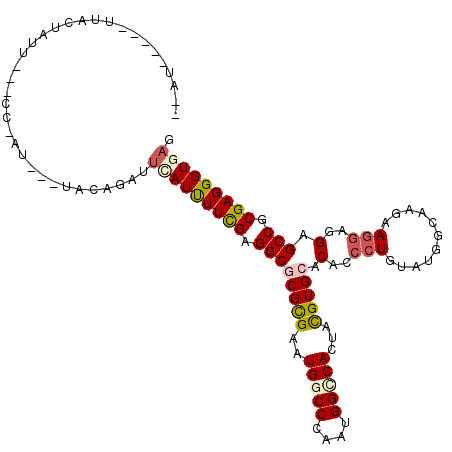
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:09:47 2006