
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,296,101 – 4,296,289 |
| Length | 188 |
| Max. P | 0.999008 |

| Location | 4,296,101 – 4,296,209 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.92 |
| Mean single sequence MFE | -38.32 |
| Consensus MFE | -32.24 |
| Energy contribution | -31.88 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.09 |
| SVM RNA-class probability | 0.913772 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4296101 108 + 22224390 -A-----------UAUAAUUACAGUCGCUGGAUGGUGGCUGGCAAAGCGGAUCCCGAGAUGCCAAAACGCAUGUAUAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAA -.-----------........((((((((....)))))))).....((((((....(.((((......)))).)..))))(((((..(((((((.....)))))))...))))))).... ( -37.70) >DroVir_CAF1 113169 111 + 1 --------UCCU-UUCCGUUGCAGUCGCUGGAUGGUCGCUGGUAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUACAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAA --------....-.((((((((.....(((((((((((((.....)))))........((((......))))......))))))))..((((((.....))))))))))))))....... ( -43.90) >DroGri_CAF1 94393 109 + 1 --------UCUU-U--AUUUGCAGUCGCUGGAUGGUCGCUGGUAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUACAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAA --------....-.--.((((((..(((((((((((((((.....)))))........((((......))))......)))))))).(((((((.....)))))))...))..)))))). ( -39.00) >DroWil_CAF1 100195 117 + 1 AUA--AAUUUCU-CUUAUUUACAGUCGUUGGAUGGUCGCUGGUAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUAUAUUCAUCCAGAUUCACCCACAACGGGUGAGCAAUGGAUGCAAAA ...--.......-......((((..((((((((..(((((.....)))))))))((......)).))))..))))....((((((..(((((((.....)))))))...))))))..... ( -32.30) >DroMoj_CAF1 137710 119 + 1 AUGAAUC-UCCUUUUCUCCAACAGUCGCUGGAUGGUCGCUGGCAAAGCGGAUCCCGAAAUGCCAAAACGCAUGUACAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAA .......-........((((.......(((((((((((((.....)))))........((((......))))......)))))))).(((((((.....)))))))...))))....... ( -35.80) >DroPer_CAF1 100656 117 + 1 GUC--UCUCCCC-CUCUGUUGCAGUCGCUGGAUGGUGGCCGGCAAGGCGGAUCCGGAGAUGCCAAAACGCAUGUAUAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAA ((.--(((((..-.((((((((.((((((....))))))..)))..)))))...))))).)).................((((((..(((((((.....)))))))...))))))..... ( -41.20) >consensus _U______UCCU_UUCAGUUACAGUCGCUGGAUGGUCGCUGGCAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUACAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAA ...........................(((((((((((((.....)))))........((((......))))......))))))))..((((((.....))))))(((.....))).... (-32.24 = -31.88 + -0.36)

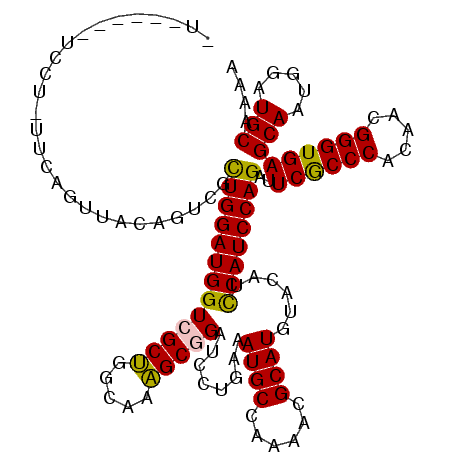

| Location | 4,296,129 – 4,296,249 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.00 |
| Mean single sequence MFE | -30.77 |
| Consensus MFE | -28.39 |
| Energy contribution | -27.87 |
| Covariance contribution | -0.53 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.92 |
| SVM decision value | 3.32 |
| SVM RNA-class probability | 0.999008 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4296129 120 + 22224390 GGCAAAGCGGAUCCCGAGAUGCCAAAACGCAUGUAUAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUU (((((.((((((....(.((((......)))).)..))))(((((..(((((((.....)))))))...))))))).((((.........))))...........))).))......... ( -31.00) >DroVir_CAF1 113200 120 + 1 GGUAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUACAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUU (((...((((((......((((......))))....))))(((((..(((((((.....)))))))...)))))))......((((........))))..........)))......... ( -29.10) >DroPse_CAF1 99344 120 + 1 GGCAAGGCGGAUCCGGAGAUGCCAAAACGCAUGUAUAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCCUUUCACAAAUUAAAAUUGACCAACAAUAUU (((((.((((((....(.((((......)))).)..))))(((((..(((((((.....)))))))...))))))).(((((.......)))))...........))).))......... ( -33.30) >DroGri_CAF1 94422 120 + 1 GGUAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUACAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUU (((...((((((......((((......))))....))))(((((..(((((((.....)))))))...)))))))......((((........))))..........)))......... ( -29.10) >DroWil_CAF1 100232 120 + 1 GGUAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUAUAUUCAUCCAGAUUCACCCACAACGGGUGAGCAAUGGAUGCAAAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUU (((......(((..(((((((...(((((.((.......((((((..(((((((.....)))))))...))))))......))))))))))))))...))).......)))......... ( -28.84) >DroPer_CAF1 100693 120 + 1 GGCAAGGCGGAUCCGGAGAUGCCAAAACGCAUGUAUAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCCUUUCACAAAUUAAAAUUGACCAACAAUAUU (((((.((((((....(.((((......)))).)..))))(((((..(((((((.....)))))))...))))))).(((((.......)))))...........))).))......... ( -33.30) >consensus GGCAAAGCGGAUCCUGAAAUGCCAAAACGCAUGUAUAUCCAUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUU (((((.((((((....(.((((......)))).)..))))(((((..(((((((.....)))))))...)))))))......((((........)))).......))).))......... (-28.39 = -27.87 + -0.53)
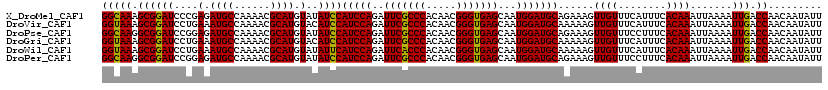
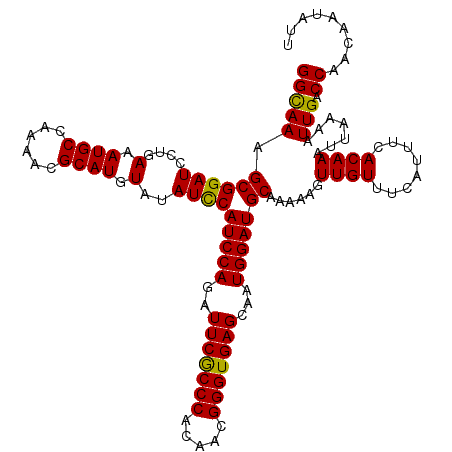
| Location | 4,296,169 – 4,296,289 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.56 |
| Mean single sequence MFE | -25.33 |
| Consensus MFE | -25.80 |
| Energy contribution | -25.30 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.12 |
| Structure conservation index | 1.02 |
| SVM decision value | 1.96 |
| SVM RNA-class probability | 0.983944 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 4296169 120 + 22224390 AUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUUAGUGAUAAACAUGGAUUUGUAAGUACUGUACGUCUUCUUU .((((..(((((((.....)))))))...))))((((((....(((((....((((....(((.((((.....)))).))))))).)))))....))))))................... ( -24.90) >DroPse_CAF1 99384 120 + 1 AUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCCUUUCACAAAUUAAAAUUGACCAACAAUAUUAGUGAUAAACAUGGAUUUGUAAGUACUGUACGUCUUCUUU (((((..(((((((.....)))))))...)))))((((((((.......)))))(((((((...((((.....))))....((.....))...)))))))......)))........... ( -25.00) >DroWil_CAF1 100272 120 + 1 AUCCAGAUUCACCCACAACGGGUGAGCAAUGGAUGCAAAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUUAGUGAUAAACAUGGAUUUGUAAGUACUGUACGUCUUCUUU .((((..(((((((.....)))))))...))))((((((....(((((....((((....(((.((((.....)))).))))))).)))))....))))))................... ( -25.20) >DroMoj_CAF1 137789 120 + 1 AUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAAAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUUAGUGAUAAACAUGGAUUUGUAAGUACUGUACGUCUUCUUU .((((..(((((((.....)))))))...))))((((((....(((((....((((....(((.((((.....)))).))))))).)))))....))))))................... ( -24.80) >DroAna_CAF1 100938 120 + 1 AUCCAGACUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUUAGUGAUAAACAUGGAUUUGUAAGUACUGUACGUCUUCUUU .((((..(((((((.....)))))))...))))((((((....(((((....((((....(((.((((.....)))).))))))).)))))....))))))................... ( -27.10) >DroPer_CAF1 100733 120 + 1 AUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCCUUUCACAAAUUAAAAUUGACCAACAAUAUUAGUGAUAAACAUGGAUUUGUAAGUACUGUACGUCUUCUUU (((((..(((((((.....)))))))...)))))((((((((.......)))))(((((((...((((.....))))....((.....))...)))))))......)))........... ( -25.00) >consensus AUCCAGAUUCGCCCACAACGGGUGAGCAAUGGAUGCAGAAAGUUGUUUCAUUUCACAAAUUAAAAUUGACCAACAAUAUUAGUGAUAAACAUGGAUUUGUAAGUACUGUACGUCUUCUUU .((((..(((((((.....)))))))...))))((((((....(((((....((((....(((.((((.....)))).))))))).)))))....))))))................... (-25.80 = -25.30 + -0.50)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:09:05 2006