
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 4,160,426 – 4,160,537 |
| Length | 111 |
| Max. P | 0.794230 |

| Location | 4,160,426 – 4,160,537 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 83.06 |
| Mean single sequence MFE | -53.73 |
| Consensus MFE | -39.76 |
| Energy contribution | -43.18 |
| Covariance contribution | 3.42 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.794230 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
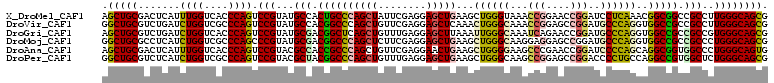
>X_DroMel_CAF1 4160426 111 + 22224390 CGCUGCCCAAGGCGGCCGCCGUUUGAGGAUCCGGUUCCGGUUUACCCAGCUUCAGCUCCUCGAAUAGCUGGGCAGUGGCAUACGGACUGGGUGACCAAAUGAGUCGCAGCU (((((((((.(((....)))(((((((((.(((....)))........((....)))))))))))...)))))))))((((.((...(((....)))..)).)).)).... ( -45.90) >DroVir_CAF1 51808 111 + 1 CGCUGCCCAAGGCGGCGGCCACCUGGGCAUCCGGCUCCGGUUUGCCCAGUUUGAGCUCCUCGAACAGCUGGGCCGUGGCAUACGGACUGGGCGACCAGAUCAGACGCAGCC .(((((((...((.((((((...((((((.(((....)))..))))))(((((((...))))))).....)))))).))....)).((((....)))).......))))). ( -57.60) >DroGri_CAF1 40443 111 + 1 CGCUGCCCACGGCGGCGGCCACCUGGGCAUCCGGUUCUGAUUUGCCCAAUUUAAGCUCCUCAAACAGCUGAGCCGUCGCAUACGGACUGGGUGACCAGAUCAGACGCAGCU .(((((((...(((((((((...((((((..((....))...)))))).....((((........))))).))))))))....)).((((....)))).......))))). ( -48.30) >DroMoj_CAF1 64315 111 + 1 CGCUGCCCAGGGCGGCGGCCACCUGGGCAUCCGGCUCCUCCUUGCCCAGCUUCAGCUCCUCGAAGAGCUGGGCCGUCGCAUACGGGCUGGGCGACCAGAUGAGGCGCAGCC .(((((((...(((((((((..(((((((...((.....)).)))))))...((((((......)))))))))))))))...((..((((....)))).)).)).))))). ( -62.40) >DroAna_CAF1 57718 111 + 1 CACUGCCCAGGGCCACCGCCUGCUGGGGAUCCGGUUCGGGCUUCCCCAGCUUCAGUUCCUCGAACAGCUGGGCGGUGGCGUACGGACUGGGUGACCAAAUGAGUCGCAGCU ..((((.....(((((((((.((((((((((((...))))..))))))))..(((((........)))))))))))))).....((((((....)).....)))))))).. ( -55.70) >DroPer_CAF1 73850 111 + 1 CGCUGCCCAGAGCCACGGCCUGGCAGGGGUCCGGCUCCGGCUUGCCCAGCUUCAGCUCCUCAAACAGCUGGGCCGUAGCGUACGGACUGGGCGACCAGAUGAGACGCAGCC .(((((((...((.((((((..((..(((((((....)))...))))(((....))).........))..)))))).))....)).((((....)))).......))))). ( -52.50) >consensus CGCUGCCCAGGGCGGCGGCCACCUGGGCAUCCGGCUCCGGCUUGCCCAGCUUCAGCUCCUCGAACAGCUGGGCCGUGGCAUACGGACUGGGCGACCAGAUGAGACGCAGCC .(((((((...((.((((((..(((((((.(((....)))..)))))))...(((((........))))))))))).))....)).((((....)))).......))))). (-39.76 = -43.18 + 3.42)



| Location | 4,160,426 – 4,160,537 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 83.06 |
| Mean single sequence MFE | -51.95 |
| Consensus MFE | -41.24 |
| Energy contribution | -41.42 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.611864 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
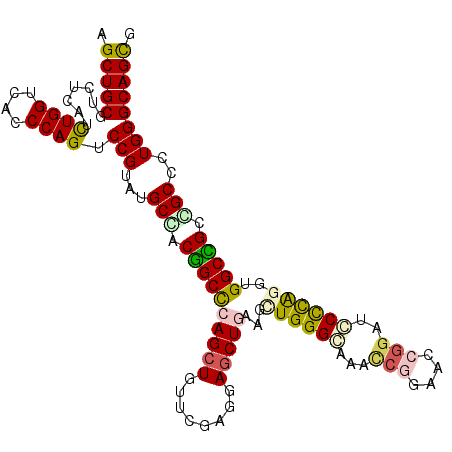
>X_DroMel_CAF1 4160426 111 - 22224390 AGCUGCGACUCAUUUGGUCACCCAGUCCGUAUGCCACUGCCCAGCUAUUCGAGGAGCUGAAGCUGGGUAAACCGGAACCGGAUCCUCAAACGGCGGCCGCCUUGGGCAGCG .(((((((((.....))))..((((..((..((((..(((((((((..(((......))))))))))))..(((....)))..........))))..))..))))))))). ( -46.20) >DroVir_CAF1 51808 111 - 1 GGCUGCGUCUGAUCUGGUCGCCCAGUCCGUAUGCCACGGCCCAGCUGUUCGAGGAGCUCAAACUGGGCAAACCGGAGCCGGAUGCCCAGGUGGCCGCCGCCUUGGGCAGCG .((((((((((.((((((.(((((((((((.....))))...((((.(....).))))...)))))))..))))))..))))).((.((((((...)))))).))))))). ( -52.80) >DroGri_CAF1 40443 111 - 1 AGCUGCGUCUGAUCUGGUCACCCAGUCCGUAUGCGACGGCUCAGCUGUUUGAGGAGCUUAAAUUGGGCAAAUCAGAACCGGAUGCCCAGGUGGCCGCCGCCGUGGGCAGCG .(((..(((((.((((((..((((((.((....))..(((((..(.....)..)))))...))))))...))))))..)))))((((((((((...))))).)))))))). ( -44.90) >DroMoj_CAF1 64315 111 - 1 GGCUGCGCCUCAUCUGGUCGCCCAGCCCGUAUGCGACGGCCCAGCUCUUCGAGGAGCUGAAGCUGGGCAAGGAGGAGCCGGAUGCCCAGGUGGCCGCCGCCCUGGGCAGCG ((((.((((......))).(((((((((((.....))))..(((((((....)))))))..))))))).....).))))...(((((((((((...))).))))))))... ( -59.20) >DroAna_CAF1 57718 111 - 1 AGCUGCGACUCAUUUGGUCACCCAGUCCGUACGCCACCGCCCAGCUGUUCGAGGAACUGAAGCUGGGGAAGCCCGAACCGGAUCCCCAGCAGGCGGUGGCCCUGGGCAGUG .(((((((((.....))))..((((.......(((((((((.....((((...))))....((((((((...(((...))).)))))))).))))))))).))))))))). ( -53.80) >DroPer_CAF1 73850 111 - 1 GGCUGCGUCUCAUCUGGUCGCCCAGUCCGUACGCUACGGCCCAGCUGUUUGAGGAGCUGAAGCUGGGCAAGCCGGAGCCGGACCCCUGCCAGGCCGUGGCUCUGGGCAGCG .(((((((((..((((((.((((((((((((...)))))..(((((.(....).)))))..)))))))..))))))...)))).....((((((....)).))))))))). ( -54.80) >consensus AGCUGCGUCUCAUCUGGUCACCCAGUCCGUAUGCCACGGCCCAGCUGUUCGAGGAGCUGAAGCUGGGCAAACCGGAACCGGAUCCCCAGGUGGCCGCCGCCCUGGGCAGCG .(((((.......((((....)))).(((...(((.((((((((((........)))))...((((((...(((....)))..))))))..))))).)))..)))))))). (-41.24 = -41.42 + 0.17)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:08:02 2006