| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 3,808,609 – 3,808,746 |
| Length | 137 |
| Max. P | 0.966260 |

| Location | 3,808,609 – 3,808,720 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 79.58 |
| Mean single sequence MFE | -41.73 |
| Consensus MFE | -25.75 |
| Energy contribution | -26.98 |
| Covariance contribution | 1.23 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.632863 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 3808609 111 + 22224390 GCAACUUGCUGACGCAACUCCUCAU-UAGUUGCCUGUAGAUCAGCCAGCUGGUUGGUAAGCUCCUGGAGCUGUUUGGCUAGCUCCGCGCUCACCUGUGCAUCGGUAUGCGCU .(((((.((((..((((((......-.))))))(((.....))).)))).)))))..........(((((((......)))))))((((..(((........)))..)))). ( -40.50) >DroGri_CAF1 9670 107 + 1 ----UCACCGGCUGCUGUUCGGCG-CUGCUGUUACGCAGCUCUUGCAGCUGAUUCUCCAGCUCCUGAAGCUGCUUGGCCAGCUCCACAUUGACCUGGGCAUCCGUGGAGGCC ----....(((((((.....((.(-((((......))))).)).)))))))..(((((((((.....))))(..((.((((.((......)).)))).))..)..))))).. ( -39.60) >DroSec_CAF1 7467 111 + 1 GCAACUUGCUGACGCAACUCCUCAU-UAGUUGCCUGUAGAUCAGCUAGCUGGUUGGUGAGCUCCUGGAGCUGUUUGGCGAGCUCCGCGCUCACCUGUGCGUCGGUAGACGCU ((..((.(((((((((.......((-((((((.(((.....))).)))))))).(((((((...(((((((........))))))).)))))))..)))))))))))..)). ( -48.80) >DroSim_CAF1 7746 111 + 1 GCAACUUGCUGACGCAACUCCUCAU-UAGUCGCCUGUAGAUCAGCUAGCUGGUUGGUGAGCUCCUGGAGCUGUUUGGCGAGCUCCGCGCUCACCUGUGCAUCGGUAGACGCU ((...((((....))))........-..((((((((((((((((....))))))(((((((...(((((((........))))))).)))))))..))))..))).))))). ( -41.20) >DroEre_CAF1 7453 111 + 1 GCAACUUGGUGACGCAACUCCUCAU-UAGUUGCUUGUAGCUCCGUUAGCUGGUUGGUGAGUUCCUGGAGCUGUUUGGCUAGCUCCGCGCUCACCUGUGCAUCGGUGGACGCU ((.((..(....)((((((......-.))))))..)).))..((((.((((((.(((((((...((((((((......)))))))).))))))).....)))))).)))).. ( -41.00) >DroYak_CAF1 7532 111 + 1 GCAACUUCCUGACGCAACUCCUCAU-UAGUUGAUUGUAGGUUCGCUAGCUGGUUGGUGAGUUCUUGAAGCUGUUUGGCCAGCUCCGCGCUCACCUGCGCAUCGGUGGACGCU ((......(((...(((((......-.)))))....)))((((((((((((((..(..((((.....))))..)..)))))))..((((......))))...))))))))). ( -39.30) >consensus GCAACUUGCUGACGCAACUCCUCAU_UAGUUGCCUGUAGAUCAGCUAGCUGGUUGGUGAGCUCCUGGAGCUGUUUGGCCAGCUCCGCGCUCACCUGUGCAUCGGUAGACGCU .....(((((((.((((((........))))))..............((.....(((((((...((((((((......)))))))).)))))))...)).)))))))..... (-25.75 = -26.98 + 1.23)


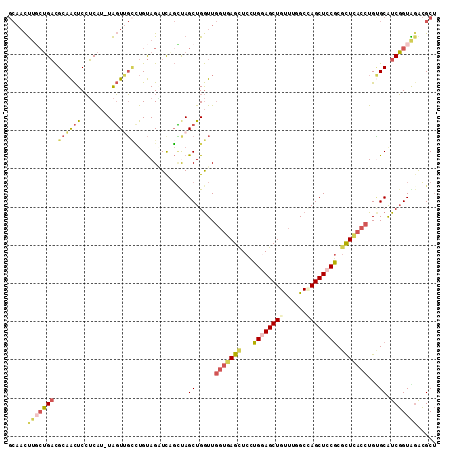
| Location | 3,808,609 – 3,808,720 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 79.58 |
| Mean single sequence MFE | -43.28 |
| Consensus MFE | -29.10 |
| Energy contribution | -30.00 |
| Covariance contribution | 0.90 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.80 |
| Structure conservation index | 0.67 |
| SVM decision value | 1.59 |
| SVM RNA-class probability | 0.966260 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 3808609 111 - 22224390 AGCGCAUACCGAUGCACAGGUGAGCGCGGAGCUAGCCAAACAGCUCCAGGAGCUUACCAACCAGCUGGCUGAUCUACAGGCAACUA-AUGAGGAGUUGCGUCAGCAAGUUGC .(.((((....)))).).(((((((..((((((........))))))....)))))))...(((((.((((((......((((((.-......)))))))))))).))))). ( -44.00) >DroGri_CAF1 9670 107 - 1 GGCCUCCACGGAUGCCCAGGUCAAUGUGGAGCUGGCCAAGCAGCUUCAGGAGCUGGAGAAUCAGCUGCAAGAGCUGCGUAACAGCAG-CGCCGAACAGCAGCCGGUGA---- ((((((((((...((....))...))))))...))))..(((((((..(.(((((......))))).)..)))))))..........-(((((.........))))).---- ( -44.10) >DroSec_CAF1 7467 111 - 1 AGCGUCUACCGACGCACAGGUGAGCGCGGAGCUCGCCAAACAGCUCCAGGAGCUCACCAACCAGCUAGCUGAUCUACAGGCAACUA-AUGAGGAGUUGCGUCAGCAAGUUGC .(((((....)))))...(((((((..((((((........))))))....)))))))...(((((.((((((......((((((.-......)))))))))))).))))). ( -50.20) >DroSim_CAF1 7746 111 - 1 AGCGUCUACCGAUGCACAGGUGAGCGCGGAGCUCGCCAAACAGCUCCAGGAGCUCACCAACCAGCUAGCUGAUCUACAGGCGACUA-AUGAGGAGUUGCGUCAGCAAGUUGC .(((((....)))))...(((((((..((((((........))))))....)))))))...(((((.((((((......((((((.-......)))))))))))).))))). ( -48.10) >DroEre_CAF1 7453 111 - 1 AGCGUCCACCGAUGCACAGGUGAGCGCGGAGCUAGCCAAACAGCUCCAGGAACUCACCAACCAGCUAACGGAGCUACAAGCAACUA-AUGAGGAGUUGCGUCACCAAGUUGC .(((((....)))))...(((((.(((((((((........))))))...(((((..((.((.......)).((.....)).....-.))..)))))))))))))....... ( -38.20) >DroYak_CAF1 7532 111 - 1 AGCGUCCACCGAUGCGCAGGUGAGCGCGGAGCUGGCCAAACAGCUUCAAGAACUCACCAACCAGCUAGCGAACCUACAAUCAACUA-AUGAGGAGUUGCGUCAGGAAGUUGC .(((((....)))))(((((((((..((((((((......)))))))..)..))))))..((.....((((.(((.((........-.)))))..))))....))....))) ( -35.10) >consensus AGCGUCCACCGAUGCACAGGUGAGCGCGGAGCUAGCCAAACAGCUCCAGGAGCUCACCAACCAGCUAGCUGAUCUACAGGCAACUA_AUGAGGAGUUGCGUCAGCAAGUUGC .(((((....)))))...(((((((..((((((........))))))....))))))).....................((((((...(((.(.....).)))...)))))) (-29.10 = -30.00 + 0.90)

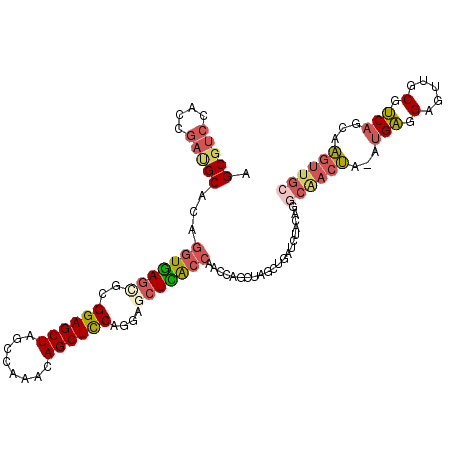
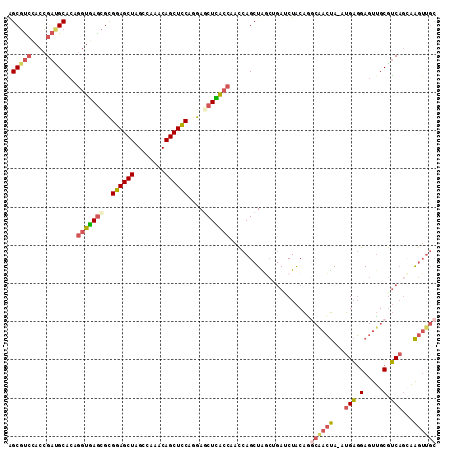
| Location | 3,808,646 – 3,808,746 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 82.47 |
| Mean single sequence MFE | -38.09 |
| Consensus MFE | -23.72 |
| Energy contribution | -24.37 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.558032 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 3808646 100 - 22224390 GCUUCCCGCAGUCCGCAAUCGGAAGCAGCGCAUACCGAUGCACAGGUGAGCGCGGAGCUAGCCAAACAGCUCCAGGAGCUUACCAACCAGCUGGCUGAUC ........(((((.((....((((((.((((.((((........)))).))))((((((........))))))....)))).)).....)).)))))... ( -38.30) >DroSec_CAF1 7504 100 - 1 GCUUCCCGCAGUCCGCAAUCGGAGGCAGCGUCUACCGACGCACAGGUGAGCGCGGAGCUCGCCAAACAGCUCCAGGAGCUCACCAACCAGCUAGCUGAUC ((((((.((.....))....)))))).(((((....)))))...(((((((..((((((........))))))....)))))))...(((....)))... ( -43.30) >DroSim_CAF1 7783 100 - 1 GCUUCCCGCAGUCCCCAAUCGGAGGCAGCGUCUACCGAUGCACAGGUGAGCGCGGAGCUCGCCAAACAGCUCCAGGAGCUCACCAACCAGCUAGCUGAUC ((((((..............)))))).(((((....)))))...(((((((..((((((........))))))....)))))))...(((....)))... ( -39.54) >DroEre_CAF1 7490 100 - 1 UUUUCCAGCAGUCCGCAAUCGGAGGCAGCGUCCACCGAUGCACAGGUGAGCGCGGAGCUAGCCAAACAGCUCCAGGAACUCACCAACCAGCUAACGGAGC ..(((((((.....(((.((((.((......)).)))))))...((((((..(((((((........))))))..)..)))))).....)))...)))). ( -35.80) >DroYak_CAF1 7569 100 - 1 UCUUCCAGCAGUCUGCAAUCGGAGGCAGCGUCCACCGAUGCGCAGGUGAGCGCGGAGCUGGCCAAACAGCUUCAAGAACUCACCAACCAGCUAGCGAACC ..(((((((....(((.(((((.((......)).)))))..)))((((((..((((((((......)))))))..)..)))))).....))).).))).. ( -36.50) >DroAna_CAF1 7869 97 - 1 CUAACCCAGAGUG---AGGCGACGGCGGCCACCACCGACGCCCAGGUUAGCCAGGAGCUGGCCAAGCAGCUGCACGACCUGACCAACCAACUGGCGGAGC ....(((((..((---.((((.(((.((...)).))).))))))((((((...(.(((((......))))).).....))))))......)))).).... ( -35.10) >consensus GCUUCCCGCAGUCCGCAAUCGGAGGCAGCGUCCACCGAUGCACAGGUGAGCGCGGAGCUAGCCAAACAGCUCCAGGAGCUCACCAACCAGCUAGCGGAUC ...........(((......)))(((.(((((....)))))...((((((...((((((........)))))).....)))))).....)))........ (-23.72 = -24.37 + 0.64)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:05:37 2006