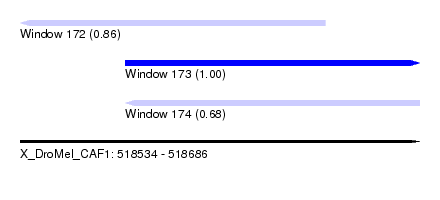
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 518,534 – 518,686 |
| Length | 152 |
| Max. P | 0.997797 |

| Location | 518,534 – 518,650 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.92 |
| Mean single sequence MFE | -29.88 |
| Consensus MFE | -20.72 |
| Energy contribution | -20.96 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.863379 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 518534 116 - 22224390 CAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAU----AUAUCACACCCAAGUAAACAUGCCCAUGGAGAUAGUAUGCAGAUAGAAAUACA ......(((((.((((((...((((((.....)))))).....)))))).....(.(((((((----...((((......))...(((....))))).))))))).).)))))....... ( -27.60) >DroSec_CAF1 4844 110 - 1 CAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAU----AUAUCACACCCAAGCAAACA------UGGAGACAGUAUGCAGAUAGAAGCACA ......(((((.((((((...((((((.....)))))).....))))))......(((((...----)))))........(((.((.------((....)))).))).)))))....... ( -29.90) >DroSim_CAF1 4864 114 - 1 CAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAUAUGUAUAUCACACCCAAGCAAACA------UGGAGACAGUAUGCAGAUAGGAGCACA ......(((((.((((((...((((((.....)))))).....))))))......(((((.......)))))........(((.((.------((....)))).))).)))))....... ( -31.20) >DroEre_CAF1 4723 114 - 1 CAACAGCUGGCAGCUGAACCCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUACGUG----UCACACCCAAGUAGACACACCCAUGGAGA-AGU-UGCAGAUAGAAAUUCA .......(((((((........))))))).(((((((..(((((((........)))))))..(((----((((......)).)))))...........-.))-)))))........... ( -31.20) >DroYak_CAF1 4885 118 - 1 CAACAGCUGUCAGCUGAACUCCGCUGCUAAUUGCAGCACAAUAUGCAGUGAAACGGAUAUUAUAUGUACAUCGCACCCAAGUAAACACGCCCAUGGAGC-AGU-UGCAGGCAGAAAUUCG .....(((..((((((..(((((((((.....)))))......(((.(((..(((.........))).))).)))...................)))))-)))-))..)))......... ( -29.50) >consensus CAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAU_UG_AUAUCACACCCAAGUAAACA__CCCAUGGAGA_AGUAUGCAGAUAGAAAUACA ...((((.....))))..(((((((((.....)))))..(((((((........))))))).................................))))...................... (-20.72 = -20.96 + 0.24)

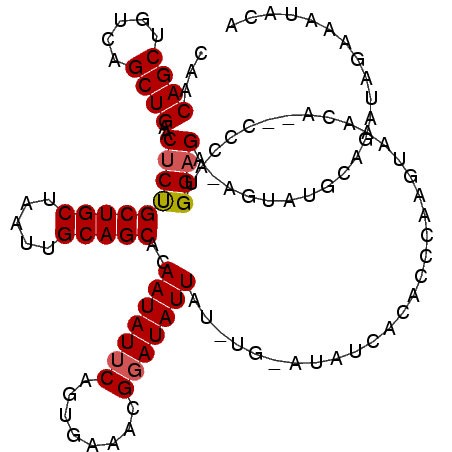
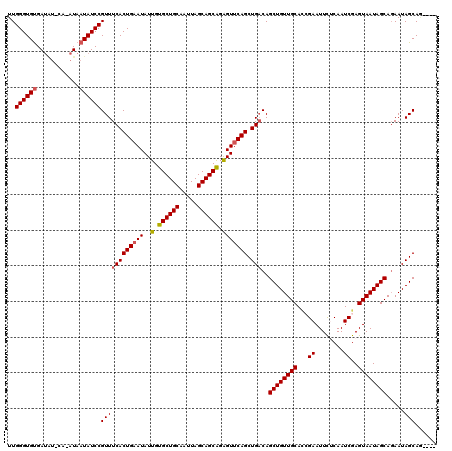
| Location | 518,574 – 518,686 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.17 |
| Mean single sequence MFE | -35.42 |
| Consensus MFE | -29.64 |
| Energy contribution | -29.92 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.66 |
| Structure conservation index | 0.84 |
| SVM decision value | 2.93 |
| SVM RNA-class probability | 0.997797 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 518574 112 + 22224390 UUGGGUGUGAUAU----AUAAUAUCCGUUUCACUGAAUAUUGUGCUGCAAUUAGCAGCAGAGUUCAGCUGACAGCUGUUGCAACGAAUUCUCAAUCGAGUAAUAGCAGAAUAGCAG---- ..((((((.....----...))))))((((((((((((..(.((((((.....)))))).))))))).)))..((((((((..(((........))).)))))))).....)))..---- ( -35.20) >DroSec_CAF1 4878 112 + 1 UUGGGUGUGAUAU----AUAAUAUCCGUUUCACUGAAUAUUGUGCUGCAAUUAGCAGCAGAGUUCAGCUGACAGCUGUUGCACGGAAUUCUCAAUCGAGUAAUAGCAGAAUAGCAG---- ..((((((.....----...))))))((((((((((((..(.((((((.....)))))).))))))).)))..((((((((.(((.........))).)))))))).....)))..---- ( -35.10) >DroSim_CAF1 4898 116 + 1 UUGGGUGUGAUAUACAUAUAAUAUCCGUUUCACUGAAUAUUGUGCUGCAAUUAGCAGCAGAGUUCAGCUGACAGCUGUUGCACGGAAUUCCCAAUCGAGUAAUAGCAGAAUAGCAA---- ..((((((.(((....))).))))))((((((((((((..(.((((((.....)))))).))))))).)))..((((((((.(((.........))).)))))))).....)))..---- ( -34.70) >DroEre_CAF1 4761 116 + 1 UUGGGUGUGA----CACGUAAUAUCCGUUUCACUGAAUAUUGUGCUGCAAUUAGCAGCAGGGUUCAGCUGCCAGCUGUUGCACCGAAUUUUCAAUCGAGUAAUAGCACAAUAGCAGAAGG ..((((((..----......))))))......(((..((((((((((......((((((((((......)))..)))))))(((((........))).))..)))))))))).))).... ( -36.10) >DroYak_CAF1 4923 116 + 1 UUGGGUGCGAUGUACAUAUAAUAUCCGUUUCACUGCAUAUUGUGCUGCAAUUAGCAGCGGAGUUCAGCUGACAGCUGUUGCAUCGAAUUCCCAAUCGAGUAAUAGCAGAAUAGCAC---- ....(((((((((.......))))).(((...(((...(((.((((((.....)))))).))).)))..))).((((((((.((((........))))))))))))......))))---- ( -36.00) >consensus UUGGGUGUGAUAU_CA_AUAAUAUCCGUUUCACUGAAUAUUGUGCUGCAAUUAGCAGCAGAGUUCAGCUGACAGCUGUUGCACCGAAUUCUCAAUCGAGUAAUAGCAGAAUAGCAG____ ..((((((............))))))((((((((((((..(.((((((.....)))))).))))))).)))..((((((((...((........))..)))))))).....)))...... (-29.64 = -29.92 + 0.28)

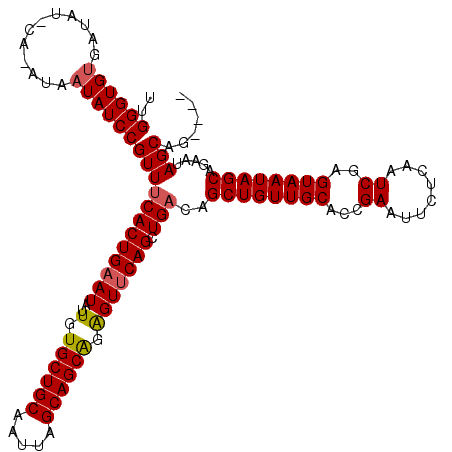
| Location | 518,574 – 518,686 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.17 |
| Mean single sequence MFE | -29.36 |
| Consensus MFE | -21.04 |
| Energy contribution | -21.24 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.680459 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
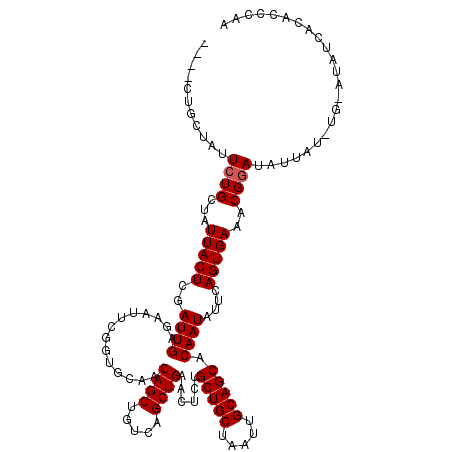
>X_DroMel_CAF1 518574 112 - 22224390 ----CUGCUAUUCUGCUAUUACUCGAUUGAGAAUUCGUUGCAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAU----AUAUCACACCCAA ----..((......)).....(((....))).(((((((((....))..(((.(((((...((((((.....)))))).....))))))))))))))).....----............. ( -24.80) >DroSec_CAF1 4878 112 - 1 ----CUGCUAUUCUGCUAUUACUCGAUUGAGAAUUCCGUGCAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAU----AUAUCACACCCAA ----..((......))........(((....((((((((((....))..(((.(((((...((((((.....)))))).....)))))))).))))).)))..----..)))........ ( -26.30) >DroSim_CAF1 4898 116 - 1 ----UUGCUAUUCUGCUAUUACUCGAUUGGGAAUUCCGUGCAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAUAUGUAUAUCACACCCAA ----..((......))..........(((((...(((((((....))..(((.(((((...((((((.....)))))).....)))))))).))))).......(((.....)))))))) ( -29.60) >DroEre_CAF1 4761 116 - 1 CCUUCUGCUAUUGUGCUAUUACUCGAUUGAAAAUUCGGUGCAACAGCUGGCAGCUGAACCCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUACGUG----UCACACCCAA ........(((((((((...((.(((.(....).)))))((((....(((((((........))))))).)))))))))))))....((((.(((.......))).----))))...... ( -36.20) >DroYak_CAF1 4923 116 - 1 ----GUGCUAUUCUGCUAUUACUCGAUUGGGAAUUCGAUGCAACAGCUGUCAGCUGAACUCCGCUGCUAAUUGCAGCACAAUAUGCAGUGAAACGGAUAUUAUAUGUACAUCGCACCCAA ----((((...((((...((((((((........))))((((.((((.....))))......(((((.....)))))......))))))))..))))(((.....)))....)))).... ( -29.90) >consensus ____CUGCUAUUCUGCUAUUACUCGAUUGAGAAUUCGGUGCAACAGCUGUCAGCUGAACUCUGCUGCUAAUUGCAGCACAAUAUUCAGUGAAACGGAUAUUAU_UG_AUAUCACACCCAA ...........((((...(((((..((((..............((((.....))))......(((((.....))))).))))....)))))..))))....................... (-21.04 = -21.24 + 0.20)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:35:01 2006