
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 3,628,390 – 3,628,487 |
| Length | 97 |
| Max. P | 0.885156 |

| Location | 3,628,390 – 3,628,487 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | forward |
| Mean pairwise identity | 68.25 |
| Mean single sequence MFE | -44.08 |
| Consensus MFE | -16.50 |
| Energy contribution | -19.17 |
| Covariance contribution | 2.67 |
| Combinations/Pair | 1.52 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.37 |
| SVM decision value | 0.94 |
| SVM RNA-class probability | 0.885156 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
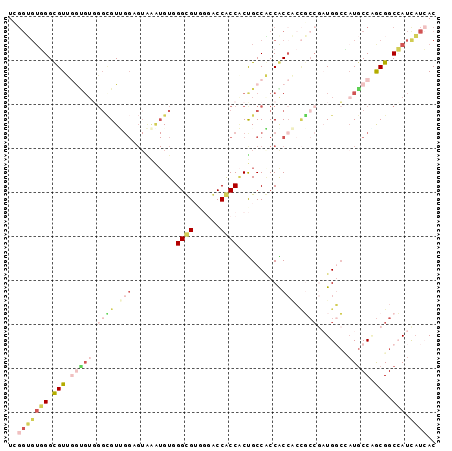
>X_DroMel_CAF1 3628390 97 + 22224390 UCGGUGUGGGUGUUGGCGUGGGCGUUGGAGUAAAUGUGGGUGUGGGACCACCACUGCCACCACCACCGCCGAUGGCCAUGCCAGCGGCCAUUAUCAC ..(((((((.(((((((((((.((((((.......((((..((((.....))))..))))........)))))).))))))))))).))).)))).. ( -50.76) >DroVir_CAF1 47950 91 + 1 UCGGUGUUGGCGUCAAUGUGGGCGUCGGCGUGGGUGUUGGCGGUUUGCCGCCACCGCCGCCGC------CACUGCCAAUGCCGACGGCAAUCAUGAC ((((((((((((((......)))...((((..((((.((((((....)))))).))))..)))------)...)))))))))))............. ( -48.30) >DroSec_CAF1 7739 91 + 1 UCGGUGUGGGUGUUGG------CGUUGGAGUAAAUGUGGGUGUGGGACCACCACUGCCACCACCACCGCCGAUGGCCAUGCCGGCGGCCAUUAUCAC (((((((((.((((((------((.(((.(.....((((..((((.....))))..))))).))).)))))))).)))))))))............. ( -40.80) >DroEre_CAF1 7861 97 + 1 UCGGUGUGGGCGUUGGCGUGGGCGUUGGAGUAAAUGUCGGUGUGGGACCACCACUGCCACCGCCACCGCAGAUGGCUAUGCCCGCGGCCAUUAUCAC ..((((((((((((((((.(((((((......))))))(((((((.....)))).))).))))))..((.....)).)))))))).)))........ ( -41.30) >DroMoj_CAF1 69652 82 + 1 U------GGGCGUCAAUGUGGGUGUG---------GUCGGCGGACUACCACCGCCGCCGCCGCCGAUGCCGCUGUCAAUGCCAGCGGCCAUCAUGAC .------....((((.....((((((---------(.((((((.......))))))))).))))((((((((((.......)))))).)))).)))) ( -40.50) >DroAna_CAF1 13076 97 + 1 UAGGGGUGGGCGUUGGUGUUGGAGUGGGCGUGAAUGUGGGCGUAGGGCCGCCCCUGCCCCCACCUCCUCCAAUGGGCUUACCUGCGGCAAUCCUCAC ..((((..(.(((.((((((((((.(((.(((.....(((.((((((....))))))))))))))))))))))......))).))).)...)))).. ( -42.80) >consensus UCGGUGUGGGCGUUGGUGUGGGCGUUGGAGUAAAUGUGGGCGUGGGACCACCACUGCCACCACCACCGCCGAUGGCCAUGCCAGCGGCCAUCAUCAC ..(((((((.(((.(((((.((((.(((.........(((((......))))).........))).)))).......))))).))).))).)))).. (-16.50 = -19.17 + 2.67)


| Location | 3,628,390 – 3,628,487 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | reverse |
| Mean pairwise identity | 68.25 |
| Mean single sequence MFE | -37.45 |
| Consensus MFE | -18.82 |
| Energy contribution | -18.93 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.68 |
| Mean z-score | -1.43 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.565504 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 3628390 97 - 22224390 GUGAUAAUGGCCGCUGGCAUGGCCAUCGGCGGUGGUGGUGGCAGUGGUGGUCCCACACCCACAUUUACUCCAACGCCCACGCCAACACCCACACCGA (((...(((((((......))))))).((((..((((.(((.((((((((........))))...))))))).))))..))))......)))..... ( -37.40) >DroVir_CAF1 47950 91 - 1 GUCAUGAUUGCCGUCGGCAUUGGCAGUG------GCGGCGGCGGUGGCGGCAAACCGCCAACACCCACGCCGACGCCCACAUUGACGCCAACACCGA (((((((((((((.......)))))))(------(((.(((((.((((((....)))))).......))))).))))..)).))))........... ( -40.00) >DroSec_CAF1 7739 91 - 1 GUGAUAAUGGCCGCCGGCAUGGCCAUCGGCGGUGGUGGUGGCAGUGGUGGUCCCACACCCACAUUUACUCCAACG------CCAACACCCACACCGA (((...(((((((......))))))).((((.(((.(((((..((((((......)).))))..)))))))).))------))......)))..... ( -33.70) >DroEre_CAF1 7861 97 - 1 GUGAUAAUGGCCGCGGGCAUAGCCAUCUGCGGUGGCGGUGGCAGUGGUGGUCCCACACCGACAUUUACUCCAACGCCCACGCCAACGCCCACACCGA (((....((((.(.((((...((((.((((....)))))))).((((((......)))).))............)))).))))).....)))..... ( -36.60) >DroMoj_CAF1 69652 82 - 1 GUCAUGAUGGCCGCUGGCAUUGACAGCGGCAUCGGCGGCGGCGGCGGUGGUAGUCCGCCGAC---------CACACCCACAUUGACGCCC------A ((((.((((.((((((.......))))))))))((..(.((((((((.......)))))).)---------).)..))....))))....------. ( -40.40) >DroAna_CAF1 13076 97 - 1 GUGAGGAUUGCCGCAGGUAAGCCCAUUGGAGGAGGUGGGGGCAGGGGCGGCCCUACGCCCACAUUCACGCCCACUCCAACACCAACGCCCACCCCUA (((.((.(((((...)))))..)).((((((..(((((((....(((((......)))))...))).))))..)))))))))............... ( -36.60) >consensus GUGAUAAUGGCCGCCGGCAUGGCCAUCGGCGGUGGCGGUGGCAGUGGUGGUCCCACACCCACAUUUACUCCAACGCCCACACCAACGCCCACACCGA (((......(((((((.(...(((......)))).)))))))..(((((......))))).........................)))......... (-18.82 = -18.93 + 0.12)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:04:13 2006