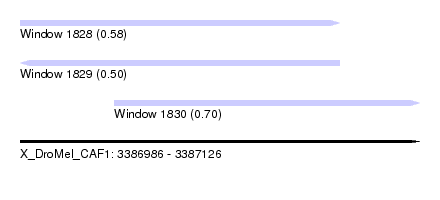
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 3,386,986 – 3,387,126 |
| Length | 140 |
| Max. P | 0.696320 |

| Location | 3,386,986 – 3,387,098 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 80.36 |
| Mean single sequence MFE | -41.28 |
| Consensus MFE | -22.89 |
| Energy contribution | -23.42 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.580373 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 3386986 112 + 22224390 UGACGAACCCUCGUUGCCAAUUGUCGAAGUGGAUGUUCGAAUCGAACGCCGUCCACCAUGUGCCGAAUGCGAUCCACAUGGCGGGCACGUGGUGGAACGCAUAGCUCUUAGA .(((((....)))))((....((.((....((.((((((...)))))))).(((((((((((((...(((.((....)).)))))))))))))))).))))..))....... ( -41.00) >DroVir_CAF1 51358 112 + 1 CGCCGAAUUGUCGUUGCCAAUUGUCGAAGUGGAUUUUCGAAACGAAUGGAAUCCACCAUGUGCCGCAUGGCAUCCACAUGGCUGGCACGUGGUGGAACGCAUUGCUCGUGGA (((.((..((.(((..(((.(((((((((.....))))))..))).)))..((((((((((((((((((.......))).)).))))))))))))))))))....))))).. ( -42.50) >DroPse_CAF1 62722 112 + 1 CGGCGAACGCUCGUUGCCAAUUGUCGAAGUGGAUUUUCGAAACGAAUGAGGUCCACCAUGUGCCGCAUGGAUUCCACACUCCGGCCACGUGGUGGAUCGCAUCGCUUGAGGG ((((((....))))))((.....((((((.....))))))..(((.((.((((((((((((((((..((.......))...))).))))))))))))).))))).....)). ( -42.80) >DroGri_CAF1 49265 112 + 1 CUGCCAAACGUCGUUGCCAAUUGUCGAAGUGGAUUUUCGAAAUGAACAGAAUCCACCAUGUGCCGCAUGCCAUCCACAUGCCUGACACGUGGUGGAACGCAACACUUCUAGA ............(((((......((((((.....))))))...........((((((((((((.(((((.......)))))..).)))))))))))..)))))......... ( -36.20) >DroMoj_CAF1 61275 112 + 1 CUGCGAAACGUCGUUGCCAAUUGUCGAAGUGGAUUUUCGAAACGAAUGGCAUCCACCAUGUGCCACUCGCCGUCCACACGGCCAGCACGUGGUGGACUCCAUUGCUUGGUGA ..((((....))))(((((.(((((((((.....))))))..))).)))))((((((((((((.....(((((....)))))..))))))))))))(.(((.....))).). ( -46.00) >DroAna_CAF1 65704 112 + 1 UAACGAAUGCUCGUUGCCAAUUGUCGAAGUGGAUGUUCGAAUCGAACGGCAUCCACCAUGUGCCGAAUGCCGUCCACACGCUGGCCACGUGGUGGAACGCAUUGUUCGGUGG ((((((....))))))((.....(((((.......)))))(((((((.((.((((((((((((((...((.((....))))))).)))))))))))..))...))))))))) ( -39.20) >consensus CGGCGAAACCUCGUUGCCAAUUGUCGAAGUGGAUUUUCGAAACGAACGGCAUCCACCAUGUGCCGCAUGCCAUCCACACGCCGGGCACGUGGUGGAACGCAUUGCUCGUAGA ((((((....)))))).......((((((.....))))))...........(((((((((((.......................)))))))))))................ (-22.89 = -23.42 + 0.53)

| Location | 3,386,986 – 3,387,098 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 80.36 |
| Mean single sequence MFE | -38.77 |
| Consensus MFE | -23.83 |
| Energy contribution | -23.92 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.61 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 3386986 112 - 22224390 UCUAAGAGCUAUGCGUUCCACCACGUGCCCGCCAUGUGGAUCGCAUUCGGCACAUGGUGGACGGCGUUCGAUUCGAACAUCCACUUCGACAAUUGGCAACGAGGGUUCGUCA .....(((((..((..(((((((.(((((....(((((...)))))..))))).)))))))..))(((((...)))))..............(((....))).))))).... ( -37.90) >DroVir_CAF1 51358 112 - 1 UCCACGAGCAAUGCGUUCCACCACGUGCCAGCCAUGUGGAUGCCAUGCGGCACAUGGUGGAUUCCAUUCGUUUCGAAAAUCCACUUCGACAAUUGGCAACGACAAUUCGGCG ....(((((((((...(((((((.(((((.((...((((...))))))))))).)))))))...)))).).))))..........((((...(((....)))....)))).. ( -35.20) >DroPse_CAF1 62722 112 - 1 CCCUCAAGCGAUGCGAUCCACCACGUGGCCGGAGUGUGGAAUCCAUGCGGCACAUGGUGGACCUCAUUCGUUUCGAAAAUCCACUUCGACAAUUGGCAACGAGCGUUCGCCG ......(((((((.(.(((((((.(((.((...((((((...))))))))))).))))))).).)).)))))(((((.......))))).....((((((....))).))). ( -36.80) >DroGri_CAF1 49265 112 - 1 UCUAGAAGUGUUGCGUUCCACCACGUGUCAGGCAUGUGGAUGGCAUGCGGCACAUGGUGGAUUCUGUUCAUUUCGAAAAUCCACUUCGACAAUUGGCAACGACGUUUGGCAG .(((((.(((((((((.((((((((((.....))))))).))).)))))))))..((((((((...(((.....))))))))))).......(((....)))..)))))... ( -40.80) >DroMoj_CAF1 61275 112 - 1 UCACCAAGCAAUGGAGUCCACCACGUGCUGGCCGUGUGGACGGCGAGUGGCACAUGGUGGAUGCCAUUCGUUUCGAAAAUCCACUUCGACAAUUGGCAACGACGUUUCGCAG .......(((((((.((((((((.(((((((((((....)))))...)))))).)))))))).)))))(((((((((.......))))).....(....)))))....)).. ( -42.60) >DroAna_CAF1 65704 112 - 1 CCACCGAACAAUGCGUUCCACCACGUGGCCAGCGUGUGGACGGCAUUCGGCACAUGGUGGAUGCCGUUCGAUUCGAACAUCCACUUCGACAAUUGGCAACGAGCAUUCGUUA ......((((((((((((...(((((.....)))))(((((((((((((.(....).)))))))))))))....))))..............(((....)))))))).))). ( -39.30) >consensus UCAACAAGCAAUGCGUUCCACCACGUGCCCGCCAUGUGGAUGGCAUGCGGCACAUGGUGGAUGCCAUUCGUUUCGAAAAUCCACUUCGACAAUUGGCAACGACGAUUCGCCG .........((((...(((((((.(((((....((((.....))))..))))).)))))))...))))....(((((.......)))))...(((....))).......... (-23.83 = -23.92 + 0.08)

| Location | 3,387,019 – 3,387,126 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 71.24 |
| Mean single sequence MFE | -49.73 |
| Consensus MFE | -24.52 |
| Energy contribution | -24.75 |
| Covariance contribution | 0.23 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.49 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.696320 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 3387019 107 + 22224390 UGUUCGAAUCGAACGCCGUCCACCAUGUGCCGAAUGCGAUCCACAUGGCGGGCACGUGGUGGAACGCAUAGCUCUUAGAUGCCGGCGGGGGGAUGUCCUG------------GCGGCGC .(((((...)))))((..(((((((((((((...(((.((....)).))))))))))))))))..)).............((((.(((((.....)))))------------.)))).. ( -50.10) >DroVir_CAF1 51391 119 + 1 UUUUCGAAACGAAUGGAAUCCACCAUGUGCCGCAUGGCAUCCACAUGGCUGGCACGUGGUGGAACGCAUUGCUCGUGGAUGGCAGCGGUGCGACGGCGGGUCGCUGCGGCCCGUGGCAC ..((((...)))).....((((((((((((((((((.......))).)).))))))))))))).(((((((((.((.....))))))))))).(.((((((((...)))))))).)... ( -58.00) >DroPse_CAF1 62755 119 + 1 UUUUCGAAACGAAUGAGGUCCACCAUGUGCCGCAUGGAUUCCACACUCCGGCCACGUGGUGGAUCGCAUCGCUUGAGGGCGCCAGCGGCGGGGCGUCGUGUGGCCACCAAAUGUGGCAC ..(((((..(((.((.((((((((((((((((..((.......))...))).))))))))))))).))))).)))))((((((........)))))).....(((((.....))))).. ( -52.20) >DroGri_CAF1 49298 119 + 1 UUUUCGAAAUGAACAGAAUCCACCAUGUGCCGCAUGCCAUCCACAUGCCUGACACGUGGUGGAACGCAACACUUCUAGAUGGCAGCGGCGAGGCGGCACGUGGACACAGUUCUCGGCAC ...((((...((((.(....).(((((((((((.((((((((((.((.....)).)))(((........))).....)))))))........))))))))))).....))))))))... ( -41.60) >DroAna_CAF1 65737 107 + 1 UGUUCGAAUCGAACGGCAUCCACCAUGUGCCGAAUGCCGUCCACACGCUGGCCACGUGGUGGAACGCAUUGUUCGGUGGUCCUGGCGGGGCGCUCUCCGU------------CCGGCGC .......(((((((.((.((((((((((((((...((.((....))))))).)))))))))))..))...))))))).(.((.(((((((....))))))------------).))).. ( -44.30) >DroPer_CAF1 56736 119 + 1 UUUUCGAAACGAAUGAGGUCCACCAUGUGCCGCAUGGAUUCCACACUCCGGCCACGUGGUGGAUCGCAUCGCUUGAGGGCGCCAGCGGCGGGGCGUCGUGUGGCCACCAAAUGUGGCAC ..(((((..(((.((.((((((((((((((((..((.......))...))).))))))))))))).))))).)))))((((((........)))))).....(((((.....))))).. ( -52.20) >consensus UUUUCGAAACGAACGGAAUCCACCAUGUGCCGCAUGCCAUCCACACGCCGGCCACGUGGUGGAACGCAUCGCUCGUGGAUGCCAGCGGCGGGGCGUCGUGUGGCCAC_____GCGGCAC ..((((...)))).....((((((((((((((..((.......))...))).)))))))))))..((..((((.(......).))))((((....))))................)).. (-24.52 = -24.75 + 0.23)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:02:16 2006