
| Sequence ID | X_DroMel_CAF1 |
|---|---|
| Location | 2,986,384 – 2,986,480 |
| Length | 96 |
| Max. P | 0.922006 |

| Location | 2,986,384 – 2,986,480 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | forward |
| Mean pairwise identity | 80.10 |
| Mean single sequence MFE | -31.22 |
| Consensus MFE | -18.11 |
| Energy contribution | -17.62 |
| Covariance contribution | -0.49 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.779713 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2986384 96 + 22224390 CAGGCCGACGCCAAUCCGCAUGAGCUCAGCUAUCGUUGGUUCAUCAACGAUGAGCUAAUGACCGGAGACUUUACCACUAAGAUGGUAAGUGCAUC---A- ..(((....))).....((((((((((.....(((((((((((((...)))))))))))))...))).)))(((((......))))).))))...---.- ( -31.80) >DroVir_CAF1 527772 92 + 1 CAGGCGGACGCCAAUCCGCCGGAGGUCAGCUAUCGUUGGUUCAUCAAUGAUGAGCUAAUCACCGGUGAUUUUACCACCAAAAUGGUAGGUCU-------- ..(((....)))....((((((.(((.((((((((((((....)))))))).)))).))).)))))).....((((((.....))).)))..-------- ( -36.00) >DroEre_CAF1 395078 100 + 1 CAGGCCGAUGCCAAUCCGCACGAGCUCAGCUACCGUUGGUUCAUCAACGAUGAGCUCAUGACCGGCGACUUUACCACCAAGAUGGUAAGCCCACCCGCAC ..(((....))).....((..(((((((.....((((((....)))))).)))))))......((.(..(((((((......)))))))..).)).)).. ( -31.70) >DroYak_CAF1 375613 97 + 1 CAGGCGGACGCCAAUCCGCAUGAGCUCAGCUAUCGCUGGUUCAUCAACGACGAGCUCAUGACCGGCGACUUUACCACCAAAAUGGUAAGCACACC---AA ..((.(..((((......((((((((((((....))).(((.......))))))))))))...))))..(((((((......)))))))..).))---.. ( -32.20) >DroMoj_CAF1 642263 92 + 1 CAGGCGGAUGCGAAUCCGCCGGAGGUCAGCUAUCGUUGGUUCAUCAAUGAUGAGCUCGUGCCUGGGGACUUUACAACGAAAAUGGUAAGUGG-------- ..(((((((....)))))))........(((((((((((((((((...)))))))..((.(....).))....))))))...))))......-------- ( -31.30) >DroAna_CAF1 563703 99 + 1 CAGGCGGAUGCCAAUCCUCACGAGCUCAGCUAUCGAUGGUUUAUCAACGAUGAACUCAUGACGGGGGAUUUUACUACAAAAAUGGUGAGUUUUUUUUCA- ..(((....)))(((((((.(((((...))).((.((((((((((...))))))).))))))))))))))((((((......))))))...........- ( -24.30) >consensus CAGGCGGACGCCAAUCCGCACGAGCUCAGCUAUCGUUGGUUCAUCAACGAUGAGCUCAUGACCGGCGACUUUACCACCAAAAUGGUAAGUCCA_C___A_ ..(((....)))...(((........((((....))))(((((((...))))))).......)))....(((((((......)))))))........... (-18.11 = -17.62 + -0.49)

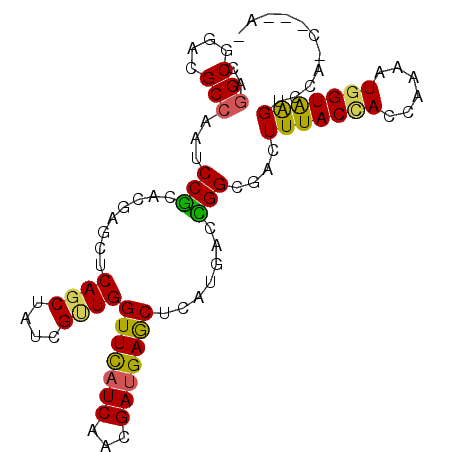
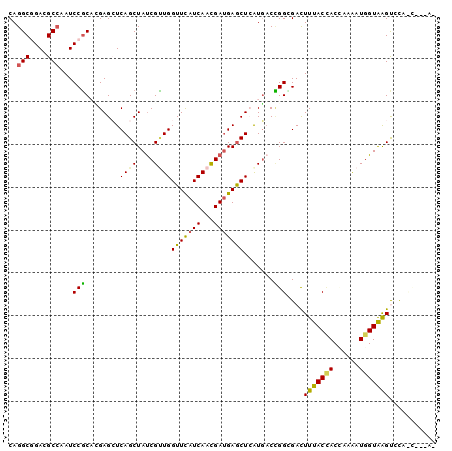
| Location | 2,986,384 – 2,986,480 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 80.10 |
| Mean single sequence MFE | -34.02 |
| Consensus MFE | -21.66 |
| Energy contribution | -24.25 |
| Covariance contribution | 2.59 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.43 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.922006 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>X_DroMel_CAF1 2986384 96 - 22224390 -U---GAUGCACUUACCAUCUUAGUGGUAAAGUCUCCGGUCAUUAGCUCAUCGUUGAUGAACCAACGAUAGCUGAGCUCAUGCGGAUUGGCGUCGGCCUG -.---(((((..(((((((....)))))))......(((((.((((((.(((((((......)))))))))))))((....)).))))))))))...... ( -34.90) >DroVir_CAF1 527772 92 - 1 --------AGACCUACCAUUUUGGUGGUAAAAUCACCGGUGAUUAGCUCAUCAUUGAUGAACCAACGAUAGCUGACCUCCGGCGGAUUGGCGUCCGCCUG --------..(((.(((.....)))))).........((.(.((((((.(((.(((......))).))))))))).).))(((((((....))))))).. ( -31.70) >DroEre_CAF1 395078 100 - 1 GUGCGGGUGGGCUUACCAUCUUGGUGGUAAAGUCGCCGGUCAUGAGCUCAUCGUUGAUGAACCAACGGUAGCUGAGCUCGUGCGGAUUGGCAUCGGCCUG ...(((((.((((((((((....))))).)))))((((((((((((((((((((((......))))))....)))))))))...)))))))....))))) ( -45.40) >DroYak_CAF1 375613 97 - 1 UU---GGUGUGCUUACCAUUUUGGUGGUAAAGUCGCCGGUCAUGAGCUCGUCGUUGAUGAACCAGCGAUAGCUGAGCUCAUGCGGAUUGGCGUCCGCCUG ..---((((..((((((((....)))))))....((((((((((((((((((((((......)))))))....))))))))...))))))))..)))).. ( -43.50) >DroMoj_CAF1 642263 92 - 1 --------CCACUUACCAUUUUCGUUGUAAAGUCCCCAGGCACGAGCUCAUCAUUGAUGAACCAACGAUAGCUGACCUCCGGCGGAUUCGCAUCCGCCUG --------.............((((((....(((....)))......(((((...)))))..))))))............(((((((....))))))).. ( -22.50) >DroAna_CAF1 563703 99 - 1 -UGAAAAAAAACUCACCAUUUUUGUAGUAAAAUCCCCCGUCAUGAGUUCAUCGUUGAUAAACCAUCGAUAGCUGAGCUCGUGAGGAUUGGCAUCCGCCUG -.............................(((((....(((((((((((.(((((((.....)))))).).))))))))))))))))(((....))).. ( -26.10) >consensus _U___G_UGCACUUACCAUUUUGGUGGUAAAGUCCCCGGUCAUGAGCUCAUCGUUGAUGAACCAACGAUAGCUGAGCUCAUGCGGAUUGGCAUCCGCCUG ............(((((((....))))))).....(((..((((((((((((((((......))))))....)))))))))))))...(((....))).. (-21.66 = -24.25 + 2.59)

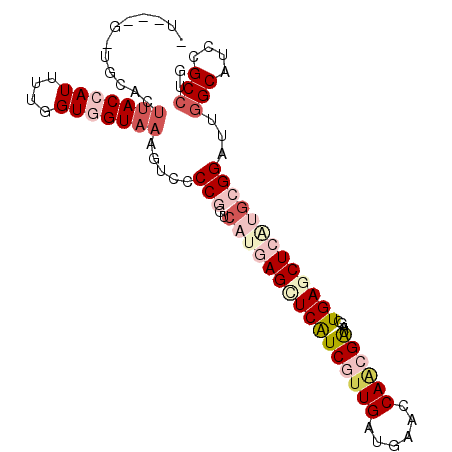
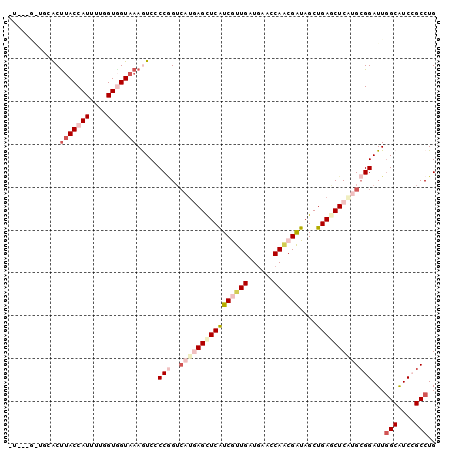
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:59:29 2006