
| Sequence ID | 4_DroMel_CAF1 |
|---|---|
| Location | 142,681 – 142,834 |
| Length | 153 |
| Max. P | 0.866595 |

| Location | 142,681 – 142,794 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | reverse |
| Mean pairwise identity | 78.11 |
| Mean single sequence MFE | -36.01 |
| Consensus MFE | -21.39 |
| Energy contribution | -20.53 |
| Covariance contribution | -0.86 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.791683 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
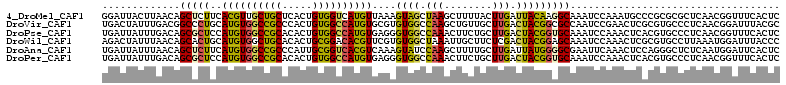
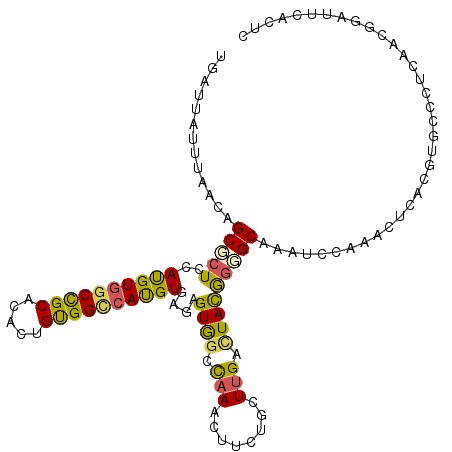
>4_DroMel_CAF1 142681 113 - 1281640 GGAUUACUUAACAGCUCUUCACGUUGCUGCUCACUGUGGUCAUGUUAAAGUAGCUAAGCUUUUACUUGAUUACAAGGCAAAUCCAAAUGCCCGCGCGCUCAACGGUUUCACUC .....................(((((..((.(..((((((((.((.(((((......))))).)).)))))))).((((........))))...).)).)))))......... ( -26.10) >DroVir_CAF1 3528 113 - 1 UGACUAUUUGACGGCCCUGCAUGUGGCCGCCCACUGUGGCCAUGUGCGUGUGGCCAAGCUGUUGCUUGACUACGGCGCCAAUCCGAACUCGCGUGCCCUCAACGGAUUUACGC ............(((((.((((((((((((.....))))))))))))..((((.(((((....))))).)))))).)))...........(((((.((.....))...))))) ( -45.80) >DroPse_CAF1 3542 113 - 1 UGAUUAUUUGACAGCGCUCCAUGUGGCCGCACACUGUGGCCAUGUGAGGGUGGCCAAACUUCUGCUUGACUACGGUGCAAAUCCAAACUCACGUGCCCUCAACGGUUUCACUC (((....((((..((((.....((((((((.....))))))))(((((..(((.(((.(....).)))((....))......)))..)))))))))..)))).....)))... ( -37.00) >DroWil_CAF1 38760 113 - 1 AGACUAUUUAACAGCACUGCAUGUGGCUGCACACUGCGGACACGUUCGUGUGGCUAAAUUGCUUCUCGACUACGGAGCAAAUCCAAACUCGCGUGCCUUAAAUGGAUUUACCC ...((((((((..((((.((...((((..(((...(((....)))..)))..))))..(((((((........)))))))..........)))))).))))))))........ ( -36.70) >DroAna_CAF1 47772 113 - 1 UGAUUAUUUAACAGCUCUUCAUGUGGCCGCCCAUUGCGGUCACGUCAAAGUAUCCAAGCUUUUGCUUGAUUAUGGGGCGAAUUCAAACUCCAGGGCUCUCAAUGGAUUCACUC .............(((((..((((((((((.....))))))))))....(((..(((((....)))))..))))))))(((((((.................))))))).... ( -33.43) >DroPer_CAF1 3705 113 - 1 UGAUUAUUUGACAGCGCUCCAUGUGGCCGCACACUGUGGCCAUGUGAGGGUGGCCAAACUUCUGCUUGACUACGGUGCAAAUCCAAACUCACGUGCCCUCAACGGUUUCACUC (((....((((..((((.....((((((((.....))))))))(((((..(((.(((.(....).)))((....))......)))..)))))))))..)))).....)))... ( -37.00) >consensus UGAUUAUUUAACAGCGCUCCAUGUGGCCGCACACUGUGGCCAUGUGAGAGUGGCCAAACUUCUGCUUGACUACGGGGCAAAUCCAAACUCACGUGCCCUCAACGGAUUCACUC .............(((((..((((((((((.....))))))))))....((((.(((........))).)))))))))................................... (-21.39 = -20.53 + -0.86)
| Location | 142,714 – 142,834 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.67 |
| Mean single sequence MFE | -40.60 |
| Consensus MFE | -19.58 |
| Energy contribution | -19.08 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.46 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.48 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.866595 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
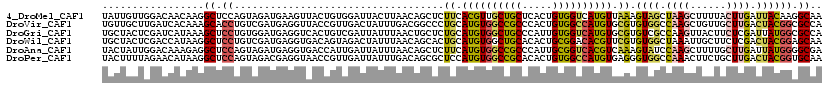
>4_DroMel_CAF1 142714 120 - 1281640 UAUUGUUGGACAACAAGGCUCCAGUAGAUGAAGUUACUGUGGAUUACUUAACAGCUCUUCACGUUGCUGCUCACUGUGGUCAUGUUAAAGUAGCUAAGCUUUUACUUGAUUACAAGGCAA ..(((((....))))).(((.((((((.(((((...((((..........))))..)))))..)))))).....((((((((.((.(((((......))))).)).)))))))).))).. ( -30.80) >DroVir_CAF1 3561 120 - 1 UGUUGCUUGAUCACAAAGCACCUGUCGAUGAGGUUACCGUUGACUAUUUGACGGCCCUGCAUGUGGCCGCCCACUGUGGCCAUGUGCGUGUGGCCAAGCUGUUGCUUGACUACGGCGCCA .(((((((.......))))....(((((((.(....)))))))).....)))(((((.((((((((((((.....))))))))))))..((((.(((((....))))).)))))).))). ( -53.00) >DroGri_CAF1 4051 120 - 1 UGCUACUCGAUCAUAAAGCUCCUGUGGAUGAGGUCACUGUCGAUUAUUUAACUGCUCUGCAUGUGGCUGCCCAUUGUGGUCAUGUGCGUGUCGCCAAGUUACUUCUCGAUUAUGGCGCCA .((..((((.(((((.......))))).))))((((..(((((.....(((((((.(((((((((((..(.....)..)))))))))).)..))..)))))....)))))..)))))).. ( -36.40) >DroWil_CAF1 38793 120 - 1 UGCUACUCGACCAUAAGGCUCCUGUCGAUGAGGUGACAGUAGACUAUUUAACAGCACUGCAUGUGGCUGCACACUGCGGACACGUUCGUGUGGCUAAAUUGCUUCUCGACUACGGAGCAA .................(((((.(((((.((((..(..((((.((.......))..))))...((((..(((...(((....)))..)))..))))..)..)))))))))...))))).. ( -40.30) >DroAna_CAF1 47805 120 - 1 UACUAUUGGACAAAGAGGCUCCAGUAGAUGAGGUGACCAUUGAUUAUUUAACAGCUCUUCAUGUGGCCGCCCAUUGCGGUCACGUCAAAGUAUCCAAGCUUUUGCUUGAUUAUGGGGCGA ..(((((((((......).))))))))((((((.(.(..((((....))))..)).))))))((((((((.....))))))))(((...(((..(((((....)))))..)))..))).. ( -41.40) >DroPer_CAF1 3738 120 - 1 UACUUUUAGAACAUAAGGCUCCAGUAGACGAGGUAACCGUUGAUUAUUUGACAGCGCUCCAUGUGGCCGCACACUGUGGCCAUGUGAGGGUGGCCAAACUUCUGCUUGACUACGGUGCAA .................((.((.((((.((((......((..(....)..))..((((((((((((((((.....))))))))))).)))))............)))).)))))).)).. ( -41.70) >consensus UACUACUCGACCAUAAGGCUCCAGUAGAUGAGGUGACCGUUGAUUAUUUAACAGCUCUGCAUGUGGCCGCCCACUGUGGUCAUGUGAGUGUGGCCAAGCUUCUGCUUGACUACGGCGCAA .................((.((...................................((.((((((((((.....)))))))))).)).((((.((((......)))).)))))).)).. (-19.58 = -19.08 + -0.50)
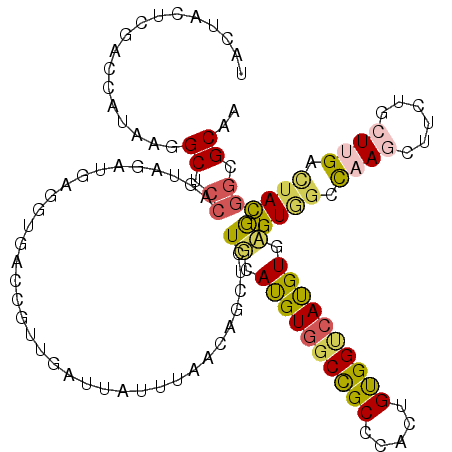
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:31:00 2006