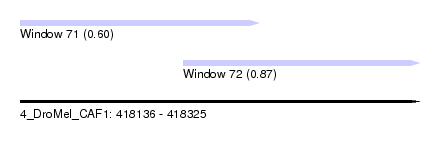
| Sequence ID | 4_DroMel_CAF1 |
|---|---|
| Location | 418,136 – 418,325 |
| Length | 189 |
| Max. P | 0.868804 |

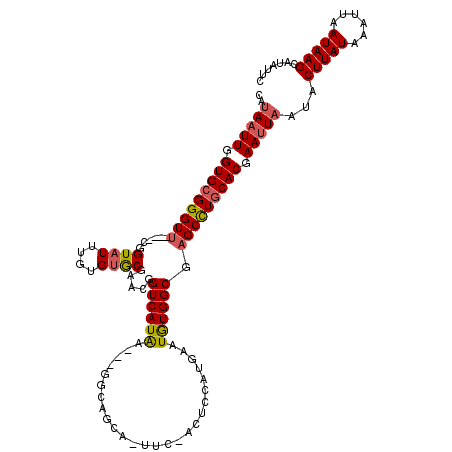
| Location | 418,136 – 418,249 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.01 |
| Mean single sequence MFE | -34.00 |
| Consensus MFE | -20.02 |
| Energy contribution | -20.86 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.16 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.13 |
| SVM RNA-class probability | 0.598218 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>4_DroMel_CAF1 418136 113 + 1281640 AGUGUACCAAAUGCACUUUAGCAUAUGUAUGUGU-UGCAUGUUGAGUGUGUGUUUUGUGUGCACAU-GCAUGGUAAAC-UCAUAAUUGGUGCGGGUU----CGGUAUUUGUGGGCGAACG ..((((((((((((......))))...((((.((-(.(((((...(((((..(.....)..)))))-)))))...)))-.)))).)))))))).(((----((.(.......).))))). ( -34.60) >DroSec_CAF1 35043 118 + 1 UGUGUACCAAAUGCACUUUAGCAUAUGUAUGUGU-UGCAUGUUGAGUGUGU-UUUUGUUUGCACAUUGCAUGGUAAAUAUAAUAAUUGGUGCGGGUAUUUGUGGUAUUUGUGUGCGAACG ........((((((((((....(((((((.....-))))))).))))))))-))..((((((((((.((((.(.............).))))((((((.....)))))))))))))))). ( -31.42) >DroSim_CAF1 26661 112 + 1 UGUGUACCAAAUGCACUUUAGCAUAUGCAUGUGU-UGCAUGUUGAGUGUGU-UUUUGU-UGCACAU-GCCUGGUAAAUAUAAUAAUUGGUGGGGGUU----CGGUAUUUGUGUGCGAACG ((((((.((((.(((((((((((..((((.....-))))))))))).))))-.)))).-)))))).-.(((.(..(((......)))..).)))(((----((.(((....)))))))). ( -32.80) >DroEre_CAF1 44046 113 + 1 AGUGUACCAAAUGCACUUGUGCAUAUGCAUGUGUGUGCAUGCUGAGUUUGU-GUUAGAAUGCACAU-GUAUGGUAAAC-UCAUAAUUGGUGCGGGUU----CGGUAUUUGUGUACGAACG ..(((((((((((((....)))))...((((..(((((((.((((......-.)))).))))))).-.))))......-......)))))))).(((----((.(((....)))))))). ( -39.30) >DroYak_CAF1 46418 113 + 1 AGUGUACCAAAAGCACUUAUGCAUAUGGAUGUGUGUGUAGUUUGAGUGUGU-GUUUGAUUGCACAU-GUAUGGUAAAC-UCAGAAUUGGUGCGGGUU----CGGUAUUUGUGUGCUAACG ..((((((((......((((((((((....))))))))))(((((((((((-((......))))))-.........))-))))).)))))))).(((----.(((((....)))))))). ( -31.90) >consensus AGUGUACCAAAUGCACUUUAGCAUAUGCAUGUGU_UGCAUGUUGAGUGUGU_UUUUGUUUGCACAU_GCAUGGUAAAC_UCAUAAUUGGUGCGGGUU____CGGUAUUUGUGUGCGAACG ...(((((((((((......((((((((((((....))))))...))))))........(((((.....((((......)))).....)))))..........))))))).))))..... (-20.02 = -20.86 + 0.84)

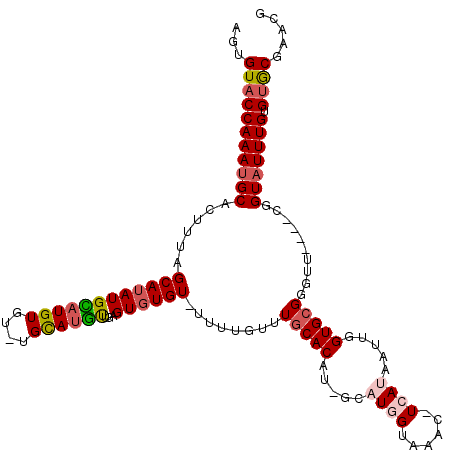
| Location | 418,213 – 418,325 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.64 |
| Mean single sequence MFE | -33.64 |
| Consensus MFE | -21.39 |
| Energy contribution | -21.67 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.56 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.86 |
| SVM RNA-class probability | 0.868804 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>4_DroMel_CAF1 418213 112 + 1281640 CAUAAUUGGUGCGGGUU----CGGUAUUUGUGGGCGAACGGUCAUAA---GGCAGCACUUCCACUCCAUGACUGUGGCGAGCCUGCACGAA-UAAUACUUAUAAAUUAAUAAGCAUAUUC ........(((((((((----((......(((((.(....(((....---)))....).))))).((((....)))))))))))))))(((-((...(((((......)))))..))))) ( -34.80) >DroSec_CAF1 35121 114 + 1 AAUAAUUGGUGCGGGUAUUUGUGGUAUUUGUGUGCGAACGGUCAUGAGGAGGCAGC------ACUCCAUGAGUAUGGCGAGCCUGCACGAAUUAAUACUUAUAAAUUAAUAAGCAUAUUC ..(((((.((((((((..((((.(((((..(((....))........((((.....------.)))))..))))).)))))))))))).)))))...(((((......)))))....... ( -35.50) >DroSim_CAF1 26737 107 + 1 AAUAAUUGGUGGGGGUU----CGGUAUUUGUGUGCGAACGGUCAUAA---GGCAGC------ACUCCAUGAGUAUGGCGAGCCUGCACGAAUUAAUACUUAUAAAUUAAUAAGCAUAUUC ..(((((.(((.(((((----(((((((..(((((.....(((....---))).))------))...)..)))))..))))))).))).)))))...(((((......)))))....... ( -31.80) >DroEre_CAF1 44123 113 + 1 CAUAAUUGGUGCGGGUU----CGGUAUUUGUGUACGAACGGUCAUAA---UGCUGCACUUCCACUCCGUGUAUGUGGCGAGCUUGCACGAAUUAAUACUUAUAAAUUAAUAAGCAUAUUC ..(((((.(((((((((----(.((((....)))).....((((((.---...(((((.........))))))))))))))))))))).)))))...(((((......)))))....... ( -32.00) >DroYak_CAF1 46495 107 + 1 CAGAAUUGGUGCGGGUU----CGGUAUUUGUGUGCUAACGGUCAUAA---UGUUGCAGUUCAAC------UAUGUGGCGAGCCUGCACGAAUUAAUACUUAUAAAUUAAUAAGCAUAUUC ...((((.(((((((((----((((((....)))))....((((((.---.((((.....))))------..)))))))))))))))).))))....(((((......)))))....... ( -34.10) >consensus CAUAAUUGGUGCGGGUU____CGGUAUUUGUGUGCGAACGGUCAUAA___GGCAGCA_UUC_ACUCCAUGAAUGUGGCGAGCCUGCACGAAUUAAUACUUAUAAAUUAAUAAGCAUAUUC ..(((((.(((((((((......((((....)))).....((((((..........................)))))).))))))))).)))))...(((((......)))))....... (-21.39 = -21.67 + 0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:32:26 2006