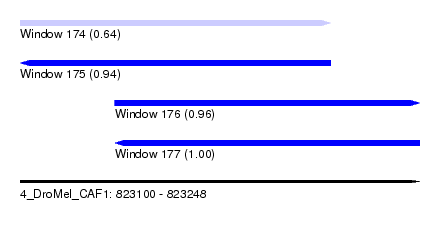
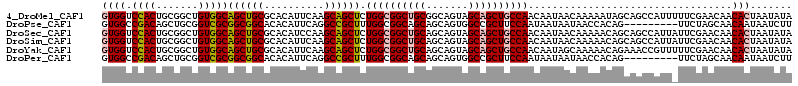
| Sequence ID | 4_DroMel_CAF1 |
|---|---|
| Location | 823,100 – 823,248 |
| Length | 148 |
| Max. P | 0.995790 |

| Location | 823,100 – 823,215 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 82.23 |
| Mean single sequence MFE | -40.52 |
| Consensus MFE | -27.74 |
| Energy contribution | -27.52 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.75 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.23 |
| SVM RNA-class probability | 0.642310 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
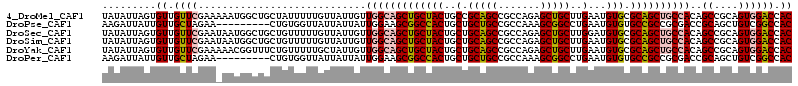
>4_DroMel_CAF1 823100 115 + 1281640 UAUAUUAGUGUUGUUCGAAAAAUGGCUGCUAUUUUUGUUAUUGUUGGCAGCUGCUACUGCCGCAGCCGCCAGAGCUGCUUGAAUGUGCGCAGCUGCCACAGCCGCAGUGGACCAC .......(((..(((((.....((((((................(((((((((((((..(.((((((....).)))))..)...))).))))))))))))))))...)))))))) ( -40.79) >DroPse_CAF1 2554 106 + 1 AAGAUUAUUGUUGCUAGAA---------CUGUGGUUAUUAUUAUUGGAAGCGGCCACUGCUGCUGCCGCCAAAGCGGCCUGAAUGUGUGCCGCCGCGACCGCAGCUGUCGGCCAC .........((((.(((..---------((((((((.......((((.((((((....))))))....)))).((((((.......).)))))...))))))))))).))))... ( -37.00) >DroSec_CAF1 1911 115 + 1 UAUAUUAGUGUUGUUCGAAUAAUGGCUGCUGUUUUUGUUAUUGUUGGCAGCUGCUACUGCUGCAGCCGCCAGAGCUGCUUGGAUGUGCGCAGCUGCCACAGCCGCAGUGGACCAC ......................((((..((((..((((.......(((((((((((((.(.((((((....).)))))..).).))).)))))))))))))..))))..).))). ( -42.31) >DroSim_CAF1 1571 115 + 1 UAUAUUAGUGUUGUUCGAAUAAUGGCUGCUGUUUUUGUUAUUGUUGGCAGCUGCUACUGCUGCAGCCGCCAGAGCUGCUUGAAUGUGCGCAGCUGCCACAGCCGCAGUGGACCAC ......................((((..((((..((((.......((((((((((((..(.((((((....).)))))..)...))).)))))))))))))..))))..).))). ( -42.51) >DroYak_CAF1 1555 115 + 1 UAUAUUAGUGUUGUUCGAAAAACGGUUUCUGUUUUUGCUAUUGUUGGCAGCUGCUACUGCUGCAGCCGCCAGAGCUGCUUGAAUGUGCGCAGCUGCCACAGCCGCAGUGGACCAC .......(((..(((((((((((((...))))))))((..((((.((((((((((((..(.((((((....).)))))..)...))).)))))))))))))..))..)))))))) ( -43.50) >DroPer_CAF1 2185 106 + 1 AAGAUUAUUGUUGCUAGAA---------CUGUGGUUAUUAUUAUUGGAAGCGGCCACUGCUGCUGCCGCCAAAGCGGCCUGAAUGUGUGCCGCCGCGACCGCAGCUGUCGGCCAC .........((((.(((..---------((((((((.......((((.((((((....))))))....)))).((((((.......).)))))...))))))))))).))))... ( -37.00) >consensus UAUAUUAGUGUUGUUCGAA_AAUGGCUGCUGUUUUUGUUAUUGUUGGCAGCUGCUACUGCUGCAGCCGCCAGAGCUGCUUGAAUGUGCGCAGCUGCCACAGCCGCAGUGGACCAC .........((.((((............................(((((((((((((..(.(((((.......)))))..)...))).))))))))))..((....)))))).)) (-27.74 = -27.52 + -0.22)
| Location | 823,100 – 823,215 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 82.23 |
| Mean single sequence MFE | -41.75 |
| Consensus MFE | -31.06 |
| Energy contribution | -30.95 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.31 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.939354 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>4_DroMel_CAF1 823100 115 - 1281640 GUGGUCCACUGCGGCUGUGGCAGCUGCGCACAUUCAAGCAGCUCUGGCGGCUGCGGCAGUAGCAGCUGCCAACAAUAACAAAAAUAGCAGCCAUUUUUCGAACAACACUAAUAUA (..(....)..)((((((((.((((((..........))))))))(((((((((.......)))))))))................))))))....................... ( -42.80) >DroPse_CAF1 2554 106 - 1 GUGGCCGACAGCUGCGGUCGCGGCGGCACACAUUCAGGCCGCUUUGGCGGCAGCAGCAGUGGCCGCUUCCAAUAAUAAUAACCACAG---------UUCUAGCAACAAUAAUCUU (((((((...(((((.(((((((((((.(.......)))))))...))))).)))))..))))))).....................---------................... ( -41.00) >DroSec_CAF1 1911 115 - 1 GUGGUCCACUGCGGCUGUGGCAGCUGCGCACAUCCAAGCAGCUCUGGCGGCUGCAGCAGUAGCAGCUGCCAACAAUAACAAAAACAGCAGCCAUUAUUCGAACAACACUAAUAUA (..(....)..)((((((((.((((((..........))))))))(((((((((.......)))))))))................))))))....................... ( -42.10) >DroSim_CAF1 1571 115 - 1 GUGGUCCACUGCGGCUGUGGCAGCUGCGCACAUUCAAGCAGCUCUGGCGGCUGCAGCAGUAGCAGCUGCCAACAAUAACAAAAACAGCAGCCAUUAUUCGAACAACACUAAUAUA (..(....)..)((((((((.((((((..........))))))))(((((((((.......)))))))))................))))))....................... ( -42.10) >DroYak_CAF1 1555 115 - 1 GUGGUCCACUGCGGCUGUGGCAGCUGCGCACAUUCAAGCAGCUCUGGCGGCUGCAGCAGUAGCAGCUGCCAACAAUAGCAAAAACAGAAACCGUUUUUCGAACAACACUAAUAUA (((.((....((((((((.((((((((..........)))))).((((((((((.......))))))))))......))....))))...)))).....))....)))....... ( -41.50) >DroPer_CAF1 2185 106 - 1 GUGGCCGACAGCUGCGGUCGCGGCGGCACACAUUCAGGCCGCUUUGGCGGCAGCAGCAGUGGCCGCUUCCAAUAAUAAUAACCACAG---------UUCUAGCAACAAUAAUCUU (((((((...(((((.(((((((((((.(.......)))))))...))))).)))))..))))))).....................---------................... ( -41.00) >consensus GUGGUCCACUGCGGCUGUGGCAGCUGCGCACAUUCAAGCAGCUCUGGCGGCUGCAGCAGUAGCAGCUGCCAACAAUAACAAAAACAGCAGCCAUU_UUCGAACAACACUAAUAUA ((((.((((.......)))))((((((..........)))))).((((((((((.......))))))))))..................................)))....... (-31.06 = -30.95 + -0.11)
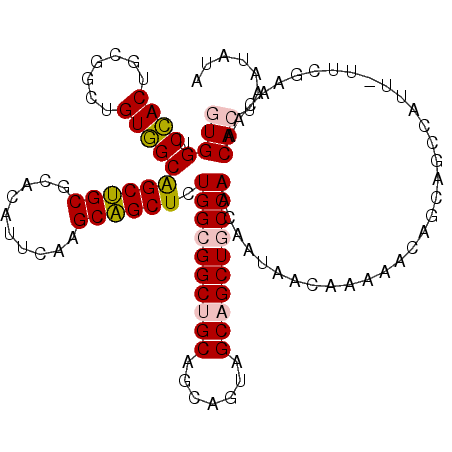
| Location | 823,135 – 823,248 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 75.65 |
| Mean single sequence MFE | -47.19 |
| Consensus MFE | -30.37 |
| Energy contribution | -28.82 |
| Covariance contribution | -1.55 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.96 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.55 |
| SVM RNA-class probability | 0.962946 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
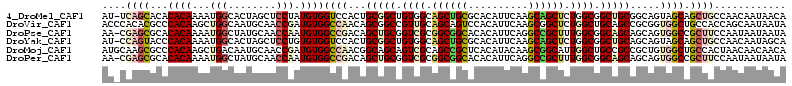
>4_DroMel_CAF1 823135 113 + 1281640 UGUUAUUGUUGGCAGCUGCUACUGCCGCAGCCGCCAGAGCUGCUUGAAUGUGCGCAGCUGCCACAGCCGCAGUGGACCACAUAGGAGCUAGUGCCAUUUUGUGUGUGCUGA-AU .........(((((((((((((..(.((((((....).)))))..)...))).))))))))))((((((((((((..(((.(((...))))))))))....)))).)))).-.. ( -44.50) >DroVir_CAF1 7494 114 + 1 UAUUAUUGCUGGUGGCAGCCACCGCGGCUGCAGCCAGAGCCGCUUGAAUGUGGACUGCUGCAACGGCCGCUGUUGGCCACAUCGGUUGCAUUGCCAGCUUGUGGGCGUGUGGGU ........(((((.((((((.....)))))).)))))((((((......)))).)).(..((.((.((((.((((((..((.....))....))))))..)))).))))..).. ( -49.90) >DroPse_CAF1 2580 113 + 1 UAUUAUUAUUGGAAGCGGCCACUGCUGCUGCCGCCAAAGCGGCCUGAAUGUGUGCCGCCGCGACCGCAGCUGUCGGCCACAUUGGUUGCAUAGCCAUUUUGUGUGCGCUCG-UU ..............((((((((.......(((((....)))))......))).))))).((((.((((((((((((((.....))))).)))))(((...))))))).)))-). ( -42.12) >DroYak_CAF1 1590 113 + 1 UGCUAUUGUUGGCAGCUGCUACUGCUGCAGCCGCCAGAGCUGCUUGAAUGUGCGCAGCUGCCACAGCCGCAGUGGACCACACAGGAGCUAGUGCCAUUUUGUGGGUACUGG-AU (((..((((.((((((((((((..(.((((((....).)))))..)...))).)))))))))))))..)))(((.....))).....((((((((........))))))))-.. ( -50.30) >DroMoj_CAF1 2562 114 + 1 UGUUGUUGUUAGUGGCAGCCACAGCGGCGGCAGCCAAUGCCGCUUGUAUGUGAGCGGCUGCGACUGCUGCCGUUGGCCACAUCGGUUGCAUUGUCAGCUUGUGGGCGCUUGCAU ..............(((((..((((((((((((.(...(((((((......)))))))...).))))))))))))(((.(((.(((((......))))).)))))))).))).. ( -54.20) >DroPer_CAF1 2211 113 + 1 UAUUAUUAUUGGAAGCGGCCACUGCUGCUGCCGCCAAAGCGGCCUGAAUGUGUGCCGCCGCGACCGCAGCUGUCGGCCACAUUGGUUGCAUAGCCAUUUUGUGUGCGCUCG-UU ..............((((((((.......(((((....)))))......))).))))).((((.((((((((((((((.....))))).)))))(((...))))))).)))-). ( -42.12) >consensus UAUUAUUGUUGGAAGCAGCCACUGCUGCUGCCGCCAAAGCCGCUUGAAUGUGCGCAGCUGCGACAGCCGCUGUCGGCCACAUAGGUUGCAUUGCCAUUUUGUGGGCGCUCG_AU ........(((((.(((((.......))))).))))).(((((..........)))))..((((.......))))((((((..((........))....)))).))........ (-30.37 = -28.82 + -1.55)

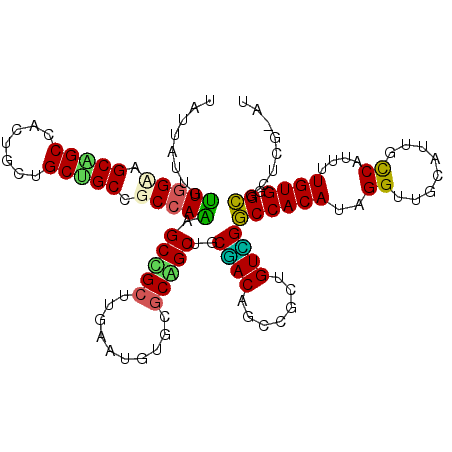
| Location | 823,135 – 823,248 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 75.65 |
| Mean single sequence MFE | -45.38 |
| Consensus MFE | -28.32 |
| Energy contribution | -29.47 |
| Covariance contribution | 1.14 |
| Combinations/Pair | 1.37 |
| Mean z-score | -3.07 |
| Structure conservation index | 0.62 |
| SVM decision value | 2.62 |
| SVM RNA-class probability | 0.995790 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
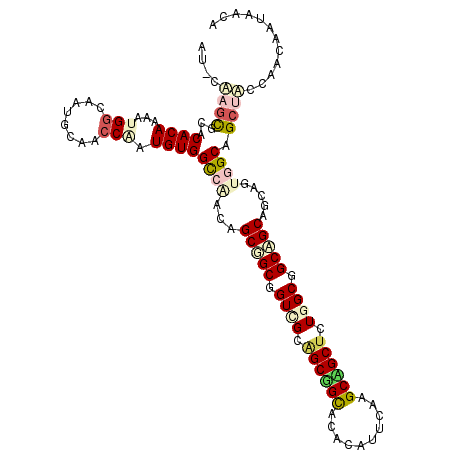
>4_DroMel_CAF1 823135 113 - 1281640 AU-UCAGCACACACAAAAUGGCACUAGCUCCUAUGUGGUCCACUGCGGCUGUGGCAGCUGCGCACAUUCAAGCAGCUCUGGCGGCUGCGGCAGUAGCAGCUGCCAACAAUAACA ..-...(.((.((((....(((....)))....)))))).)...((((((((.(.((((((..........)))))).).))))))))((((((....)))))).......... ( -42.40) >DroVir_CAF1 7494 114 - 1 ACCCACACGCCCACAAGCUGGCAAUGCAACCGAUGUGGCCAACAGCGGCCGUUGCAGCAGUCCACAUUCAAGCGGCUCUGGCUGCAGCCGCGGUGGCUGCCACCAGCAAUAAUA ................(((((...((((((......((((......))))))))))((((.((((......(((((((.....).)))))).))))))))..)))))....... ( -45.00) >DroPse_CAF1 2580 113 - 1 AA-CGAGCGCACACAAAAUGGCUAUGCAACCAAUGUGGCCGACAGCUGCGGUCGCGGCGGCACACAUUCAGGCCGCUUUGGCGGCAGCAGCAGUGGCCGCUUCCAAUAAUAAUA ..-.(((((..(((....(((((((.........)))))))...(((((.(((((((((((.(.......)))))))...))))).))))).)))..)))))............ ( -46.30) >DroYak_CAF1 1590 113 - 1 AU-CCAGUACCCACAAAAUGGCACUAGCUCCUGUGUGGUCCACUGCGGCUGUGGCAGCUGCGCACAUUCAAGCAGCUCUGGCGGCUGCAGCAGUAGCAGCUGCCAACAAUAGCA ..-(((((..(((((....(((....)))....)))))...)))).)(((((((.((((((..........))))))))(((((((((.......)))))))))....))))). ( -46.70) >DroMoj_CAF1 2562 114 - 1 AUGCAAGCGCCCACAAGCUGACAAUGCAACCGAUGUGGCCAACGGCAGCAGUCGCAGCCGCUCACAUACAAGCGGCAUUGGCUGCCGCCGCUGUGGCUGCCACUAACAACAACA .(((((((........))).....))))......(..((((.((((.(((((((..((((((........))))))..))))))).))))...))))..).............. ( -45.60) >DroPer_CAF1 2211 113 - 1 AA-CGAGCGCACACAAAAUGGCUAUGCAACCAAUGUGGCCGACAGCUGCGGUCGCGGCGGCACACAUUCAGGCCGCUUUGGCGGCAGCAGCAGUGGCCGCUUCCAAUAAUAAUA ..-.(((((..(((....(((((((.........)))))))...(((((.(((((((((((.(.......)))))))...))))).))))).)))..)))))............ ( -46.30) >consensus AU_CAAGCGCACACAAAAUGGCAAUGCAACCAAUGUGGCCAACAGCGGCGGUCGCAGCGGCACACAUUCAAGCAGCUCUGGCGGCAGCAGCAGUGGCAGCUACCAACAAUAACA ....((((...((((...(((........))).))))((((...(((((.((((.((((((..........)))))).)))).))))).....)))).))))............ (-28.32 = -29.47 + 1.14)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:34:58 2006