
| Sequence ID | 4_DroMel_CAF1 |
|---|---|
| Location | 787,433 – 787,593 |
| Length | 160 |
| Max. P | 0.946946 |

| Location | 787,433 – 787,553 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.06 |
| Mean single sequence MFE | -38.50 |
| Consensus MFE | -23.99 |
| Energy contribution | -25.25 |
| Covariance contribution | 1.26 |
| Combinations/Pair | 1.57 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.21 |
| SVM RNA-class probability | 0.930458 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
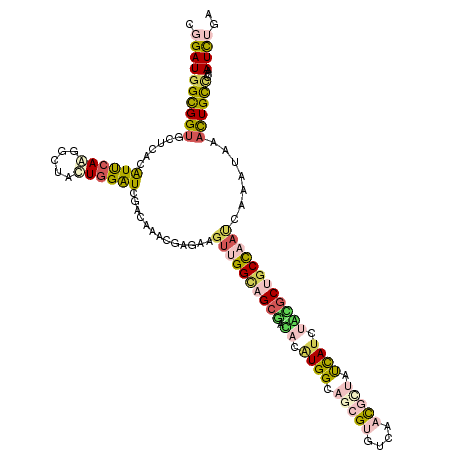
>4_DroMel_CAF1 787433 120 + 1281640 CGGAUGGCGGUGCUCACAUUCAAGGCUAUUGGAUCGAUAAGCGUGAAGUAGGCAGCAACACAUGGCAGCGUGUAAACGCUACCAUCUGUGCUGCCAAUCAAAUAAACUGUAUCAAUCUCA .((((.(((((....((.((((..(((((((...)))).))).)))))).(((((((.((.((((.(((((....))))).)))).)))))))))..........)))))....)))).. ( -41.40) >DroVir_CAF1 17132 120 + 1 CGGAUGGUGGUGCACAUAUUCAAGGCUAUUGGGUUGACAAGCGUGAAGUUGGCAGCGAUACCUGGCAACGGGUUAACCCCAUCAUUAACGCUGCCAACCAAAUUAACUGCCAUAAUCUGA (((((.(((((.......((((..(((..((......))))).))))((((((((((..(((((....)))))(((........))).))))))))))..........))))).))))). ( -41.90) >DroGri_CAF1 18081 120 + 1 CUGAUGGCGGUGCUCAUGUCCAGGGCUACUGGGUGGACAAACGGGAACUUGGCAGCGAUACGUGGCAGCGAAUCAAUAAUAUUAUUCAGGCAACCAAUCAAAUCAAUUGCCACAAUCUCA .((((..((.(((.(((((.....(((.(..(((...(....)...)))..).)))...)))))))).)).)))).............(((((.............)))))......... ( -28.82) >DroWil_CAF1 16034 120 + 1 CGGAUGGCGGUGCUCACAUUCAAGGCUACUGGAUCGACAAACGAGAAGUUGGCAGCGAGACAUGGCAGCGUGUCAACGCUAUCAUCUCCGCUGCUAAUCAGAUCAACUGCCCCAAUCUGA ((((((((((((((.........)))..(((..(((.....)))...((((((((((.((.(((..(((((....)))))..))).)))))))))))))))....))))))...))))). ( -45.30) >DroAna_CAF1 20059 120 + 1 CUGAUGGCGGCUCUCACAUUCAAGGCUAUUGGAUUGAUAAACGCGAAGCGGGUAGUGACACAUGGCAGCGUGUGAAUGUUACCAUUUGCUCAGCCACUCAAAUAAACUGCCCACAUUUAA ..(((((((..(.(((.((((((.....))))))))).)..))).....(((((((......((((((((.(((........))).))))..)))).........))))))).))))... ( -33.06) >DroPer_CAF1 15610 120 + 1 CGGAUGGCGGUGCUCACAUUCAAGGCUACUUGGUCGAUAAACGAGAGGUUGGUAGCGACACAUGGCAGCGUGUGAACGCAAUCAUCUCUGCUGCCAACCAAAUAAACUGUUCCAAUCUGA (((((.(((((.(((.....((((....))))((......))))).(((((((((((....((((..((((....))))..))))...)))))))))))......)))))....))))). ( -40.50) >consensus CGGAUGGCGGUGCUCACAUUCAAGGCUACUGGAUCGACAAACGAGAAGUUGGCAGCGACACAUGGCAGCGUGUCAACGCUAUCAUCUACGCUGCCAAUCAAAUAAACUGCCCCAAUCUGA .((((((((((......((((((.....)))))).............((((((((((.((.((((.(((((....))))).)))).)))))))))))).......))))))...)))).. (-23.99 = -25.25 + 1.26)

| Location | 787,473 – 787,593 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.00 |
| Mean single sequence MFE | -38.48 |
| Consensus MFE | -26.60 |
| Energy contribution | -25.05 |
| Covariance contribution | -1.55 |
| Combinations/Pair | 1.48 |
| Mean z-score | -2.56 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.37 |
| SVM RNA-class probability | 0.946946 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
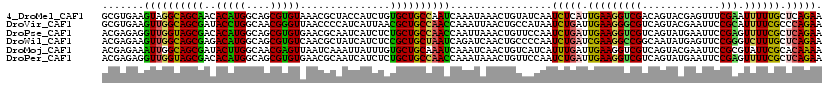
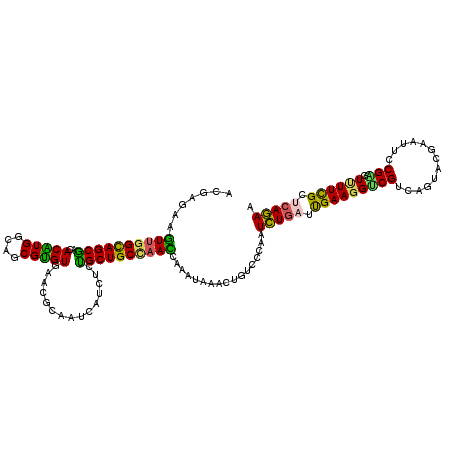
>4_DroMel_CAF1 787473 120 + 1281640 GCGUGAAGUAGGCAGCAACACAUGGCAGCGUGUAAACGCUACCAUCUGUGCUGCCAAUCAAAUAAACUGUAUCAAUCUCAUUGAAGGUCGACAGUACGAGUUUCGAAUUUUUGCUCAGAA ..........(((((((.((.((((.(((((....))))).)))).)))))))))...........(((...(((.....((((((.(((......))).))))))....)))..))).. ( -39.50) >DroVir_CAF1 17172 120 + 1 GCGUGAAGUUGGCAGCGAUACCUGGCAACGGGUUAACCCCAUCAUUAACGCUGCCAACCAAAUUAACUGCCAUAAUCUGAUUGAAGGGCGUCAGUACGAAUUCCGCAUUUUCGCCCAGAA .......((((((((((..(((((....)))))(((........))).))))))))))...........................(((((..(((.((.....)).)))..))))).... ( -37.60) >DroPse_CAF1 15662 120 + 1 ACGAGAGGUUGGUAGCGACACAUGGCAGCGUGUGAACGCAAUCAUCUCUGCUGCCAACCAAUUAAACUGUUCCAAUCUGAUUGAAGGUCGUCAGUAUGAAUUCCGAGUUUUCGCUCAGAA ......(((((((((((....((((..((((....))))..))))...)))))))))))................(((((.(((((((((((.....))....))).)))))).))))). ( -37.30) >DroWil_CAF1 16074 120 + 1 ACGAGAAGUUGGCAGCGAGACAUGGCAGCGUGUCAACGCUAUCAUCUCCGCUGCUAAUCAGAUCAACUGCCCCAAUCUGAUCGAAGGCCGGCAAUAUGAGUUCCGGGUCUUUGCUCAGAA ..((...((((((((((.((.(((..(((((....)))))..))).))))))))))))....))...........(((((.((((((((((.(((....))))).)))))))).))))). ( -49.30) >DroMoj_CAF1 16819 120 + 1 ACGAGAAAUUGGCAGCGAUACUUGGCAACGAGUUAAUCAAAUUAUUUGUGCUGCAAAUCAAAUCAACUGUCAUCAUUUGAUUGAAGGUCGUCAGUACGAAUUCCGCGUAUUCGCACAAAA .(((....)))...((((.(((((....)))))..........((((((((((...(((.((((((.((....)).))))))...)))...)))))))))).........))))...... ( -29.90) >DroPer_CAF1 15650 120 + 1 ACGAGAGGUUGGUAGCGACACAUGGCAGCGUGUGAACGCAAUCAUCUCUGCUGCCAACCAAAUAAACUGUUCCAAUCUGAUUGAAGGUCGUCAGUAUGAAUUCCGAGUUUUCGCUCAGAA ......(((((((((((....((((..((((....))))..))))...)))))))))))................(((((.(((((((((((.....))....))).)))))).))))). ( -37.30) >consensus ACGAGAAGUUGGCAGCGACACAUGGCAGCGUGUGAACGCAAUCAUCUCUGCUGCCAACCAAAUAAACUGUCCCAAUCUGAUUGAAGGUCGUCAGUACGAAUUCCGAGUUUUCGCUCAGAA .......((((((((((..(((((....)))))...............)))))))))).................(((((.(((((((((.............))).)))))).))))). (-26.60 = -25.05 + -1.55)
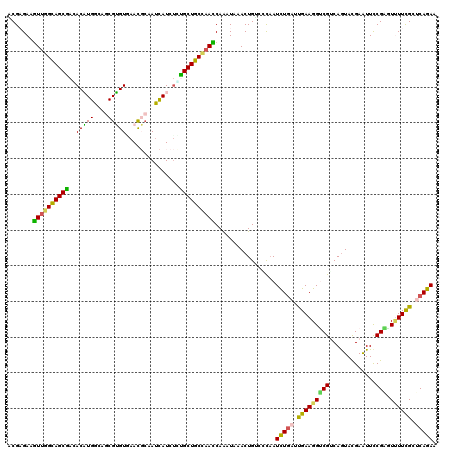
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:34:50 2006