
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 25,815,376 – 25,815,535 |
| Length | 159 |
| Max. P | 0.612977 |

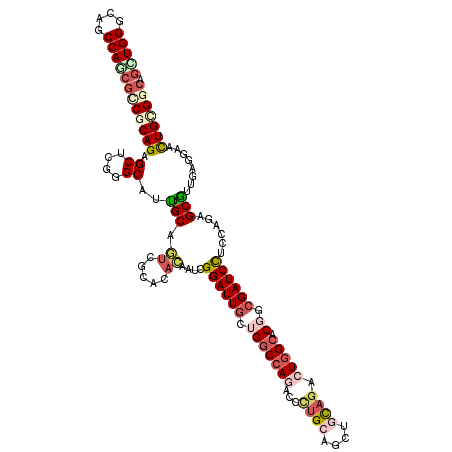
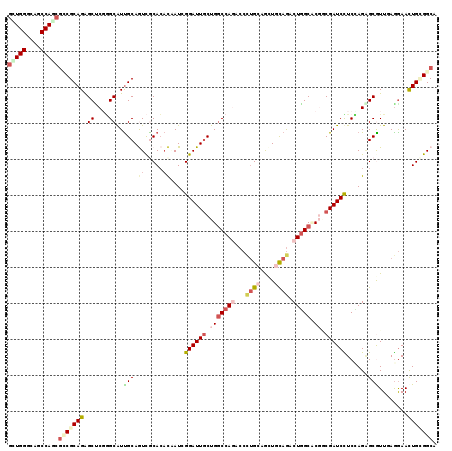
| Location | 25,815,376 – 25,815,495 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.94 |
| Mean single sequence MFE | -49.20 |
| Consensus MFE | -28.49 |
| Energy contribution | -30.22 |
| Covariance contribution | 1.73 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.68 |
| Structure conservation index | 0.58 |
| SVM decision value | -0.00 |
| SVM RNA-class probability | 0.532131 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 25815376 119 - 27905053 ACUGGGCAACCAUCUUCGCAGAGCUCGAGCAUUGCAAUCACACACUAUCGGAUUGCUGGCCAAUCCUUGGAGCUGCAGACUGGCCCUGCGAUCUUCUAGAGCGUUUAAAAACUGUGGCA ..(((....))).....((((.((....)).)))).....(((((((..(((((((.(((((.((..((......)))).)))))..)))))))..)))...(((....)))))))... ( -34.90) >DroVir_CAF1 77198 119 - 1 GCUGGGCAGCCAGCGGCGCAGAGCGCGUGCAUCGCAAUUGCAGUUAAUCGGAUUGCUGGCCAAACCCUGCAGCUGCAGGCUCGCCCGCCGAUCCUCGAGUGCGUUGAGGAAUUGCGGCA ...((((((((.(((((((((...(((.....)))....((((((.....))))))..........)))).))))).)))).))))((((.(((((((.....)))))))....)))). ( -55.40) >DroPse_CAF1 62570 119 - 1 GUUGGGCAGCCAGCGCCGCAGAGCCCGGGCGUUGCAGUCGCACACGAUGGGAUUGCUGGCCAGACCCUGCAGCUGCAGACUGGCACGGCGAUCCUCCAGAGCAUUGAGGAACUGCGGCA (((((....)))))(((((((..((((((((((((....))).))...(((((((((((((((...((((....)))).))))).)))))))))).....)).))).))..))))))). ( -60.80) >DroWil_CAF1 62681 119 - 1 GCUGGGUAGCCAACGUCGCAGGGCUCUGGCAUUGCAGUCGCACACUAUUGGAUUGCUGUCCAGACCCUGCAAUUGUAGACUGGCUCGACGAUCUUCCAGGGCGUUAAGGAAUUGGGGCA (((......((((((((((((((.(((((.((.((((((.((......)))))))).))))))))))))).........((((...(.....)..))))))))))..))......))). ( -44.70) >DroAna_CAF1 61945 119 - 1 GUUGGGCAACCAACGGCGCAGAGCUCUGGCAUUGCAGUCACACACAAUAGGAUUACUGGCCAGUCCGCGAAGCUGUAAGCUGGCUCUUCGAUCUUCGAGAGCGUUUAGGAAUUGAGGCA (((((....)))))(((((((.((....)).)))).)))........(((.....)))(((..(((....(((.....))).(((((.((.....))))))).....))).....))). ( -38.60) >DroPer_CAF1 66067 119 - 1 GUUGGGCAGCCAGCGCCGCAGAGCCCGGGCGUUGCAGUCGCACACGAUGGGAUUGCUGGCCAGACCCUGCAGCUGCAGACUGGCACGGCGAUCCUCCAGAGCAUUGAGGAACUGCGGCA (((((....)))))(((((((..((((((((((((....))).))...(((((((((((((((...((((....)))).))))).)))))))))).....)).))).))..))))))). ( -60.80) >consensus GCUGGGCAGCCAGCGCCGCAGAGCUCGGGCAUUGCAGUCGCACACAAUCGGAUUGCUGGCCAGACCCUGCAGCUGCAGACUGGCACGGCGAUCCUCCAGAGCGUUGAGGAACUGCGGCA (((((....)))))(((((((.((....))..(((.((.....))....((((((.(((((((...((((....)))).))))).)).))))))......)))........))))))). (-28.49 = -30.22 + 1.73)

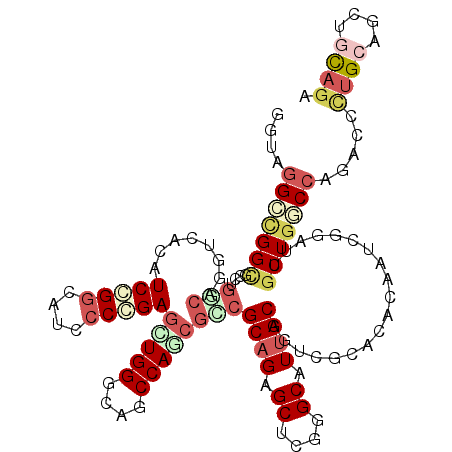
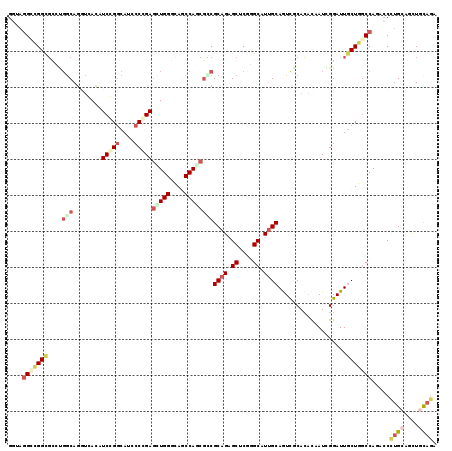
| Location | 25,815,416 – 25,815,535 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 76.47 |
| Mean single sequence MFE | -52.27 |
| Consensus MFE | -27.76 |
| Energy contribution | -29.68 |
| Covariance contribution | 1.92 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.612977 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 25815416 119 - 27905053 GGUAGGCAGGCGAGGCACAGGUGACAUCCGGCAUACCGGAACUGGGCAACCAUCUUCGCAGAGCUCGAGCAUUGCAAUCACACACUAUCGGAUUGCUGGCCAAUCCUUGGAGCUGCAGA .....((..((((((....(....).(((((....)))))..(((....))).))))))..(((((..((...((((((.(........)))))))..))((.....)))))))))... ( -39.70) >DroVir_CAF1 77238 119 - 1 GAUAGGAUGGUUCGCGGCAGACGAGCUCCGGCAUGCCCGAGCUGGGCAGCCAGCGGCGCAGAGCGCGUGCAUCGCAAUUGCAGUUAAUCGGAUUGCUGGCCAAACCCUGCAGCUGCAGG ....(((..((((((....).))))))))(((.(((((.....)))))))).(((((((((...(((.....)))....((((((.....))))))..........)))).)))))... ( -46.90) >DroPse_CAF1 62610 119 - 1 GGAAGGCCGGUGCCUGGCAGGUCACAUCGGGCAUCCCCGAGUUGGGCAGCCAGCGCCGCAGAGCCCGGGCGUUGCAGUCGCACACGAUGGGAUUGCUGGCCAGACCCUGCAGCUGCAGA ....(((((((((.((((.(.(((..(((((....)))))..))).).)))))))))((((..((((..((((((....))).))).)))).)))).)))).....((((....)))). ( -56.80) >DroWil_CAF1 62721 119 - 1 AAUAGGGCGGCGCUUGGCACCUCAGCUCCGACAUGCCGGAGCUGGGUAGCCAACGUCGCAGGGCUCUGGCAUUGCAGUCGCACACUAUUGGAUUGCUGUCCAGACCCUGCAAUUGUAGA ......((((((.((((((((.((((((((......))))))))))).)))))))))((((((.(((((.((.((((((.((......)))))))).)))))))))))))....))... ( -63.60) >DroAna_CAF1 61985 119 - 1 GGUAUGGUGGGGACUGGCAGGUGACAUCCGGCAUCCCCGAGUUGGGCAACCAACGGCGCAGAGCUCUGGCAUUGCAGUCACACACAAUAGGAUUACUGGCCAGUCCGCGAAGCUGUAAG ..(((((((.((((((((.(((((..(((((.....))..(((((....)))))(((((((.((....)).)))).)))..........)))))))).)))))))).)...)))))).. ( -47.90) >DroPer_CAF1 66107 119 - 1 GGAAGGCCGGCGCCUGGCAGGUCACAUCGGGCAUCCCCGAGUUGGGCAGCCAGCGCCGCAGAGCCCGGGCGUUGCAGUCGCACACGAUGGGAUUGCUGGCCAGACCCUGCAGCUGCAGA ....(((((((((.((((.(.(((..(((((....)))))..))).).)))))))))((((..((((..((((((....))).))).)))).)))).)))).....((((....)))). ( -58.70) >consensus GGUAGGCCGGCGCCUGGCAGGUCACAUCCGGCAUCCCCGAGCUGGGCAGCCAGCGCCGCAGAGCUCGGGCAUUGCAGUCGCACACAAUCGGAUUGCUGGCCAGACCCUGCAGCUGCAGA ....(((((((....(((........(((((....)))))(((((....))))))))((((.((....)).))))...................))))))).....((((....)))). (-27.76 = -29.68 + 1.92)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:41:20 2006