
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
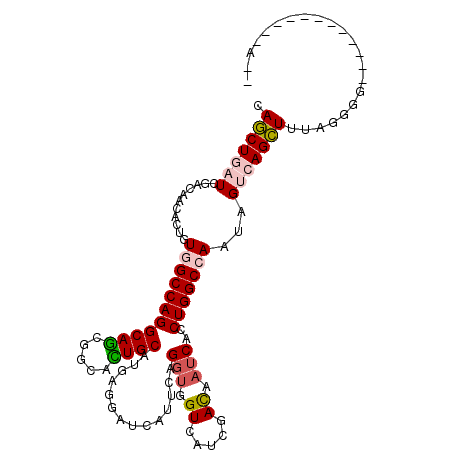
| Location | 25,754,204 – 25,754,308 |
| Length | 104 |
| Max. P | 0.925536 |

| Location | 25,754,204 – 25,754,308 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 73.74 |
| Mean single sequence MFE | -32.42 |
| Consensus MFE | -19.18 |
| Energy contribution | -19.66 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.925536 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 25754204 104 + 27905053 CAGCUGAUCGACAACACGGUGGCCAGGCAGCGCCACUGCUUGAUGAUCAUCCAGCGGGUCAUCGACAACCACCUGGCCAACAGCCAGCUCUAGAGC----UCU-A--GGGC ..((((((((......))))((((((((((.....))))(((((((((........))))))))).......))))))..))))..(((((((...----.))-)--)))) ( -43.10) >DroVir_CAF1 25954 91 + 1 CAGCUAAUUGAUAAUACCAUAGCCAGGCAGCGGGAUUGCAUGCGGAUUAUUCAGGUGGUAAUCGAUAAUCAUCUGGCCAACAGUCAGCUUU-------------------- .((((.((((.((......))(((((((((((....))).))).(((((((..(((....))))))))))..)))))...)))).))))..-------------------- ( -22.30) >DroPse_CAF1 23695 97 + 1 CAGCUGAUCGACAACACUGUGGCCAGGCAGCGGCACUGCAUGAGGAUCAUUCAGGUCGUCAUCGACAAUCAUCUGGCCAAUAGUCAGCUUUAGGGG------------A-- .(((((((..(((....)))((((((((((.....))))((((.((((.....)))).))))..........))))))....))))))).......------------.-- ( -33.70) >DroMoj_CAF1 23472 109 + 1 CAGCUGAUAGACAAUACUAUAGCCAGGCAAAGGGAUUGCAUGCGCAUCAUACAGGUGGUCAUCGACAAUCACCUGGCCAAUAGCCAGCUCUGAGUAAAGCUAACG--GAAU ...(((.(((....((((..(((..(((....((..(((....))).....((((((((........)))))))).))....))).)))...))))...))).))--)... ( -29.50) >DroAna_CAF1 27286 103 + 1 CAACUUAUUGAUAAUACGGUGGCCAGGCAGCGGCACUGCAUGGAGAUUAUCCAGGUGGUGAUUAAUAAUCACCUGGCCAAUAGUCAGUUGUAAGCU--------AGAGCAA ((((((((((......))))).(((.((((.....)))).))).(((((((((((((((........)))))))))...)))))))))))...((.--------...)).. ( -32.20) >DroPer_CAF1 23757 97 + 1 CAGCUGAUCGACAACACUGUGGCCAGGCAGCGGCACUGCAUGAGGAUCAUUCAGGUCGUCAUCGACAAUCAUCUGGCCAAUAGUCAGCUUUAGGGG------------A-- .(((((((..(((....)))((((((((((.....))))((((.((((.....)))).))))..........))))))....))))))).......------------.-- ( -33.70) >consensus CAGCUGAUCGACAACACUGUGGCCAGGCAGCGGCACUGCAUGAGGAUCAUUCAGGUGGUCAUCGACAAUCACCUGGCCAAUAGUCAGCUUUAGGGG____________A__ .(((((((...........(((((((((((.....))))..............(((.((.....)).)))..)))))))...)))))))...................... (-19.18 = -19.66 + 0.47)


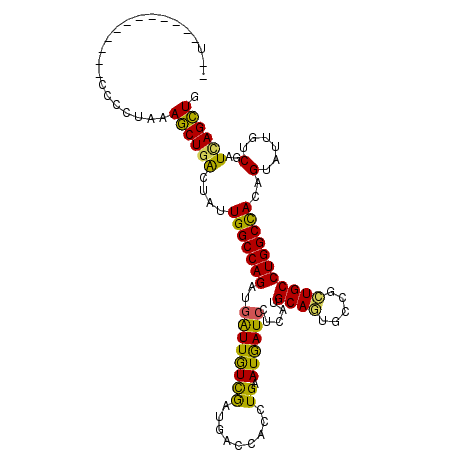
| Location | 25,754,204 – 25,754,308 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 73.74 |
| Mean single sequence MFE | -30.24 |
| Consensus MFE | -22.79 |
| Energy contribution | -21.38 |
| Covariance contribution | -1.41 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.02 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.855287 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 25754204 104 - 27905053 GCCC--U-AGA----GCUCUAGAGCUGGCUGUUGGCCAGGUGGUUGUCGAUGACCCGCUGGAUGAUCAUCAAGCAGUGGCGCUGCCUGGCCACCGUGUUGUCGAUCAGCUG ((.(--(-((.----...)))).)).(((((.((((((((..((.(((...)))((((((..((.....))..)))))).))..)))))))).((......))..))))). ( -43.00) >DroVir_CAF1 25954 91 - 1 --------------------AAAGCUGACUGUUGGCCAGAUGAUUAUCGAUUACCACCUGAAUAAUCCGCAUGCAAUCCCGCUGCCUGGCUAUGGUAUUAUCAAUUAGCUG --------------------..((((((.((.(((((((..((((((((.........)).)))))).(((.((......)))))))))))).........)).)))))). ( -22.90) >DroPse_CAF1 23695 97 - 1 --U------------CCCCUAAAGCUGACUAUUGGCCAGAUGAUUGUCGAUGACGACCUGAAUGAUCCUCAUGCAGUGCCGCUGCCUGGCCACAGUGUUGUCGAUCAGCUG --.------------.......((((((..((((((.........))))))(((((((((.(((.....)))((((.....)))).......))).))))))..)))))). ( -28.00) >DroMoj_CAF1 23472 109 - 1 AUUC--CGUUAGCUUUACUCAGAGCUGGCUAUUGGCCAGGUGAUUGUCGAUGACCACCUGUAUGAUGCGCAUGCAAUCCCUUUGCCUGGCUAUAGUAUUGUCUAUCAGCUG ....--.(((((((((....)))))))))...((((((((..(..(((...)))....((((((.....))))))......)..))))))))................... ( -31.50) >DroAna_CAF1 27286 103 - 1 UUGCUCU--------AGCUUACAACUGACUAUUGGCCAGGUGAUUAUUAAUCACCACCUGGAUAAUCUCCAUGCAGUGCCGCUGCCUGGCCACCGUAUUAUCAAUAAGUUG ......(--------((((((......((...((((((((((((.....))))))...((((.....)))).((((.....)))).))))))..))........))))))) ( -28.04) >DroPer_CAF1 23757 97 - 1 --U------------CCCCUAAAGCUGACUAUUGGCCAGAUGAUUGUCGAUGACGACCUGAAUGAUCCUCAUGCAGUGCCGCUGCCUGGCCACAGUGUUGUCGAUCAGCUG --.------------.......((((((..((((((.........))))))(((((((((.(((.....)))((((.....)))).......))).))))))..)))))). ( -28.00) >consensus __U____________CCCCUAAAGCUGACUAUUGGCCAGAUGAUUGUCGAUGACCACCUGAAUGAUCCUCAUGCAGUGCCGCUGCCUGGCCACAGUAUUGUCGAUCAGCUG ......................((((((....(((((((..((((((((.........)).)))))).....((((.....)))))))))))..(......)..)))))). (-22.79 = -21.38 + -1.41)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:41:00 2006