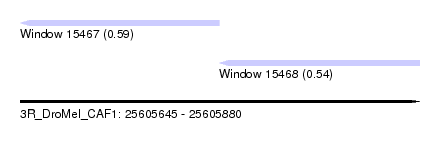
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 25,605,645 – 25,605,880 |
| Length | 235 |
| Max. P | 0.588895 |

| Location | 25,605,645 – 25,605,762 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 84.27 |
| Mean single sequence MFE | -52.08 |
| Consensus MFE | -37.66 |
| Energy contribution | -38.33 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.588895 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
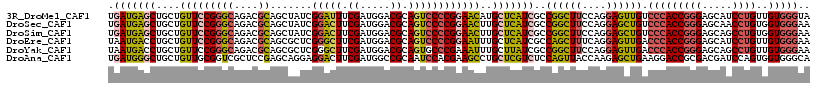
>3R_DroMel_CAF1 25605645 117 - 27905053 UGAUGAGCUGCUGUUCCGGGCAGACGCAGCUAUCGGAUUUCGAUGGACGCAGUCCCCGGAACAUGCUCAUCGCCGGCUUCCAGGAGUUGUCCCACCGGGAGCAUCCUGUUGUGGGUA .(((((((...(((((((((..(((((..((((((.....))))))..)).)))))))))))).)))))))(((.((...(((((((..((((...)))))).)))))..)).))). ( -58.70) >DroSec_CAF1 32642 117 - 1 UGAUGAGCUGCUGUUCCGGGCAGACGCAGCUAUCGGACUUCGAUGGACGCAGUCCCCGGAACUUGCUCAUCGCCGGCUUCCAGGAGCUGUCCCACCGGGAGCAACCUGUGGUGGGAA .(((((((....((((((((..(((((..((((((.....))))))..)).)))))))))))..)))))))..((((((....))))))((((((((((.....)))..))))))). ( -61.30) >DroSim_CAF1 33451 117 - 1 UGAUGAGCUGCUGUUCCGGGCAGACGCAGCUAUCGGACUUCGAUGGACGCAGUCCCCGGAACUUGCUCAUCGCCGGCUUCCAGGAGCUGUCCCACCGGGAGCAGCCUGUGGUGGGAA .(((((((....((((((((..(((((..((((((.....))))))..)).)))))))))))..)))))))..((((((....))))))((((((((((.....)))..))))))). ( -61.50) >DroEre_CAF1 33610 117 - 1 UAAUGACCUGCUGUUCCGGGCAGACGCAGCGCUCGGGCUUCGAUGGACGCAGUCCCCGGAAUUUGCUCAUCGCCAGCUUUCAGGAGUUGACCCACCGGGAGCAUCCUGUUGUGGGAA ....((...((.((((((((..(((...(((..((........))..))).)))))))))))..))...))..((((((....)))))).(((((((((.....))))..))))).. ( -42.70) >DroYak_CAF1 34744 117 - 1 UAAUGACCUGCUGUUCCGGGCAGACGCAGCGCUCGGGCUUCGAUGGACGCAGUGCCCGAAAUUUGCUUAUCGCCGGCUUCCAGGAGUUGACCCACCGGGAGCAGCCUGUUGUGGGAA ..(..((..((((((((.((.....((((...((((((..((.....))....))))))...)))).......((((((....)))))).....)).))))))))..))..)..... ( -41.10) >DroAna_CAF1 34463 117 - 1 UGAUGGGCUGCUGUUGCGGUCGCUCCGAGCAGGAGGACUUCGAUGGCCGCAAUCCACGAAGCCUGCUCGUCUCCAGUUACCAAGAGCUGAAGGACCGCGACGAUCCAGUGGUGGGCA ......((((.((((((((((....((((((((....(((((.(((.......)))))))))))))))).((.(((((......))))).))))))))))))...))))........ ( -47.20) >consensus UGAUGAGCUGCUGUUCCGGGCAGACGCAGCUAUCGGACUUCGAUGGACGCAGUCCCCGGAACUUGCUCAUCGCCGGCUUCCAGGAGCUGACCCACCGGGAGCAUCCUGUGGUGGGAA .(((((((....(((((((((....)).......(((((.((.....)).))))))))))))..)))))))..((((((....)))))).(((((((((.....))))..))))).. (-37.66 = -38.33 + 0.67)

| Location | 25,605,762 – 25,605,880 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 76.05 |
| Mean single sequence MFE | -49.40 |
| Consensus MFE | -27.06 |
| Energy contribution | -26.62 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.50 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.535086 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
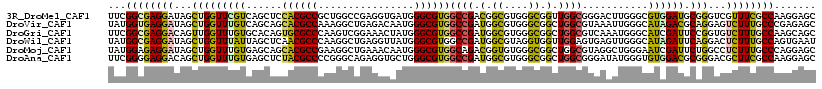
>3R_DroMel_CAF1 25605762 118 - 27905053 UUCGGCGAGGAUAGCUGGUUCGUCAGCUCCACGCCGCUGGCCGAGGUGAUGGGCGUGGCCGACGGCGUGGGCGGUUGGCGGGACUUGGGCGUGGAUGCGGGUCGUUUCGCCAAGGAGC ..((((.......))))((((....((.((((((((.((((((..((.....)).)))))).))))))))))..((((((..((...(((.((....)).)))))..)))))).)))) ( -55.70) >DroVir_CAF1 43486 118 - 1 UAUGGUGAGGAUAGCUGGUUUGUCAGCAGCACGCCAAAGGCUGAGACAAUGGGCGUGGCCGAUGGCGUGGGCGGCUGGCGUAAAUUGGGCAUAGACGCAGGAGUCUUUGCCCGAGAGC .............(((.....((((((..(((((((..(((((...(....)...)))))..)))))))....)))))).....(((((((.((((......)))).))))))).))) ( -48.80) >DroGri_CAF1 44837 118 - 1 UUCGGCGAGGACAGUUGGUUUGUGCACAGUGCGCCCAAGUCGGAAACUAUGGGCGUGGCCGAUGGCGUGGGCGGCUGGCGUCAAAUGGGCAUCGAUUCCGGUGUCUUUGCCAAGCAGC ...(((((((((.(((.((((((((.((((.((((((.(((((..((.......))..)))))....))))))))))))).))))).)))((((....)))))))))))))....... ( -55.00) >DroWil_CAF1 38319 118 - 1 UAUGGCGAGGAUAGCUGGUUUAUUAGCUCAACGCCCAAGGCUGAGGUUAUGGGCGUGGCCGAUGGCGUAGGUGGUUGGAGUGAGUUGGGCAUAGAUUCAGGACUCUUUGCCAGUGAAU ..(((((((((...(((((((((..((((((((((((..((....))..)))))....(((((.((....)).))))).....)))))))))))).))))...)))))))))...... ( -47.70) >DroMoj_CAF1 40049 118 - 1 UAUGGAGAGGAUAGCUGGUUUGUGAGCAGCACGCCGAAGGCUGAAACAAUGGGCGUGGCAGACGGUGUGGGCGGCUGGCGUAGGCUGGGAAUCGAUUCUGGCCUCUUUGCCCAGGAGC ..(((.(((((..((..(((.....((..(((((((....(((..((.......))..))).))))))).)))))..))...(((..(((.....)))..))))))))..)))..... ( -41.00) >DroAna_CAF1 34580 118 - 1 UUCGGGGAGGACAGCUGGUUUGUGAGCUCUACGCCCCGGGCAGAGGUGCUGGGCGUGGCCGAUGGCGUGGGCGGCUGGCGGGAUAUGGGUGUGGACGCGGGACGCUUCGCCAAGGAGC .((((.......((((.(....).))))((((((((.(.((....)).).))))))))))))(((((.(((((.((.(((...(((....)))..))).)).))))))))))...... ( -48.20) >consensus UACGGCGAGGAUAGCUGGUUUGUGAGCACCACGCCCAAGGCUGAGACAAUGGGCGUGGCCGAUGGCGUGGGCGGCUGGCGUGAAUUGGGCAUAGAUGCAGGACUCUUUGCCAAGGAGC ...((((((((...(((((((((......((((((................))))))((((.(.((....)).).))))...........)))))).)))...))))))))....... (-27.06 = -26.62 + -0.44)
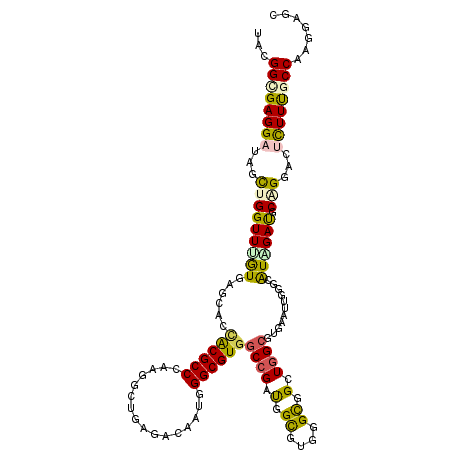
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:39:44 2006