
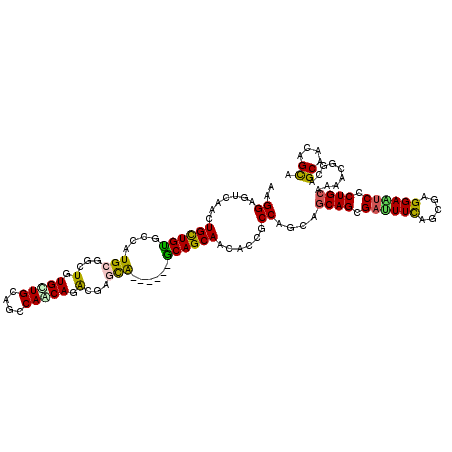
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 25,059,227 – 25,059,341 |
| Length | 114 |
| Max. P | 0.879084 |

| Location | 25,059,227 – 25,059,341 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.97 |
| Mean single sequence MFE | -46.60 |
| Consensus MFE | -24.30 |
| Energy contribution | -23.50 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.879084 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 25059227 114 + 27905053 AAGGAGUCAACUGCUGUGCCAUGAGGCUGUGUUGCACCCAGCAGACGAGCA------GCAGCAACUCGACCAGUUGCAGUGAUUUCAGCGAGGAGUCCCUGCAAAACGGAAGCAACAGCA ...((((...((((((((((....)))(((.((((.....))))))).)))------))))..))))..((..((((((.((((((.....)))))).))))))...))..((....)). ( -40.30) >DroVir_CAF1 3011 120 + 1 AUGGCGUCAACUGUUGCGCCAUGCGUCUGUGCUGUAGCCAACAGGCCAGCACCGCGGGCAGCAAUAGCGCCAGCAGCAGCGAUUUCAGCGAGGAAUCCCUGCAGAAUGGCAGCAACAGCA ..........((((((((((.((((...((((((..(((....)))))))))))))))).((....))((((...((((.((((((.....)))))).))))....)))).))))))).. ( -53.20) >DroGri_CAF1 3498 120 + 1 AUGGCGUCAAUUGUUGUGCGAUGCGUCUGUGUUGCAGCCAACAGGCAAGUGCCGCGGGCAGCAACAGUGCCAGCAGCAGUGAUUUCAGUGAGGAAUCCCUGCAAAAUGGGAGCAACAGUA ...((.((.(((((((((((((((....))))))).(((....(((....)))...))).)))))))).(((...((((.((((((.....)))))).))))....)))))))....... ( -43.80) >DroEre_CAF1 5788 114 + 1 AGGGAGUCAACUGCUGUGCCAUGAGGCUCUGCUGCAGCCAGCAGACGAGCG------GCAGCAACUCAGCCAGUUGCAGCGAUUUUAGCGAGGAAACGCUGCAAAACGGAAGCAACAGCA ...((((...((((((((((....)))(((((((....)))))))...)))------))))..))))..((..((((((((.((((......))))))))))))...))..((....)). ( -45.10) >DroAna_CAF1 3075 114 + 1 ACGGAGUCAACUGCUGUGCCAUGCGGCUCUGCUGCAACCAGCAGACGAGCA------ACAGCAACUCGGCCAGCUGCAGCGACUUCAGCGAGGAGUCCCUGCAGAACGGCAGCAACAGCA ...((((....(((((((((....)))(((((((....)))))))......------)))))))))).(((..((((((.((((((.....)))))).))))))...))).((....)). ( -49.90) >DroMoj_CAF1 3176 120 + 1 AAGGCAUCAACUGUUGCGCGAUGCGCCUGUGCUGCAGCCAACAGGCGAGCAUGGCGGGCAGCACUACCGCCAGCAACAGCGACUUCAGCGAGGAAUCCCUGCAGAAUGGCAGCAACAGCA ..(.((((..(((((((((....(((((((((....))..))))))).)).((((((.........)))))))))))))........((.(((....))))).).))).).((....)). ( -47.30) >consensus AAGGAGUCAACUGCUGUGCCAUGCGGCUGUGCUGCAGCCAACAGACGAGCA______GCAGCAACACCGCCAGCAGCAGCGAUUUCAGCGAGGAAUCCCUGCAAAACGGCAGCAACAGCA ..((.......((((((....(((...(.(((((....))))).)...)))......))))))......))....((((.((((((.....)))))).)))).........((....)). (-24.30 = -23.50 + -0.80)


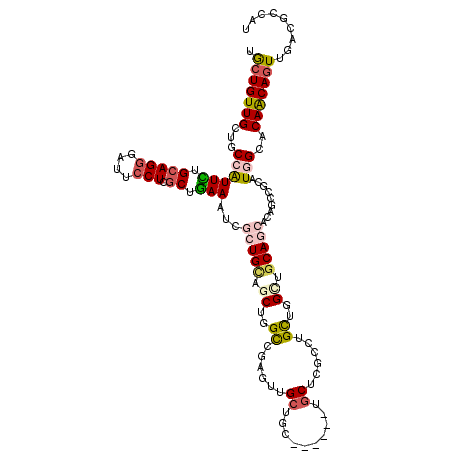
| Location | 25,059,227 – 25,059,341 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.97 |
| Mean single sequence MFE | -46.07 |
| Consensus MFE | -23.94 |
| Energy contribution | -24.50 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.576490 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 25059227 114 - 27905053 UGCUGUUGCUUCCGUUUUGCAGGGACUCCUCGCUGAAAUCACUGCAACUGGUCGAGUUGCUGC------UGCUCGUCUGCUGGGUGCAACACAGCCUCAUGGCACAGCAGUUGACUCCUU .((((((((....((((((((((....))).)).)))))....))))).))).((((((((((------((...((.(((.....)))))...(((....))).)))))).))))))... ( -36.90) >DroVir_CAF1 3011 120 - 1 UGCUGUUGCUGCCAUUCUGCAGGGAUUCCUCGCUGAAAUCGCUGCUGCUGGCGCUAUUGCUGCCCGCGGUGCUGGCCUGUUGGCUACAGCACAGACGCAUGGCGCAACAGUUGACGCCAU .((((((((.((((((((((((.((((.(.....).)))).))))(((.(((((....)).))).)))(((((((((....)))..)))))))))...)))))))))))))......... ( -53.40) >DroGri_CAF1 3498 120 - 1 UACUGUUGCUCCCAUUUUGCAGGGAUUCCUCACUGAAAUCACUGCUGCUGGCACUGUUGCUGCCCGCGGCACUUGCCUGUUGGCUGCAACACAGACGCAUCGCACAACAAUUGACGCCAU ...((((((..(((....(((((((((.(.....).))))..((((((.(((((....).)))).)))))))))))....)))..)))))).....((.(((.........))).))... ( -37.10) >DroEre_CAF1 5788 114 - 1 UGCUGUUGCUUCCGUUUUGCAGCGUUUCCUCGCUAAAAUCGCUGCAACUGGCUGAGUUGCUGC------CGCUCGUCUGCUGGCUGCAGCAGAGCCUCAUGGCACAGCAGUUGACUCCCU ((((((.(((......(((((((((((........))).))))))))..((((..(((((.((------(((......)).))).)))))..))))....)))))))))........... ( -44.70) >DroAna_CAF1 3075 114 - 1 UGCUGUUGCUGCCGUUCUGCAGGGACUCCUCGCUGAAGUCGCUGCAGCUGGCCGAGUUGCUGU------UGCUCGUCUGCUGGUUGCAGCAGAGCCGCAUGGCACAGCAGUUGACUCCGU .(((((((.((((((..(((((.((((.(.....).)))).)))))((.((((((((......------.)))))(((((((....)))))))))))))))))))))))))......... ( -53.50) >DroMoj_CAF1 3176 120 - 1 UGCUGUUGCUGCCAUUCUGCAGGGAUUCCUCGCUGAAGUCGCUGUUGCUGGCGGUAGUGCUGCCCGCCAUGCUCGCCUGUUGGCUGCAGCACAGGCGCAUCGCGCAACAGUUGAUGCCUU .(((..((((((((....((((.((((.(.....).)))).))))...))))))))(((((((..((((.((......)))))).))))))).)))(((((((......).))))))... ( -50.80) >consensus UGCUGUUGCUGCCAUUCUGCAGGGAUUCCUCGCUGAAAUCGCUGCAGCUGGCCGAGUUGCUGC______UGCUCGCCUGCUGGCUGCAGCACAGCCGCAUGGCACAACAGUUGACGCCAU .(((((((...((((((.(((((....))).)).)))...(((((.((..((......((..........))......))..)).))))).........)))..)))))))......... (-23.94 = -24.50 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:35:23 2006