
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 24,634,765 – 24,634,869 |
| Length | 104 |
| Max. P | 0.948056 |

| Location | 24,634,765 – 24,634,869 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 69.41 |
| Mean single sequence MFE | -40.88 |
| Consensus MFE | -22.65 |
| Energy contribution | -22.72 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.60 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948056 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
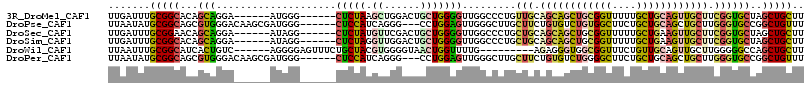
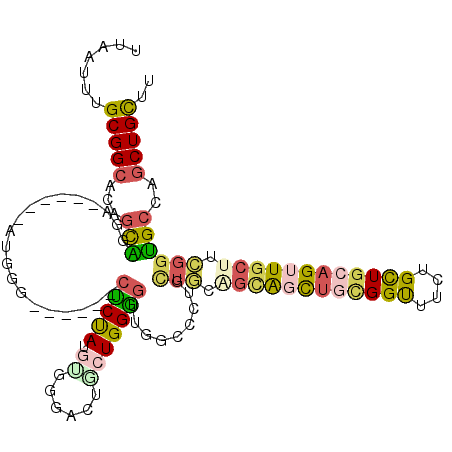
>3R_DroMel_CAF1 24634765 104 + 27905053 UUGAUUUGCGGCACAGCAGGA------AUGGG------CUCUAAGCUGGACUGCUGGGGUUGGCCCUGUUGCAGCAGCUGCGGUUUUUGCUGCAGUUGCUUCGGUGCUAGCUGCUU .......(((((.((((((..------.(.((------(.....))).).))))))....((((.(((....((((((((((((....)))))))))))).))).))))))))).. ( -48.30) >DroPse_CAF1 128508 107 + 1 UUAAUAUGCGGCAGCGUGGGACAAGCGAUGGG------CUCCAUCAGGG---CCUGGAGUUGGGCUUGCUUCUGUGUCUGUGGCUUCUGCUGCAGCUGCUUGGGUGCCGGCUGUUU .......((.((((((..((((((((..(.((------(((((......---..))))))).)))))).)))..)).)))).)).......((((((((......).))))))).. ( -40.40) >DroSec_CAF1 114884 104 + 1 UUGAUUUGCGGAACAGCAGGA------AUAGG------CUCUAUGUUCGACUGCUGGGGUUGGCCCUGCUGCAGCAGCUGCGGUUUUUGCUGAAGUUGCUUCGGUGCUAGCUGCUU .......((....((((((((------(((..------.....)))))..)))))).(((((((...((((.(((((((.((((....)))).))))))).))))))))))))).. ( -39.80) >DroSim_CAF1 117633 104 + 1 UUGAUUUGCGGCACAGCAGGA------AUAGG------CUCUAGGUUGGACUGCUGGGGUUGGCCCUGCUGCAGCAGCUGCGGUUUUUGCUGAAGUUGCUUCGGUGCUAGCUGCUU ......(((((((((((((..------.(((.------(....).)))..))))))(((....))))))))))((((((((.......(((((((...))))))))).)))))).. ( -43.21) >DroWil_CAF1 125849 101 + 1 UUAAUUUGCGGCAUCACUGUC------AGGGGAGUUUCUGCUACGUGGGGUAACUGGUUUUG---------AGAGGGUGGCGGUUUCUGUUGCAGUUGCUUGGGGGCCAGCUGCUU .......((((((((.((.((------((((.((((.((.(.....).)).))))..)))))---------).)))))...(((..(((..((....)).)))..))).))))).. ( -30.90) >DroPer_CAF1 108288 107 + 1 UUAAUAUGCGGCAGCGUGGGACAAGCGAUGGG------CUCCAUCAGGG---CCUGGAGUUGGGCUUGCUUCUGUGUCUGGGGCUUCUGCUGCAGCUGCUUGGGUGCCGGCUGUUU .......((((.(((.(.(((((((((((((.------..))))).(((---(((......)))))))))....))))).).))).)))).((((((((......).))))))).. ( -42.70) >consensus UUAAUUUGCGGCACAGCAGGA______AUGGG______CUCUAUGUGGGACUGCUGGGGUUGGCCCUGCUGCAGCAGCUGCGGUUUCUGCUGCAGUUGCUUCGGUGCCAGCUGCUU .......(((((...(((....................(((((.((......))))))).........(((.((((((((((((....)))))))))))).))))))..))))).. (-22.65 = -22.72 + 0.06)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:29:33 2006