
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 24,546,539 – 24,546,698 |
| Length | 159 |
| Max. P | 0.975449 |

| Location | 24,546,539 – 24,546,658 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 79.94 |
| Mean single sequence MFE | -37.57 |
| Consensus MFE | -22.93 |
| Energy contribution | -24.40 |
| Covariance contribution | 1.47 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.845462 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24546539 119 + 27905053 UUCCACCAUCAUCCACCGACAGCUAAUCAAAUUCGCCACCCAGCAUGCCGAUGCCCCGCAGCAGCAGAAGCAACUAAUGCUGCUGCUCCUGGAGAUCCAGUCGGAGCCGAAGCUGCAAC .................(.(((((..((......((......))...((((......(((((((((..((...))..)))))))))..((((....))))))))....))))))))... ( -35.40) >DroVir_CAF1 39656 111 + 1 UUGUGCC--------UUCACAGCUAAUCAAAUUUGCCACACAACACGCGGAGGCACCGCAACAGCAACAACAGCUGAUGCGACUGCUGCUCGAGAUUCAAUCGGAGGCCAAGCUGCAGC (((.(((--------(((.((((..........((((.(.(.....).)..)))).((((.((((.......)))).))))...))))...((.......)))))))))))........ ( -32.40) >DroPse_CAF1 53210 111 + 1 UCCGCC-------C-CCAACAGCUAAUCAAAUUUGCCACCCAGCAUGCGGAUGCUCCUCAGCAGCAGAAGCAACUGAUGCUCCUGCUGCUGGAGAUCCAGUCGGAGCCGAAGCUGAAUC ......-------.-....(((((.(((.....(((......)))....)))(((((((((((((((.((((.....)))).)))))))))).(((...))))))))...))))).... ( -41.40) >DroMoj_CAF1 57643 111 + 1 UUGUGCC--------UCCGCAGCUAAUCAAAUUUGCCACGCAGCACGCUGAUGCGCCGCAGCAGCAGCAGCAGCUGAUGCGGCUGCUGCUCGAGAUUCCAUCGGAGGCCAAGCUGCAGC (((.(((--------((((.....((((.....(((..((((.(.....).))))..)))((((((((.(((.....))).))))))))....))))....))))))))))........ ( -46.10) >DroAna_CAF1 44895 111 + 1 UCCCGCC--------CAAACAGCUAAUCAAAUUCGCCACCCAGCACGAGGAUGCGCCUCAUCAGCAGAAGCAACUGAUGUUGCUGCUCCUAGAGAUUCAGUCGGAGCCGAAGCUGAACC .......--------....(((((((((......((......))...((((.((((..((((((.........))))))..)).))))))...))))...(((....)))))))).... ( -28.70) >DroPer_CAF1 32604 112 + 1 UCCGCC-------CCCCAACAGCUAAUCAAAUUUGCCACCCAGCAUGCGGAUGCUCCUCAGCAGCAGAAGCAACUGAUGCUCCUGCUGCUGGAGAUCCAGUCGGAGCCGAAGCUGAAUC ......-------......(((((.(((.....(((......)))....)))(((((((((((((((.((((.....)))).)))))))))).(((...))))))))...))))).... ( -41.40) >consensus UCCCGCC________CCAACAGCUAAUCAAAUUUGCCACCCAGCACGCGGAUGCGCCGCAGCAGCAGAAGCAACUGAUGCUGCUGCUGCUGGAGAUCCAGUCGGAGCCGAAGCUGAAGC ...................(((((..((..................((((.....))))((((((((.((((.....)))).)))))))).))..(((....))).....))))).... (-22.93 = -24.40 + 1.47)



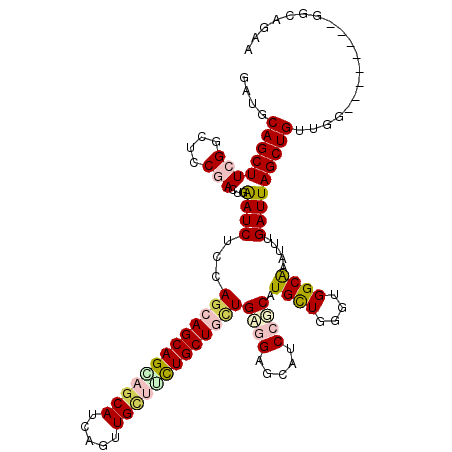
| Location | 24,546,539 – 24,546,658 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.94 |
| Mean single sequence MFE | -47.70 |
| Consensus MFE | -28.47 |
| Energy contribution | -29.42 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.906241 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24546539 119 - 27905053 GUUGCAGCUUCGGCUCCGACUGGAUCUCCAGGAGCAGCAGCAUUAGUUGCUUCUGCUGCUGCGGGGCAUCGGCAUGCUGGGUGGCGAAUUUGAUUAGCUGUCGGUGGAUGAUGGUGGAA .((.((.(.((((((((..((((....))))..(((((((((..((...))..))))))))))))))(((((((.(((((....((....)).))))))))))))...))).).)).)) ( -47.50) >DroVir_CAF1 39656 111 - 1 GCUGCAGCUUGGCCUCCGAUUGAAUCUCGAGCAGCAGUCGCAUCAGCUGUUGUUGCUGUUGCGGUGCCUCCGCGUGUUGUGUGGCAAAUUUGAUUAGCUGUGAA--------GGCACAA (((((.((((((..((.....))...)))))).)))))((((.((((.......)))).))))((((((.((((.((((.....((....))..)))))))).)--------))))).. ( -39.80) >DroPse_CAF1 53210 111 - 1 GAUUCAGCUUCGGCUCCGACUGGAUCUCCAGCAGCAGGAGCAUCAGUUGCUUCUGCUGCUGAGGAGCAUCCGCAUGCUGGGUGGCAAAUUUGAUUAGCUGUUGG-G-------GGCGGA ........((((.(((((((.(((((((((((((((((((((.....)))))))))))))..)))).))))...((((....)))).............)))))-)-------).)))) ( -53.40) >DroMoj_CAF1 57643 111 - 1 GCUGCAGCUUGGCCUCCGAUGGAAUCUCGAGCAGCAGCCGCAUCAGCUGCUGCUGCUGCUGCGGCGCAUCAGCGUGCUGCGUGGCAAAUUUGAUUAGCUGCGGA--------GGCACAA ((((((((....((......)).......(((((((((.((....)).)))))))))))))))))((....))(((((.((..((...........))..))..--------))))).. ( -54.20) >DroAna_CAF1 44895 111 - 1 GGUUCAGCUUCGGCUCCGACUGAAUCUCUAGGAGCAGCAACAUCAGUUGCUUCUGCUGAUGAGGCGCAUCCUCGUGCUGGGUGGCGAAUUUGAUUAGCUGUUUG--------GGCGGGA .....(((....)))(((.(..(((....(((((((((.......)))))))))((((((.((.(((((((.......)))).)))...)).)))))).)))..--------).))).. ( -39.10) >DroPer_CAF1 32604 112 - 1 GAUUCAGCUUCGGCUCCGACUGGAUCUCCAGCAGCAGGAGCAUCAGUUGCUUCUGCUGCUGAGGAGCAUCCGCAUGCUGGGUGGCAAAUUUGAUUAGCUGUUGGGG-------GGCGGA (((((((..(((....))))))))))..((((((((((((((.....)))))))))))))).......(((((.(.((.((..((...........))..)).)).-------)))))) ( -52.20) >consensus GAUGCAGCUUCGGCUCCGACUGAAUCUCCAGCAGCAGCAGCAUCAGUUGCUUCUGCUGCUGAGGAGCAUCCGCAUGCUGGGUGGCAAAUUUGAUUAGCUGUUGG________GGCAGAA ....((((((((....)))...((((...(((((((((((((.....)))))))))))))((((.....)))).((((....)))).....)))))))))................... (-28.47 = -29.42 + 0.95)


| Location | 24,546,578 – 24,546,698 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.00 |
| Mean single sequence MFE | -55.10 |
| Consensus MFE | -28.18 |
| Energy contribution | -30.63 |
| Covariance contribution | 2.45 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.96 |
| SVM RNA-class probability | 0.889531 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24546578 120 + 27905053 CCAGCAUGCCGAUGCCCCGCAGCAGCAGAAGCAACUAAUGCUGCUGCUCCUGGAGAUCCAGUCGGAGCCGAAGCUGCAACGUCUGCUCCGAAGUCUGGGCACCGAGCAUGAGGCAAAGCU ((..(((((((.(((((.(((((((((..((...))..)))))))))..((((....))))(((((((.((.(......).)).))))))).....))))).)).))))).))....... ( -54.10) >DroVir_CAF1 39687 120 + 1 ACAACACGCGGAGGCACCGCAACAGCAACAACAGCUGAUGCGACUGCUGCUCGAGAUUCAAUCGGAGGCCAAGCUGCAGCGUUUGCUGCGCAGCUUGGGUCCUGUGCAGGAGGCUCGUCU .......((((((....((((.((((.......)))).)))).)).)))).((((.(((...((.((((((((((((.(((.....))))))))))))..))).))...))).))))... ( -47.40) >DroPse_CAF1 53241 120 + 1 CCAGCAUGCGGAUGCUCCUCAGCAGCAGAAGCAACUGAUGCUCCUGCUGCUGGAGAUCCAGUCGGAGCCGAAGCUGAAUCGCCUGCUGCGUAGCCUGGGCCCCGAGCAGGAGGCUCGUCU .((((....((((.((((..((((((((.((((.....)))).))))))))))))))))..(((....))).))))....((((.((((...((....)).....)))).))))...... ( -56.70) >DroMoj_CAF1 57674 120 + 1 GCAGCACGCUGAUGCGCCGCAGCAGCAGCAGCAGCUGAUGCGGCUGCUGCUCGAGAUUCCAUCGGAGGCCAAGCUGCAGCGUGUGCUGCGCAGCCUGGGCCCCGCGCAGGAAGCGCGCCU ...((.((((..(((((((.(((((((((.(((.....))).)))))))))))..........((.((((..(((((.(((.....))))))))...)))))))))))...)))).)).. ( -65.40) >DroAna_CAF1 44926 120 + 1 CCAGCACGAGGAUGCGCCUCAUCAGCAGAAGCAACUGAUGUUGCUGCUCCUAGAGAUUCAGUCGGAGCCGAAGCUGAACCGGCUGCUCCGAAGCCUAGGGGCGGUACAAGAGGCUAAGAU ...(((......)))((((((((((.........)))))..((((((((((((.(......(((((((...(((((...))))))))))))..)))))))))))))...)))))...... ( -50.30) >DroPer_CAF1 32636 120 + 1 CCAGCAUGCGGAUGCUCCUCAGCAGCAGAAGCAACUGAUGCUCCUGCUGCUGGAGAUCCAGUCGGAGCCGAAGCUGAAUCGCCUGCUGCGUAGCCUGGGCCCCGAGCAGGAGGCUCGUCU .((((....((((.((((..((((((((.((((.....)))).))))))))))))))))..(((....))).))))....((((.((((...((....)).....)))).))))...... ( -56.70) >consensus CCAGCACGCGGAUGCGCCGCAGCAGCAGAAGCAACUGAUGCUGCUGCUGCUGGAGAUCCAGUCGGAGCCGAAGCUGAAGCGUCUGCUGCGAAGCCUGGGCCCCGAGCAGGAGGCUCGUCU .((((..((((.....))))((((((((.((((.....)))).)))))))).....(((....)))......))))....((((.((((...((....)).....)))).))))...... (-28.18 = -30.63 + 2.45)


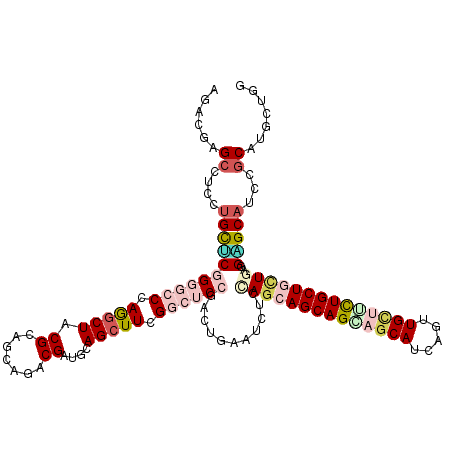
| Location | 24,546,578 – 24,546,698 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.00 |
| Mean single sequence MFE | -58.60 |
| Consensus MFE | -33.46 |
| Energy contribution | -35.72 |
| Covariance contribution | 2.26 |
| Combinations/Pair | 1.32 |
| Mean z-score | -3.34 |
| Structure conservation index | 0.57 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975449 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24546578 120 - 27905053 AGCUUUGCCUCAUGCUCGGUGCCCAGACUUCGGAGCAGACGUUGCAGCUUCGGCUCCGACUGGAUCUCCAGGAGCAGCAGCAUUAGUUGCUUCUGCUGCUGCGGGGCAUCGGCAUGCUGG .......((.(((((.((((((((.....(((((((.((.((....)).)).)))))))((((....))))..(((((((((..((...))..))))))))).)))))))))))))..)) ( -62.10) >DroVir_CAF1 39687 120 - 1 AGACGAGCCUCCUGCACAGGACCCAAGCUGCGCAGCAAACGCUGCAGCUUGGCCUCCGAUUGAAUCUCGAGCAGCAGUCGCAUCAGCUGUUGUUGCUGUUGCGGUGCCUCCGCGUGUUGU .((((.((.....((((.(((.(((((((((..(((....))))))))))))..))).............((((((((.(((........))).)))))))).))))....)).)))).. ( -47.00) >DroPse_CAF1 53241 120 - 1 AGACGAGCCUCCUGCUCGGGGCCCAGGCUACGCAGCAGGCGAUUCAGCUUCGGCUCCGACUGGAUCUCCAGCAGCAGGAGCAUCAGUUGCUUCUGCUGCUGAGGAGCAUCCGCAUGCUGG ...(.(((....(((((((((((.(((((.(((.....)))....))))).))))))))..(((((((((((((((((((((.....)))))))))))))..)))).))))))).))).) ( -63.40) >DroMoj_CAF1 57674 120 - 1 AGGCGCGCUUCCUGCGCGGGGCCCAGGCUGCGCAGCACACGCUGCAGCUUGGCCUCCGAUGGAAUCUCGAGCAGCAGCCGCAUCAGCUGCUGCUGCUGCUGCGGCGCAUCAGCGUGCUGC .((((((((...(((((((((.(((((((((..(((....)))))))))))).))))..........(((((((((((.(((.....))).))))))))).)))))))..)))))))).. ( -71.10) >DroAna_CAF1 44926 120 - 1 AUCUUAGCCUCUUGUACCGCCCCUAGGCUUCGGAGCAGCCGGUUCAGCUUCGGCUCCGACUGAAUCUCUAGGAGCAGCAACAUCAGUUGCUUCUGCUGAUGAGGCGCAUCCUCGUGCUGG ......(((((..(((..((.((((((.(((((.(.((((((.......)))))).)..))))).).))))).))((((((....))))))..)))....)))))((((....))))... ( -44.60) >DroPer_CAF1 32636 120 - 1 AGACGAGCCUCCUGCUCGGGGCCCAGGCUACGCAGCAGGCGAUUCAGCUUCGGCUCCGACUGGAUCUCCAGCAGCAGGAGCAUCAGUUGCUUCUGCUGCUGAGGAGCAUCCGCAUGCUGG ...(.(((....(((((((((((.(((((.(((.....)))....))))).))))))))..(((((((((((((((((((((.....)))))))))))))..)))).))))))).))).) ( -63.40) >consensus AGACGAGCCUCCUGCUCGGGGCCCAGGCUACGCAGCAGACGAUGCAGCUUCGGCUCCGACUGAAUCUCCAGCAGCAGCAGCAUCAGUUGCUUCUGCUGCUGAGGAGCAUCCGCAUGCUGG ......((....(((((((((((.(((((.((.......))....))))).))))))...........((((((((((((((.....))))))))))))))..)))))...))....... (-33.46 = -35.72 + 2.26)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:28:58 2006