
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 24,392,515 – 24,392,627 |
| Length | 112 |
| Max. P | 0.583434 |

| Location | 24,392,515 – 24,392,607 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 92 |
| Reading direction | reverse |
| Mean pairwise identity | 81.43 |
| Mean single sequence MFE | -28.02 |
| Consensus MFE | -17.27 |
| Energy contribution | -17.02 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.548716 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
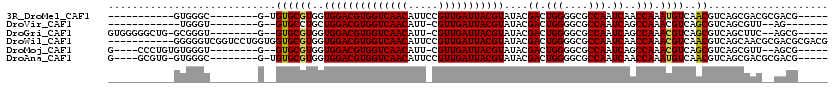
>3R_DroMel_CAF1 24392515 92 - 27905053 CGUGGUCAACAUUCCGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAAUGUCAACGUCAGCGACGCGACGUUUGUCAUGUUGACUGU .(..((((((((...(((((((.(((...(.....).)))..)))))))((((((((..((((...)))).))))))))..))))))))..) ( -30.30) >DroVir_CAF1 206248 74 - 1 CGUGGUCAACAUU-CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAGCCAAACGUCAGCGUCAGCGUU--AG---------------GCGA ((((((((((...-.)))))))))).....((.(((....))).)).(((.(((((........)))))--.)---------------)).. ( -25.60) >DroGri_CAF1 201645 86 - 1 CGUGGUCAACAUU-CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAGCCAAACGUCAGCGUCAGCUUC--AGCGUUAGUU---UUGGGUGA ((((((((((...-.))))))))))...(((.(((((((((......))...(((....)))..)))))--))))).....---........ ( -25.80) >DroYak_CAF1 222947 92 - 1 CGUGGUCAACAUUCCGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAAUGUCAACGUCAGCGACGCGACGUUUGUCAUGUUGACUGU .(..((((((((...(((((((.(((...(.....).)))..)))))))((((((((..((((...)))).))))))))..))))))))..) ( -30.30) >DroMoj_CAF1 181678 76 - 1 CGUGGUCAACAUU-CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAGCCAAACGUCAGCGUCAGCGUU--AGCG-------------GCCA ((((((((((...-.)))))))))).........(((((........)))...((..((((....))))--..))-------------.)). ( -25.80) >DroAna_CAF1 218176 92 - 1 CGUGGUCAACAUUCCGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAAUGUCAACGUCAGCGACGCGACGUUUGUCAUGUUGACUGU .(..((((((((...(((((((.(((...(.....).)))..)))))))((((((((..((((...)))).))))))))..))))))))..) ( -30.30) >consensus CGUGGUCAACAUU_CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAACGUCAACGUCAGCGAC__AACGUUUGUC___UUGACUGA ((((((((((.....)))))))))).....(((...((((.............))))...)))............................. (-17.27 = -17.02 + -0.25)



| Location | 24,392,532 – 24,392,627 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.70 |
| Mean single sequence MFE | -39.18 |
| Consensus MFE | -24.25 |
| Energy contribution | -24.00 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.583434 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24392532 95 - 27905053 -----------GUGGGC--------G-UGUGCGUGGUGGACGUGGUCAACAUUCCGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAAUGUCAACGUCAGCGACGCGACG----- -----------...(((--------(-(.(..((.(((.(((((((((((.....))))))))))).))).))..).)))))............(((..((((...)))).))).----- ( -34.90) >DroVir_CAF1 206252 88 - 1 ------------UGGGU--------G--GUGCCUGCUGGACGUGGUCAACAUU-CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAGCCAAACGUCAGCGUCAGCGUU--AG------- ------------((..(--------(--(((((((.((.(((((((((((...-.)))))))))))...)).)..))))))))..))((...(((....)))..))...--..------- ( -33.10) >DroGri_CAF1 201659 101 - 1 GUGGGGGCUG-GCGGGU--------G--GUGCGUGGUGGACGUGGUCAACAUU-CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAGCCAAACGUCAGCGUCAGCUUC--AGCG----- ...((.((((-(((..(--------(--((..((.(((.(((((((((((...-.))))))))))).))).))(((....)))....))))..))))))).)).((...--.)).----- ( -45.50) >DroWil_CAF1 197342 109 - 1 -----------GGGGGUCGGUCCUGGUGGUGCGUGGUGGACGUGGUCAACAUUCCGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAACGUCAACGUCAGCAACGCGACGCGACG -----------...(((.(((..(((((.(.(((.(((.(((((((((((.....))))))))))).))).)).).).)))))))).)))...((((..((((.((...)))))).)))) ( -41.50) >DroMoj_CAF1 181682 98 - 1 G----CCCUGUGUGGGU--------G--GUGCGUGGUGGACGUGGUCAACAUU-CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAGCCAAACGUCAGCGUCAGCGUU--AGCG----- (----(((.....))))--------(--((((((.(((.(((((((((((...-.))))))))))).))).))....)))))...........((..((((....))))--..))----- ( -38.30) >DroAna_CAF1 218193 101 - 1 G----GCGUG-GUGGGC--------G-UGUGCGUGGUGGACGUGGUCAACAUUCCGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAAUGUCAACGUCAGCGACGCGACG----- .----((((.-((.(((--------(-(..((((..((((((((((((((.....)))))))))))....((.(((....))).))..))).))))..))))).)).))))....----- ( -41.80) >consensus ___________GUGGGU________G__GUGCGUGGUGGACGUGGUCAACAUU_CGUUGAUUACGUAUACGACUGGGGCGCCAAUCAACCAAACGUCAACGUCAGCGAC__AACG_____ ............................((((((..((((((((((((((.....)))))))))))....((.(((....))).))..))).))))..)).................... (-24.25 = -24.00 + -0.25)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:27:31 2006