
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
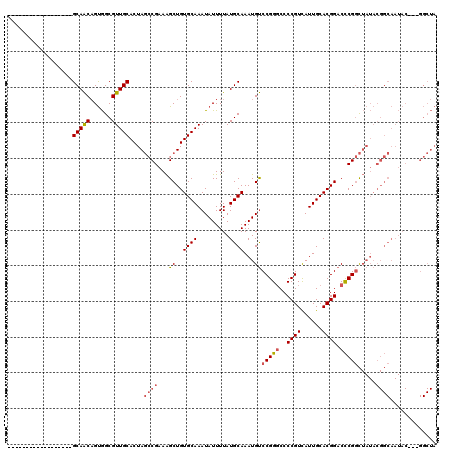
| Location | 24,390,465 – 24,390,632 |
| Length | 167 |
| Max. P | 0.710467 |

| Location | 24,390,465 – 24,390,564 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.65 |
| Mean single sequence MFE | -33.87 |
| Consensus MFE | -25.98 |
| Energy contribution | -26.57 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.710467 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24390465 99 + 27905053 ------------------GCAACAGUGGCGUUGCACUAGCCGAAAGCUGUGCAAAUAUUUUAUGCAAAUGUCCGGGCCCCGUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUA ------------------(((((......)))))..((((((...((((((((.........)))..(((.(((((..((((......)))).))))).))))))))....)---))))) ( -36.50) >DroGri_CAF1 199219 80 + 1 ------------------GCAGCCGCAGCGUUGCACUAUACGAAGGCUGUGCAAAUAUUUUAUGCAAAUGCCCGG-CCCCGUUAUUGCACGGACUCGCU--------------------- ------------------((.((((...(((........)))..(((..((((.........))))...))))))-).((((......))))....)).--------------------- ( -21.30) >DroSec_CAF1 212640 99 + 1 ------------------GCAACAGUGGCGUUGCACUAGCCGAAAGCUGUGCAAAUAUUUUAUGCAAAUGUCCGGGCCCCGUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUA ------------------(((((......)))))..((((((...((((((((.........)))..(((.(((((..((((......)))).))))).))))))))....)---))))) ( -36.50) >DroSim_CAF1 221284 99 + 1 ------------------GCAACAGUGGCGUUGCACUAGCCGAAAGCUGUGCAAAUAUUUUAUGCAAAUGUCCGGGCCCCGUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUA ------------------(((((......)))))..((((((...((((((((.........)))..(((.(((((..((((......)))).))))).))))))))....)---))))) ( -36.50) >DroEre_CAF1 227130 99 + 1 ------------------GCAACAGUGGCGUUGCACUAGCCGAAAGCUGUGCAAAUAUUUUAUGCAAAUGUCCGGGCCCCGUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUA ------------------(((((......)))))..((((((...((((((((.........)))..(((.(((((..((((......)))).))))).))))))))....)---))))) ( -36.50) >DroYak_CAF1 220871 120 + 1 GCAAAAGCAACAGAAACAGCAACAGUGGCGUUGCACUAGCCGAAAGCUGUGCAAAUAUUUUAUGCAAAUGUCCGGGCCCCGUCAUUGCACGGACCCGGCUAUACGGCAAUACUAUGGCUA ((....)).........(((...((((...((((((.((.(....)))))))))........(((..(((.(((((..((((......)))).))))).)))...))).))))...))). ( -35.90) >consensus __________________GCAACAGUGGCGUUGCACUAGCCGAAAGCUGUGCAAAUAUUUUAUGCAAAUGUCCGGGCCCCGUCAUUGCACGGACCCGGCUAUACGGCAAUAC___GGCUA ..................(((((......)))))....((((...((..((((.........))))...))(((((..((((......)))).))))).....))))............. (-25.98 = -26.57 + 0.59)


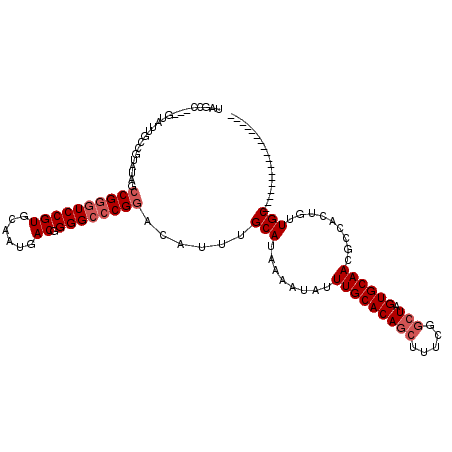
| Location | 24,390,465 – 24,390,564 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.65 |
| Mean single sequence MFE | -34.55 |
| Consensus MFE | -24.71 |
| Energy contribution | -25.55 |
| Covariance contribution | 0.83 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.66 |
| Structure conservation index | 0.72 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.514350 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24390465 99 - 27905053 UAGCC---GUAUUGCCGUAUAGCCGGGUCCGUGCAAUGACGGGGCCCGGACAUUUGCAUAAAAUAUUUGCACAGCUUUCGGCUAGUGCAACGCCACUGUUGC------------------ (((((---(....((.((...(((((((((((......)).)))))))).)....(((.........))))).))...))))))..((((((....))))))------------------ ( -36.30) >DroGri_CAF1 199219 80 - 1 ---------------------AGCGAGUCCGUGCAAUAACGGGG-CCGGGCAUUUGCAUAAAAUAUUUGCACAGCCUUCGUAUAGUGCAACGCUGCGGCUGC------------------ ---------------------.((....((((......))))((-((((((...((((.........))))..)))......(((((...))))))))))))------------------ ( -23.50) >DroSec_CAF1 212640 99 - 1 UAGCC---GUAUUGCCGUAUAGCCGGGUCCGUGCAAUGACGGGGCCCGGACAUUUGCAUAAAAUAUUUGCACAGCUUUCGGCUAGUGCAACGCCACUGUUGC------------------ (((((---(....((.((...(((((((((((......)).)))))))).)....(((.........))))).))...))))))..((((((....))))))------------------ ( -36.30) >DroSim_CAF1 221284 99 - 1 UAGCC---GUAUUGCCGUAUAGCCGGGUCCGUGCAAUGACGGGGCCCGGACAUUUGCAUAAAAUAUUUGCACAGCUUUCGGCUAGUGCAACGCCACUGUUGC------------------ (((((---(....((.((...(((((((((((......)).)))))))).)....(((.........))))).))...))))))..((((((....))))))------------------ ( -36.30) >DroEre_CAF1 227130 99 - 1 UAGCC---GUAUUGCCGUAUAGCCGGGUCCGUGCAAUGACGGGGCCCGGACAUUUGCAUAAAAUAUUUGCACAGCUUUCGGCUAGUGCAACGCCACUGUUGC------------------ (((((---(....((.((...(((((((((((......)).)))))))).)....(((.........))))).))...))))))..((((((....))))))------------------ ( -36.30) >DroYak_CAF1 220871 120 - 1 UAGCCAUAGUAUUGCCGUAUAGCCGGGUCCGUGCAAUGACGGGGCCCGGACAUUUGCAUAAAAUAUUUGCACAGCUUUCGGCUAGUGCAACGCCACUGUUGCUGUUUCUGUUGCUUUUGC ((((.(((((...((.(((...((((((((((......)).)))))))).....))).........(((((((((.....))).)))))).)).))))).))))................ ( -38.60) >consensus UAGCC___GUAUUGCCGUAUAGCCGGGUCCGUGCAAUGACGGGGCCCGGACAUUUGCAUAAAAUAUUUGCACAGCUUUCGGCUAGUGCAACGCCACUGUUGC__________________ ......................((((((((((......)).))))))))......(((........(((((((((.....))).)))))).........))).................. (-24.71 = -25.55 + 0.83)

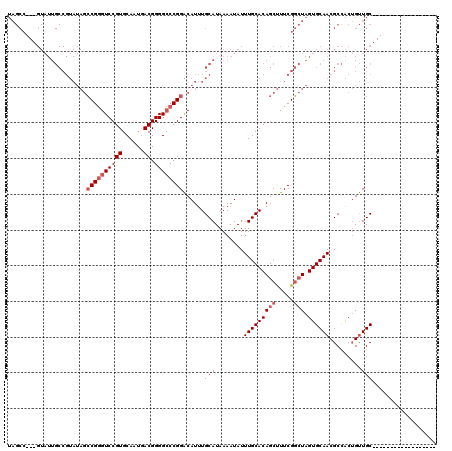
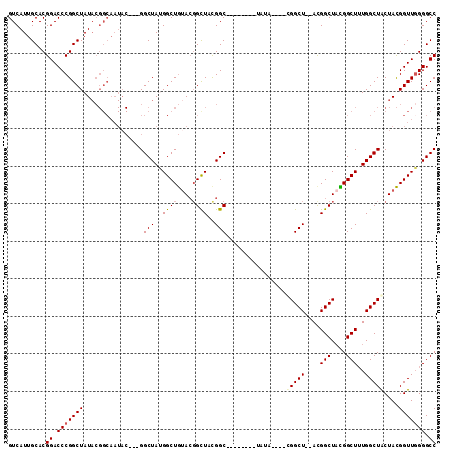
| Location | 24,390,527 – 24,390,632 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 82.08 |
| Mean single sequence MFE | -42.15 |
| Consensus MFE | -23.31 |
| Energy contribution | -23.44 |
| Covariance contribution | 0.13 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.25 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.542832 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24390527 105 + 27905053 GUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUAUGGCUGUACGGUUCCGGCUAUACGGCUAUA----UGGCU--AUGGCUAUGGCUUUGGCUACUACGGUUGGGGCC (((((.((.((((.((((((....)))..(((---(((....))))))))).))))(((....)))....----..)).--)))))...(((((..(((.....)))..))))) ( -41.20) >DroSec_CAF1 212702 97 + 1 GUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUACGGCUGUACGGCUCCGGC--------UAUA----UGGCU--ACGGCUACGGCUUUGGCUACUACGGUUCGGGCC ......((.((((((.((((....(((..(((---(((....))))))..)))..)))--------)...----(((((--(.(((....))).))))))....)))))).)). ( -40.90) >DroSim_CAF1 221346 97 + 1 GUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUACGGCUGUACGGCUCCGGC--------UAUA----UGGCU--ACGGCUACGGCUUUGGCUACUACGGUUGGGGCC (((...((.((....)))).....)))..(((---(((....)))))).(((((((((--------..((----(((((--(.(((....))).)))))).))..))))))))) ( -45.00) >DroEre_CAF1 227192 103 + 1 GUCAUUGCACGGACCCGGCUAUACGGCAAUAC---GGCUAUGGCUGUACGGCUACGGCUA--CGGCU--A----CGGCUAUAUGGCUACGGCUUUGGCUACUACGGUUGGGGCC ((.((.((.((....)))).))))(((.(((.---.((..(((((((..(((....))))--)))))--)----..))..))).)))..(((((..(((.....)))..))))) ( -40.90) >DroYak_CAF1 220951 108 + 1 GUCAUUGCACGGACCCGGCUAUACGGCAAUACUAUGGCUAUGGCUGCACGGCUAUGGCUA--CGGCUAUA----CGGCUAUAUGGCUACGGCUUUGGCUACUACGGUUGGGGCC (((........)))..(((((((..((.((((..(((((((((((....)))))))))))--..).))).----..))..)))))))..(((((..(((.....)))..))))) ( -43.10) >DroAna_CAF1 215716 89 + 1 GUCAUUGCACGGACCCGGCUAU-------------GGCUAUGGCUAUA--GUUACGGC--------AAAAGCAUCGGCU--ACGGCUUUGGCUAUGGCUGCUAUGGUUGGGGCC ..........((.(((((((((-------------(((...(((((((--((((.(((--------...(((....)))--...))).))))))))))))))))))))))).)) ( -41.80) >consensus GUCAUUGCACGGACCCGGCUAUACGGCAAUAC___GGCUAUGGCUGUACGGCUACGGC________UAUA____CGGCU__ACGGCUACGGCUUUGGCUACUACGGUUGGGGCC ..........((.(((((((....(......)...(((((.((((((..((((..(((..................)))....)))))))))).))))).....))))))).)) (-23.31 = -23.44 + 0.13)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:27:29 2006