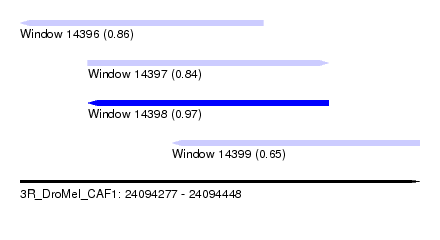
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 24,094,277 – 24,094,448 |
| Length | 171 |
| Max. P | 0.971634 |

| Location | 24,094,277 – 24,094,381 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 81.41 |
| Mean single sequence MFE | -26.33 |
| Consensus MFE | -17.12 |
| Energy contribution | -17.24 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.47 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.84 |
| SVM RNA-class probability | 0.864658 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24094277 104 - 27905053 CAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUUUUGGCUGUGUCGGCAGCAAAACAAUUAUUCGUUCCAUAC-------UUC-----A--UCGCCGAGCUGCGCUUUUCAUUCCU ..((((((((((((...)))))))))))).........((..(((..((((((..(((........)))......-------.((-----.--.....))))))).)....))).)). ( -26.00) >DroPse_CAF1 300830 102 - 1 CAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGACUGUGUGGGCAAUAAAACAAUUAUUCA-GCUACAC-------UUC-----C--ACACAAAGCUGCGCUUUCGACUCUU ..((((((((((((...))))))))))))......(-(((.((((((((((((((.....)))))..-)).....-------..)-----)--)))))((((...))))))))..... ( -27.01) >DroSim_CAF1 281357 103 - 1 CAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGGCUGUGUCGGCAGCAAAACAAUUAUUCGUGCCAUAC-------UUC-----A--UCGUCGAGCUGCGCUUUUCAUUCCU ..(((((.....))))).......((((..((((.(-(((((((....)))))....))).))))..))))....-------...-----.--..(..((((...))))..)...... ( -27.50) >DroYak_CAF1 293876 108 - 1 CAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGGCUGUGUCGGCUACAAAACAAUUAUUCCUGCCAUACUUCAUACUUC-----A--UCGCC--GCUGCGCUUUUCACUCCU ..((((((((((((...)))))))))))).......-.(((.((((.(((..................)))))))..........-----.--..((.--...))))).......... ( -25.17) >DroAna_CAF1 290657 109 - 1 CAGUCCUUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UAGCUGUACGGGCAAUAAAACAAUUAUUCAUAGCAUAC-------UUUUCUGCACUGUGCUGAGCUGAGCU-UUGACUCUU .(((((((((((((...)))))))))........((-((((((((((((.(((((.....))))))...(((...-------.....))).))))))..)))))))..-..))))... ( -25.30) >DroPer_CAF1 302666 102 - 1 CAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGACUGUGUGGGCAAUAAAACAAUUAUUCA-GCUACAC-------UUC-----C--ACACAAAGCUGCGCUUUCGACUCUU ..((((((((((((...))))))))))))......(-(((.((((((((((((((.....)))))..-)).....-------..)-----)--)))))((((...))))))))..... ( -27.01) >consensus CAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU_UGGCUGUGUCGGCAACAAAACAAUUAUUCAUGCCAUAC_______UUC_____A__UCGCCGAGCUGCGCUUUUCACUCCU ((((((((((((((...))))))))))...............((((.(((..................))))))).........................)))).............. (-17.12 = -17.24 + 0.11)



| Location | 24,094,306 – 24,094,409 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 79.87 |
| Mean single sequence MFE | -32.38 |
| Consensus MFE | -16.32 |
| Energy contribution | -16.38 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.842715 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24094306 103 + 27905053 ---GUAUGGAACGAAUAAUUGUUUUGCUGCCGACACAGCCAAAAUAUUUAUGCCUAAUUGGGUGCCAAUUAGGCACUGCCGGCCACCACCUU--GU--GUAGAUC--------CCUGC ---(((.(((....((((.((((((((((......))).))))))).))))((((((((((...)))))))))).(((((((........))--).--)))).))--------).))) ( -30.60) >DroPse_CAF1 300859 111 + 1 ---GUGUAGC-UGAAUAAUUGUUUUAUUGCCCACACAGUCA-AAUAUUUAUGCCUAAUUGGGUGCCAAUUAGGCACUGCCGGUCUACACUUUCUGU--GUAGAUUUGGUAGGACUUUC ---((((.((-..(((((.....)))))))..))))((((.-........(((((((((((...)))))))))))((((((((((((((.....))--))))).)))))))))))... ( -43.20) >DroEre_CAF1 295038 102 + 1 ---GUAUGGCACGAAUAAUUGUUUUGUUGCCGACACAGCCA-AAUAUUUAUGCCUAAUUGGGUGCCAAUUAGGCACUGCCGGCCACCACUUU--GU--GUAGAUC--------CCGGC ---((.(((((((((.......)))).))))))).......-........(((((((((((...)))))))))))..(((((...(.((...--..--)).)...--------))))) ( -31.80) >DroYak_CAF1 293907 105 + 1 GAAGUAUGGCAGGAAUAAUUGUUUUGUAGCCGACACAGCCA-AAUAUUUAUGCCUAAUUGGGUGCCAAUUAGGCACUGCUGGCCACCACUUU--GU--GUAGAUC--------CCUGC (((((.((((.((.((((.....))))..))....((((..-........(((((((((((...)))))))))))..))))))))..)))))--..--((((...--------.)))) ( -30.30) >DroAna_CAF1 290692 96 + 1 ---GUAUGCUAUGAAUAAUUGUUUUAUUGCCCGUACAGCUA-AAUAUUUAUGCCUAAUUGGGUGCCAAUUAAGGACUGCCGGUCACCAUUUU--CUCAGUGG---------------- ---((((((.(((((.......))))).))..)))).....-.........((((((((((...))))))).((....)))))..(((((..--...)))))---------------- ( -15.20) >DroPer_CAF1 302695 111 + 1 ---GUGUAGC-UGAAUAAUUGUUUUAUUGCCCACACAGUCA-AAUAUUUAUGCCUAAUUGGGUGCCAAUUAGGCACUGCCGGUCUACACUUUCUGU--GUAGAUUUGGUAGGACUUUC ---((((.((-..(((((.....)))))))..))))((((.-........(((((((((((...)))))))))))((((((((((((((.....))--))))).)))))))))))... ( -43.20) >consensus ___GUAUGGCAUGAAUAAUUGUUUUAUUGCCCACACAGCCA_AAUAUUUAUGCCUAAUUGGGUGCCAAUUAGGCACUGCCGGCCACCACUUU__GU__GUAGAUC________CCUGC ..............((((.((((..((((......))))...)))).))))((((((((((...)))))))))).((((...................))))................ (-16.32 = -16.38 + 0.06)


| Location | 24,094,306 – 24,094,409 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 79.87 |
| Mean single sequence MFE | -32.85 |
| Consensus MFE | -16.94 |
| Energy contribution | -17.00 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.32 |
| Structure conservation index | 0.52 |
| SVM decision value | 1.68 |
| SVM RNA-class probability | 0.971634 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24094306 103 - 27905053 GCAGG--------GAUCUAC--AC--AAGGUGGUGGCCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUUUUGGCUGUGUCGGCAGCAAAACAAUUAUUCGUUCCAUAC--- ...((--------(((....--..--...((.((.(((((((((((((((((((...))))))))))))..............))))))).))...))........)))))....--- ( -33.76) >DroPse_CAF1 300859 111 - 1 GAAAGUCCUACCAAAUCUAC--ACAGAAAGUGUAGACCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGACUGUGUGGGCAAUAAAACAAUUAUUCA-GCUACAC--- ...((((...((...(((((--((.....)))))))..))..((((((((((((...)))))))))))).......-.))))((((((..(((((.....)))))..-.))))))--- ( -35.00) >DroEre_CAF1 295038 102 - 1 GCCGG--------GAUCUAC--AC--AAAGUGGUGGCCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGGCUGUGUCGGCAACAAAACAAUUAUUCGUGCCAUAC--- (((((--------...((((--..--...))))...))))).((((((((((((...)))))))))))).......-(((((((...(....)...))).........))))...--- ( -36.50) >DroYak_CAF1 293907 105 - 1 GCAGG--------GAUCUAC--AC--AAAGUGGUGGCCAGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGGCUGUGUCGGCUACAAAACAAUUAUUCCUGCCAUACUUC (((((--------(((...(--((--...)))((((((.(((((((((((((((...)))))))))).(((....)-)))))))...)))))).........))))))))........ ( -39.40) >DroAna_CAF1 290692 96 - 1 ----------------CCACUGAG--AAAAUGGUGACCGGCAGUCCUUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UAGCUGUACGGGCAAUAAAACAAUUAUUCAUAGCAUAC--- ----------------.((((...--.....)))).((((((((.(((((((((...)))))))))..........-..))))).)))...........................--- ( -17.42) >DroPer_CAF1 302695 111 - 1 GAAAGUCCUACCAAAUCUAC--ACAGAAAGUGUAGACCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UGACUGUGUGGGCAAUAAAACAAUUAUUCA-GCUACAC--- ...((((...((...(((((--((.....)))))))..))..((((((((((((...)))))))))))).......-.))))((((((..(((((.....)))))..-.))))))--- ( -35.00) >consensus GCAGG________GAUCUAC__AC__AAAGUGGUGACCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU_UGGCUGUGUCGGCAACAAAACAAUUAUUCAUGCCAUAC___ ..................................(.((.(((((((((((((((...))))))))))))..............))).)))............................ (-16.94 = -17.00 + 0.06)



| Location | 24,094,342 – 24,094,448 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 86.36 |
| Mean single sequence MFE | -32.45 |
| Consensus MFE | -23.38 |
| Energy contribution | -23.68 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.07 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.650518 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 24094342 106 - 27905053 UAAACAAAACAGCAU-GGGUGUAAUGGAAACGAAGGGUAUGCAGG--------GAUCUACAC--AAGGUGGUGGCCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUUUUG ...........((.(-((((((..((....))..((((((((..(--------(..((((..--...))))...)).))).))))).....))))))).....))............ ( -30.30) >DroPse_CAF1 300894 106 - 1 UAAGUAAAAUGGAC----------CCGAAAGGAAAAGUAGGAAAGUCCUACCAAAUCUACACAGAAAGUGUAGACCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UG ((((((...(((..----------((....))....(((((.....)))))....(((((((.....))))))))))...((((((((((((...))))))))))))...))))-)) ( -38.50) >DroSec_CAF1 281712 105 - 1 UAAACAAAACGGAAU-GGGUGUAAUGGAAACGAAGGGUAUGCAGG--------GAUCUACAC--AAAGUGGUGGCCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UG ..............(-((((((..((....))..((((((((..(--------(..((((..--...))))...)).))).))))).....)))))))................-.. ( -29.30) >DroSim_CAF1 281422 105 - 1 UAAACAAAACGGCAU-GGGUGUAAUGGAAACGAAGGGUAUGCAGG--------GAUCUACAC--AAAGUGGUGGCCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UG ....((((.((((.(-((((.(........(....)........)--------.)))))(((--......)))))))...((((((((((((...)))))))))))).....))-)) ( -30.89) >DroEre_CAF1 295074 105 - 1 UUAACAAAACGGCAU-GGGUGUAAUGGAAACGAAGGGUAUGCCGG--------GAUCUACAC--AAAGUGGUGGCCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UG ...........((.(-((((((..((....))..(((((((((((--------...((((..--...))))...)))))).))))).....))))))).....)).........-.. ( -35.90) >DroYak_CAF1 293946 106 - 1 UAAACAAAACGGCAUUGGGUGUAAUGGGAACGAAGGGUAUGCAGG--------GAUCUACAC--AAAGUGGUGGCCAGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU-UG ...........(((((((((((..((....))..((((((((..(--------(..((((..--...))))...)).))).))))).....)))))))))...)).........-.. ( -29.80) >consensus UAAACAAAACGGCAU_GGGUGUAAUGGAAACGAAGGGUAUGCAGG________GAUCUACAC__AAAGUGGUGGCCGGCAGUGCCUAAUUGGCACCCAAUUAGGCAUAAAUAUU_UG .........((((...........((....))........................((((.......))))..))))...((((((((((((...)))))))))))).......... (-23.38 = -23.68 + 0.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:24:10 2006