
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 3,018,439 – 3,018,597 |
| Length | 158 |
| Max. P | 0.999264 |

| Location | 3,018,439 – 3,018,538 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 82.73 |
| Mean single sequence MFE | -34.12 |
| Consensus MFE | -27.44 |
| Energy contribution | -27.88 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.38 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.556459 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3018439 99 + 27905053 --------------------UAGUCCAAGGUGUCGUCGCCCACUAGCAGCGGUGCUGCUGGCGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCGCCGCCAGCCGCGCAGGCUGGC --------------------........((((.((..((......)).((((((((((....)).))))))))....................)).))))(((((((.....))))))) ( -39.50) >DroVir_CAF1 7354 119 + 1 CCCAAUCCCACGCCCAACAUCAGUCCAAGGUAUCGUCGCCCACCAGUAGCGGUGCUGCUGGCGCUAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCUCACAGGCUGGU ...........((.......((((....((((((((..(......)..))))))))))))(((....))).))...........................(((((((.....))))))) ( -32.00) >DroGri_CAF1 6468 102 + 1 -----------------ACCCAGUCCAAGUUAUCGUCGCCCACCAGUAGCGGUGCUGCUGGCGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCACCACCAGCCUCGCAGGCUGGU -----------------...........((((..(((((.(.((((((((...)))))))).)...)))..))..)))).....................(((((((.....))))))) ( -30.00) >DroWil_CAF1 7409 103 + 1 GCC----------------AUAGUCCAAGGUAUCAUCGCCCACUAGUAGCGGUGCUGGUGGCUCUAGCGCUGCACUAACAAAUCCUUUUCUGUCGUCACCGCCAGCCUCGCAGGCUGGU ((.----------------((((.....(((......)))...((((.((((((((((.....))))))))))))))............)))).))....(((((((.....))))))) ( -35.50) >DroMoj_CAF1 6354 102 + 1 -----------------CAUCAGUCCAAGGUAUCGUCGCCCACUAGCAGCGGUGCUGGUGGUGCUAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAGGCUGGU -----------------...........(((..((..((......)).((((((((((.....))))))))))....................))..)))(((((((.....))))))) ( -36.80) >DroAna_CAF1 4399 94 + 1 -------------------CUAGUCCAAGGUAUCGUCGCCCACCAGUAGCGGAGCUGCUGGC------GCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAGGCUGGC -------------------.((((.((.(((......)))(.((((((((...)))))))).------).)).)))).......................(((((((.....))))))) ( -30.90) >consensus ___________________CCAGUCCAAGGUAUCGUCGCCCACCAGUAGCGGUGCUGCUGGCGCUAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCGCAGGCUGGU ............................(((..((..((......)).(((((((((.......)))))))))....................))..)))(((((((.....))))))) (-27.44 = -27.88 + 0.45)

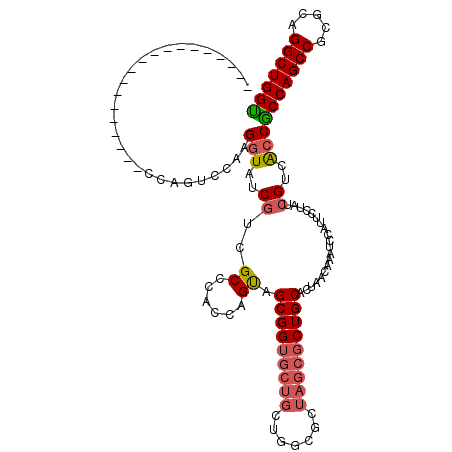
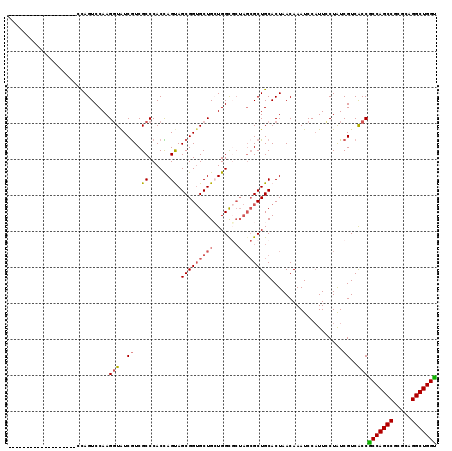
| Location | 3,018,458 – 3,018,578 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.61 |
| Mean single sequence MFE | -54.12 |
| Consensus MFE | -46.52 |
| Energy contribution | -46.63 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.58 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.80 |
| SVM RNA-class probability | 0.997122 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
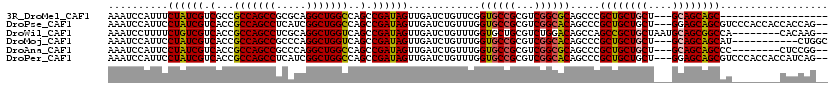
>3R_DroMel_CAF1 3018458 120 + 27905053 CCACUAGCAGCGGUGCUGCUGGCGCUAGCGCUGCACUAACAAAUCCAUUUCUAUCGUCGCCGCCAGCCGCGCAGGCUGGCCAGCCGAUAGUUGAUCUGUUCGGUGCCGCGUCGGCGCAGC ....(((((((...)))))))((((..((((.(((((((((.(((.....((((((.(...(((((((.....)))))))..).))))))..))).)))).))))).))))..))))... ( -61.60) >DroPse_CAF1 5613 120 + 1 CCACUAGUAGCGGUGCUGCUGGCUCAAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCUCAUCGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGC ....((((.((((((((.........)))))))))))).(((((..(((.((((((.(...(((((((.....)))))))..).))))))..)))..)))))((((((...))))))... ( -49.00) >DroWil_CAF1 7432 120 + 1 CCACUAGUAGCGGUGCUGGUGGCUCUAGCGCUGCACUAACAAAUCCUUUUCUGUCGUCACCGCCAGCCUCGCAGGCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCUGCGUCUGGACAGC (((((((((....))))))))).((((((((.(((((((((.(((.....((((((.(...(((((((.....)))))))..).))))))..))).)).))))))).))).))))).... ( -53.70) >DroMoj_CAF1 6376 120 + 1 CCACUAGCAGCGGUGCUGGUGGUGCUAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAGGCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGC .((((((((....))))))))((((..((((.(((((((((.(((.....((((((.(...(((((((.....)))))))..).))))))..))).)).))))))).))))..))))... ( -59.10) >DroAna_CAF1 4419 114 + 1 CCACCAGUAGCGGAGCUGCUGGC------GCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCGCAGC ...((......)).((.((((((------((.(((((((((.(((.....((((((.(...(((((((.....)))))))..).))))))..))).)).))))))).))))))))))... ( -52.30) >DroPer_CAF1 5673 120 + 1 CCACUAGUAGCGGUGCUGCUGGCUCAAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCUCAUCGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGC ....((((.((((((((.........)))))))))))).(((((..(((.((((((.(...(((((((.....)))))))..).))))))..)))..)))))((((((...))))))... ( -49.00) >consensus CCACUAGUAGCGGUGCUGCUGGCUCUAGCGCUGCACUAACAAAUCCAUUCCUAUCGUCACCGCCAGCCGCACAGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGC ((.((((((((...)))))))).....((((.(((((((((.(((.....((((((.(...(((((((.....)))))))..).))))))..))).)))).))))).)))).))...... (-46.52 = -46.63 + 0.11)

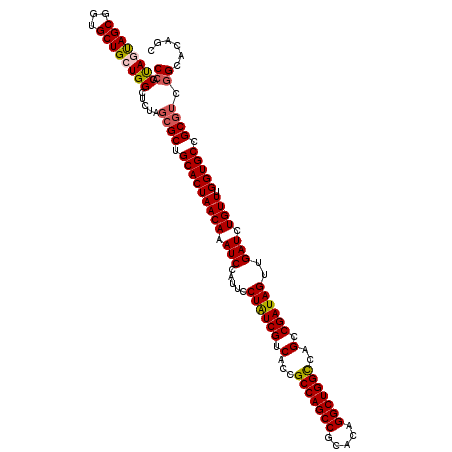
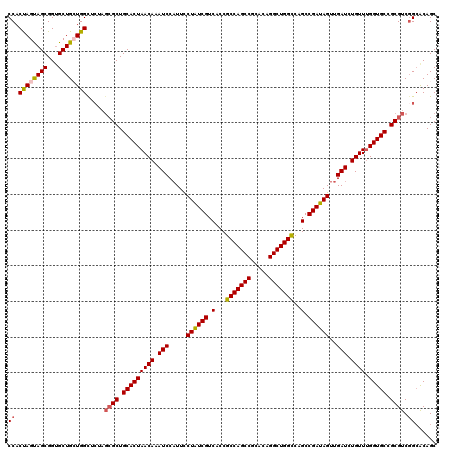
| Location | 3,018,458 – 3,018,578 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.61 |
| Mean single sequence MFE | -55.43 |
| Consensus MFE | -50.14 |
| Energy contribution | -50.56 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.25 |
| Structure conservation index | 0.90 |
| SVM decision value | 3.47 |
| SVM RNA-class probability | 0.999264 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3018458 120 - 27905053 GCUGCGCCGACGCGGCACCGAACAGAUCAACUAUCGGCUGGCCAGCCUGCGCGGCUGGCGGCGACGAUAGAAAUGGAUUUGUUAGUGCAGCGCUAGCGCCAGCAGCACCGCUGCUAGUGG (((((((((...))))....((((((((..((((((((((.((((((.....))))))))))..)))))).....))))))))...))))).....(((.((((((...)))))).))). ( -58.50) >DroPse_CAF1 5613 120 - 1 GCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCGAUGAGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUUGAGCCAGCAGCACCGCUACUAGUGG ((((.((((((((.((((..((((((((..((((((..(.(((((((.....))))))).)...)))))).....)))))))).)))).))).))).))))))....((((.....)))) ( -58.50) >DroWil_CAF1 7432 120 - 1 GCUGUCCAGACGCAGCACCAAACAGAUCAACUAUCGGCUGACCAGCCUGCGAGGCUGGCGGUGACGACAGAAAAGGAUUUGUUAGUGCAGCGCUAGAGCCACCAGCACCGCUACUAGUGG ((((((.((.(((.((((..((((((((..((.(((((((.((((((.....))))))))))..))).)).....)))))))).)))).))))).)).....)))).((((.....)))) ( -46.90) >DroMoj_CAF1 6376 120 - 1 GCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGACCAGCCUGGGCGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUAGCACCACCAGCACCGCUGCUAGUGG ...((((...(((.((((..((((((((..((((((((((.((((((.....))))))))))..)))))).....)))))))).)))).)))...))))(((.((((....)))).))). ( -54.40) >DroAna_CAF1 4419 114 - 1 GCUGCGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCUGGGCGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGC------GCCAGCAGCUCCGCUACUGGUGG (((((((((...))))....((((((((..((((((..(.(((((((.....))))))).)...)))))).....))))))))...)))))------(((((.(((...))).))))).. ( -55.80) >DroPer_CAF1 5673 120 - 1 GCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCGAUGAGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUUGAGCCAGCAGCACCGCUACUAGUGG ((((.((((((((.((((..((((((((..((((((..(.(((((((.....))))))).)...)))))).....)))))))).)))).))).))).))))))....((((.....)))) ( -58.50) >consensus GCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCUGCGAGGCUGGCGGUGACGAUAGGAAUGGAUUUGUUAGUGCAGCGCUAGAGCCAGCAGCACCGCUACUAGUGG (((((.....(((.((((..((((((((..((((((((((.((((((.....))))))))))..)))))).....)))))))).)))).))).........))))).((((.....)))) (-50.14 = -50.56 + 0.42)

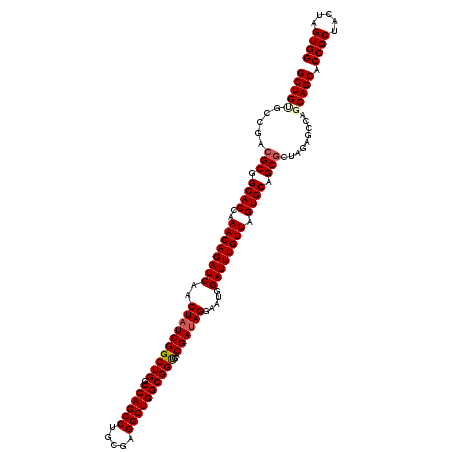
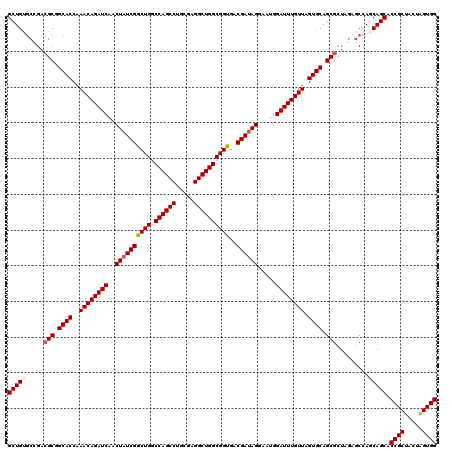
| Location | 3,018,498 – 3,018,597 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.99 |
| Mean single sequence MFE | -43.50 |
| Consensus MFE | -42.42 |
| Energy contribution | -42.23 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967626 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3018498 99 + 27905053 AAAUCCAUUUCUAUCGUCGCCGCCAGCCGCGCAGGCUGGCCAGCCGAUAGUUGAUCUGUUCGGUGCCGCGUCGGCGCAGCCCGCUGCUGCU---GCAGCAGC------------------ .....((...((((((.(...(((((((.....)))))))..).)))))).))........((.((.(((....))).))))(((((((..---.)))))))------------------ ( -43.40) >DroPse_CAF1 5653 115 + 1 AAAUCCAUUCCUAUCGUCACCGCCAGCCUCAUCGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGCCCGCUGCUGCU---GGAGCAGCGUCCCACCACCACCAG-- ..........((((((.(...(((((((.....)))))))..).)))))).....(((....((((((...)))))).(..(((((((...---..)))))))..).........)))-- ( -42.50) >DroWil_CAF1 7472 110 + 1 AAAUCCUUUUCUGUCGUCACCGCCAGCCUCGCAGGCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCUGCGUCUGGACAGCCAGCCGCUGCUAAUGCAGCGGCCA--------CACAAG-- ......((..((((((.(...(((((((.....)))))))..).))))))..)).(((((..(........)..)))))...((((((((....))))))))..--------......-- ( -45.00) >DroMoj_CAF1 6416 106 + 1 AAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAGGCUGGUCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGCCCGCUGCUGCU---GCAGCAGCAU-----------CUGGC ..........((((((.(...(((((((.....)))))))..).))))))............((((((...)))))).(((.(.((((((.---...)))))).-----------).))) ( -44.10) >DroAna_CAF1 4453 107 + 1 AAAUCCAUUCCUAUCGUCACCGCCAGCCGCCCAGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCGCAGCCCGCUGCUGCU---GCAGCAGCCC--------CUCCGG-- ....((....((((((.(...(((((((.....)))))))..).))))))...........((.((.(((....))).))))(((((((..---.)))))))..--------....))-- ( -42.60) >DroPer_CAF1 5713 115 + 1 AAAUCCAUUCCUAUCGUCACCGCCAGCCUCAUCGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGCCCGCUGCUGCU---GGAGCAGCGUCCCACCACCAUCAG-- ..........((((((.(...(((((((.....)))))))..).)))))).....(((..((((((((...))))...(..(((((((...---..)))))))..)....)))).)))-- ( -43.40) >consensus AAAUCCAUUCCUAUCGUCACCGCCAGCCGCACAGGCUGGCCAGCCGAUAGUUGAUCUGUUUGGUGCCGCGUCGGCACAGCCCGCUGCUGCU___GCAGCAGCCU________CACCAG__ ..........((((((.(...(((((((.....)))))))..).))))))............((((((...)))))).....((((((((....)))))))).................. (-42.42 = -42.23 + -0.19)
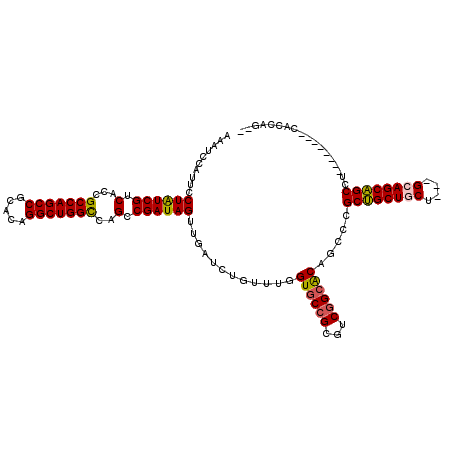
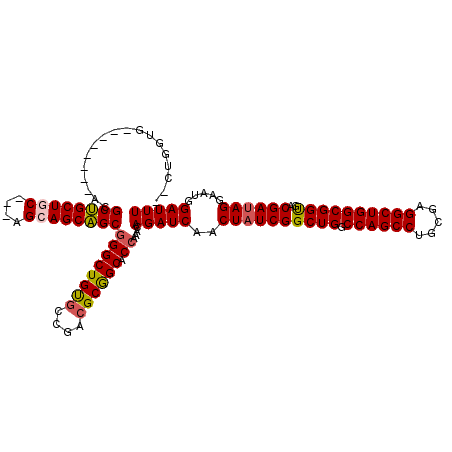
| Location | 3,018,498 – 3,018,597 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.99 |
| Mean single sequence MFE | -50.30 |
| Consensus MFE | -48.88 |
| Energy contribution | -48.93 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.97 |
| SVM decision value | 2.30 |
| SVM RNA-class probability | 0.992000 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 3018498 99 - 27905053 ------------------GCUGCUGC---AGCAGCAGCGGGCUGCGCCGACGCGGCACCGAACAGAUCAACUAUCGGCUGGCCAGCCUGCGCGGCUGGCGGCGACGAUAGAAAUGGAUUU ------------------(((((((.---..)))))))((((((((....)))))).))....(((((..((((((((((.((((((.....))))))))))..)))))).....))))) ( -49.80) >DroPse_CAF1 5653 115 - 1 --CUGGUGGUGGUGGGACGCUGCUCC---AGCAGCAGCGGGCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCGAUGAGGCUGGCGGUGACGAUAGGAAUGGAUUU --(((.(((((.((.(.(((((((..---...)))))))(((...)))..).)).)))))..)))(((..((((((..(.(((((((.....))))))).)...)))))).....))).. ( -52.70) >DroWil_CAF1 7472 110 - 1 --CUUGUG--------UGGCCGCUGCAUUAGCAGCGGCUGGCUGUCCAGACGCAGCACCAAACAGAUCAACUAUCGGCUGACCAGCCUGCGAGGCUGGCGGUGACGACAGAAAAGGAUUU --.(((.(--------((((((((((....))))))))).(((((......))))))))))..(((((..((.(((((((.((((((.....))))))))))..))).)).....))))) ( -46.40) >DroMoj_CAF1 6416 106 - 1 GCCAG-----------AUGCUGCUGC---AGCAGCAGCGGGCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGACCAGCCUGGGCGGCUGGCGGUGACGAUAGGAAUGGAUUU (((..-----------.((((((...---.))))))...))).((((((...)))))).....(((((..((((((((((.((((((.....))))))))))..)))))).....))))) ( -50.40) >DroAna_CAF1 4453 107 - 1 --CCGGAG--------GGGCUGCUGC---AGCAGCAGCGGGCUGCGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCUGGGCGGCUGGCGGUGACGAUAGGAAUGGAUUU --((....--------))(((((((.---..)))))))((((((((....)))))).))....(((((..((((((..(.(((((((.....))))))).)...)))))).....))))) ( -50.30) >DroPer_CAF1 5713 115 - 1 --CUGAUGGUGGUGGGACGCUGCUCC---AGCAGCAGCGGGCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCGAUGAGGCUGGCGGUGACGAUAGGAAUGGAUUU --....(((((.((.(.(((((((..---...)))))))(((...)))..).)).)))))...(((((..((((((..(.(((((((.....))))))).)...)))))).....))))) ( -52.20) >consensus __CUGGUG________ACGCUGCUGC___AGCAGCAGCGGGCUGUGCCGACGCGGCACCAAACAGAUCAACUAUCGGCUGGCCAGCCUGCGAGGCUGGCGGUGACGAUAGGAAUGGAUUU ..................((((((((....))))))))((((((((....)))))).))....(((((..((((((((((.((((((.....))))))))))..)))))).....))))) (-48.88 = -48.93 + 0.06)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:54:50 2006