
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 23,645,154 – 23,645,288 |
| Length | 134 |
| Max. P | 0.837611 |

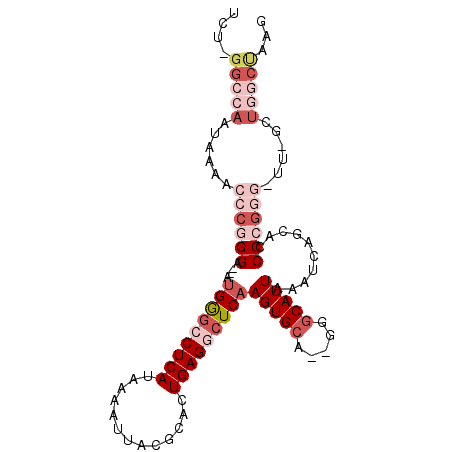
| Location | 23,645,154 – 23,645,249 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 84.20 |
| Mean single sequence MFE | -35.80 |
| Consensus MFE | -21.14 |
| Energy contribution | -24.05 |
| Covariance contribution | 2.92 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.89 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.74 |
| SVM RNA-class probability | 0.837611 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 23645154 95 - 27905053 UCU-GGCCAAUAAAACCCGGGA--AUGGGCCUCAUAAAAUUACGCACUGAGCCUCAAGUGCA--GGGCAUUAAAUCAGCACCCCAGG-UUGGCUGGCUAAG ..(-(((((....((((.(((.--.((((.((((.............)))).)))).((((.--((........)).))))))).))-))...)))))).. ( -29.12) >DroPse_CAF1 109773 100 - 1 GCUGGGCCAAUAAAACCCGGGAAAACGGGGCUCAUAAAAUUACGCACUGAGGCUCAAGUGCAAUGGCCAUUAAAUCCACACCCGGAG-UCUGGUCUCCGGA ....(((((......((((......))))..............(((((........)))))..))))).............((((((-......)))))). ( -36.20) >DroSec_CAF1 107004 94 - 1 UCU-GGCCAAUAAAACCCGGGA--AUGAGCCUCAUAAAAUUACGCACUGAGGCUCAAGUGCA--GGGCAUUAAAUCAGCACCCCGGG-UU-GCUGGCUAAG ..(-(((((....((((((((.--.(((((((((.............))))))))).((((.--((........)).))))))))))-))-..)))))).. ( -42.92) >DroSim_CAF1 105598 94 - 1 UCU-GGCCAAUAAAACCCGGGA--AUGGGCCUCAUAAAAUUACGCACUGAGGCUCAAGUGCA--GGGCAUUAAAUCAGCACCCCGGG-UU-GCUGGCUAAG ..(-(((((....((((((((.--.(((((((((.............))))))))).((((.--((........)).))))))))))-))-..)))))).. ( -42.02) >DroYak_CAF1 110986 95 - 1 UCU-GGCCAAUAAAACCCGGGA--AUGGACCUCAUAAAAUUACGCACUGAGGCUCAAGUGCA--GGGCAUUAAAUCAGCACCCCGGUUUU-GCUGGCUAAG ..(-(((((.(((((((.(((.--.((..(((((.............)))))..)).((((.--((........)).)))))))))))))-).)))))).. ( -33.82) >DroAna_CAF1 108132 79 - 1 --U-GGCUAAUAAAAC-CGGGA--AUGGGCCUCAUAAAAUUACGCACUGAGGCUCAAGUGCU--GGGCAUUAAAUCAGCACCCGGAU-------------- --.-...........(-((((.--.(((((((((.............)))))))))..((((--((........)))))))))))..-------------- ( -30.72) >consensus UCU_GGCCAAUAAAACCCGGGA__AUGGGCCUCAUAAAAUUACGCACUGAGGCUCAAGUGCA__GGGCAUUAAAUCAGCACCCCGGG_UU_GCUGGCUAAG ....(((((......((((((....(((((((((.............)))))))))(((((.....)))))..........))))))......)))))... (-21.14 = -24.05 + 2.92)


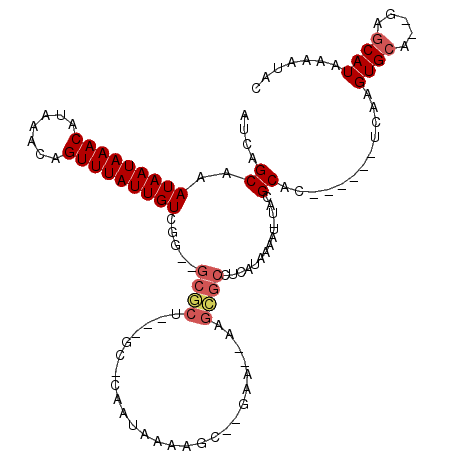
| Location | 23,645,177 – 23,645,288 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.53 |
| Mean single sequence MFE | -26.49 |
| Consensus MFE | -9.85 |
| Energy contribution | -10.35 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.05 |
| Mean z-score | -3.24 |
| Structure conservation index | 0.37 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.563655 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 23645177 111 - 27905053 AUCAGCAAAUAAUAAACAUAAACAGUUUAUUGUCGGC-GCUCU-G-GC-CAAUAAAACCCGGGA--AUGGGCCUCAUAAAAU-UACGCACUGAGCCUCAAGUGCA--GGGCAUUAAAUCA ....((..(((((((((.......)))))))))..))-(((((-(-((-(..............--...)))).........-...(((((........))))))--))))......... ( -30.13) >DroVir_CAF1 147038 102 - 1 AUCAGCAAAUAAUAAACAUAAACAGUUUAUUGUCGG--GCGCU---GCCCAAUAAAAGC--GAA--AAGCGCCUCAUAAAAU-UACGCAC------UCAAGUGCA--GAGCAUAAAAUAC ....((..(((((((((.......))))))))).((--(((((---.............--...--.)))))))........-...((((------....)))).--..))......... ( -24.13) >DroGri_CAF1 139565 101 - 1 AUCAGCAAAUAAUAAACAUAAACAGUUUAUUGUCGA--GCGCU---GC-UAAUAAAAGC--GAA--AAGCGCCUCAUAAAAU-UACGCAC------UCAAGUGCA--GAGCAUAAAAUAC ....((..(((((((((.......))))))))).((--(((((---((-(......)))--...--.)))).))).......-...((((------....)))).--..))......... ( -23.70) >DroWil_CAF1 124488 108 - 1 AUCAGCAAAUAAUAAACAUAAACCGUUUAUUGUCGAC-GCUGG-GAGC-UAAUAAAAGU--GAA--AAGGCCCUCAUAAAAACGAGGCAACAA--UUCAAGUGCA--ACGCAUAAAC-UG .(((((..(((((((((.......)))))))))....-)))))-..((-......(..(--(((--..(..((((........))))...)..--))))..)...--..))......-.. ( -19.50) >DroMoj_CAF1 156288 101 - 1 AUCAGCAAAUAAUAAACAUAAACAGUUUAUUGUCGG--GCGCU---GC-UAAUAAAAGC--GAA--AAGCGCCUCAUAAAAU-UACGCAC------UCAAGUGCA--GAGCAUAAAAUAC ....((..(((((((((.......))))))))).((--(((((---((-(......)))--...--.)))))))........-...((((------....)))).--..))......... ( -26.30) >DroPer_CAF1 108110 117 - 1 AUCAGCAAAUAAUAAACAUAAACAGUUUAUUGUCGGGGGCGCUGG-GC-CAAUAAAACCCGGGAAAACGGGGCUCAUAAAAU-UACGCACUGAGGCUCAAGUGCAAUGGCCAUUAAAUCC ..((((....(((((((.......)))))))((.....))))))(-((-((......((((......))))...........-...(((((........)))))..)))))......... ( -35.20) >consensus AUCAGCAAAUAAUAAACAUAAACAGUUUAUUGUCGG__GCGCU___GC_CAAUAAAAGC__GAA__AAGCGCCUCAUAAAAU_UACGCAC______UCAAGUGCA__GAGCAUAAAAUAC ....((..(((((((((.......))))))))).....((((..........................))))..............))............((((.....))))....... ( -9.85 = -10.35 + 0.50)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:20:48 2006