
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 22,697,167 – 22,697,260 |
| Length | 93 |
| Max. P | 0.986418 |

| Location | 22,697,167 – 22,697,260 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 64.67 |
| Mean single sequence MFE | -24.55 |
| Consensus MFE | -11.67 |
| Energy contribution | -11.87 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.48 |
| SVM decision value | 2.04 |
| SVM RNA-class probability | 0.986418 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
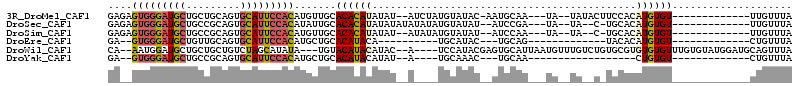
>3R_DroMel_CAF1 22697167 93 + 27905053 GAGAGUGGGAUGCUGCUGCAGUGCAUUCCACAUGUUGCACACAUAUAU--AUCUAUGUAUAC-AAUGCAA---UA--UAUACUUCCACAUGUGU-------------UUGUUUA ....(((((((((.((....)))))))))))((((((((...((((((--....))))))..-..)))))---))--)((((........))))-------------....... ( -25.10) >DroSec_CAF1 4020 91 + 1 GAGAGUGGGAUGCUGCCGCAGUGCAUUCCACAUAUUGCACACAUAUAUAUAUAUAUGUAUAU--AUCCGA---UA--UA--C-UGCACAUGUGU-------------UUGUUUA ....(((((((((.........))))))))).....(((((.(((((....)))))((((((--.....)---))--))--)-......)))))-------------....... ( -22.10) >DroSim_CAF1 3978 89 + 1 GAGAGUGGGAUGCUGCCGCAGUGCAUUCCACAUGUUGCACACAUAUAU--AUAUAUGUAUAU--AUCCAA---UA--UA--C-UGCACAUGUGU-------------UUGUUUA ....(((((((((.........))))))))).....(((.((((((..--......((((((--.....)---))--))--)-.....))))))-------------.)))... ( -22.62) >DroEre_CAF1 4394 73 + 1 GA--GUGGGAUGCUGUUGCAGUGCAUUCCACAUGCUGCACAUACA----------UGCAUAC---UGCAG-------------UACACAUGUGU-------------CUGUUUA ..--(((((((((.........))))))))).....(((.(((((----------((..(((---....)-------------))..)))))))-------------.)))... ( -25.10) >DroWil_CAF1 4770 103 + 1 CA--AAUGGAUGCUGCUGCUGUCUAGCAUAUA---UGUACAUACAUAC--A----UCCAUACGAGUGCAUUAAUGUUUGUCUGUGCGUGUGUGUUUGUGUAUGGAUGCAGUUUA ..--..(((((((....)).)))))((((.((---((((((.((((((--(----(.((((.((..(((....)))...)))))).)))))))).)))))))).))))...... ( -30.10) >DroYak_CAF1 6164 72 + 1 GA--GUGGGAUGCUGCCGCAGUGCAUUCCACAUGCUGCACAUACAUAU--A----UGCAAAC---UGCAA------------------CUGUGU-------------CUGUUUA ..--(((((((((.........))))))))).......(((.(((((.--.----(((....---.))).------------------.)))))-------------.)))... ( -22.30) >consensus GA__GUGGGAUGCUGCCGCAGUGCAUUCCACAUGUUGCACACACAUAU__A____UGCAUAC__AUGCAA___UA__UA__C_UGCACAUGUGU_____________CUGUUUA ....(((((((((.........))))))))).......((((((............................................)))))).................... (-11.67 = -11.87 + 0.20)

| Location | 22,697,167 – 22,697,260 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 64.67 |
| Mean single sequence MFE | -21.80 |
| Consensus MFE | -6.13 |
| Energy contribution | -7.38 |
| Covariance contribution | 1.25 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.28 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.887921 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
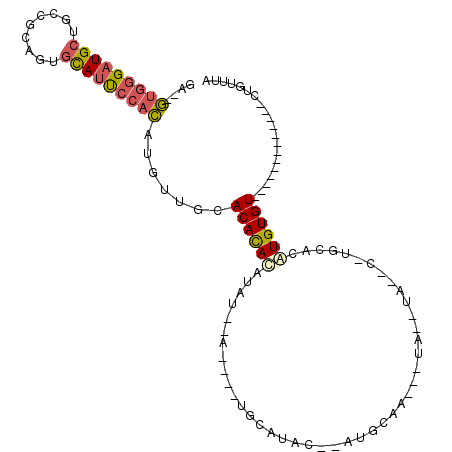
>3R_DroMel_CAF1 22697167 93 - 27905053 UAAACAA-------------ACACAUGUGGAAGUAUA--UA---UUGCAUU-GUAUACAUAGAU--AUAUAUGUGUGCAACAUGUGGAAUGCACUGCAGCAGCAUCCCACUCUC .......-------------.(((((((....(((..--..---.)))..(-(((((((((...--...)))))))))))))))))((.((...(((....)))...)).)).. ( -24.00) >DroSec_CAF1 4020 91 - 1 UAAACAA-------------ACACAUGUGCA-G--UA--UA---UCGGAU--AUAUACAUAUAUAUAUAUAUGUGUGCAAUAUGUGGAAUGCACUGCGGCAGCAUCCCACUCUC .......-------------(((((((((..-(--((--((---(.(...--.....)))))))...))))))))).......((((.((((.........)))).)))).... ( -20.60) >DroSim_CAF1 3978 89 - 1 UAAACAA-------------ACACAUGUGCA-G--UA--UA---UUGGAU--AUAUACAUAUAU--AUAUAUGUGUGCAACAUGUGGAAUGCACUGCGGCAGCAUCCCACUCUC .......-------------.((((((((((-.--((--((---(...((--((((....))))--))..)))))))).)))))))((.((...(((....)))...)).)).. ( -21.80) >DroEre_CAF1 4394 73 - 1 UAAACAG-------------ACACAUGUGUA-------------CUGCA---GUAUGCA----------UGUAUGUGCAGCAUGUGGAAUGCACUGCAACAGCAUCCCAC--UC ...(((.-------------..(((((((((-------------(....---)))))))----------))).))).......((((.((((.........)))).))))--.. ( -21.90) >DroWil_CAF1 4770 103 - 1 UAAACUGCAUCCAUACACAAACACACACGCACAGACAAACAUUAAUGCACUCGUAUGGA----U--GUAUGUAUGUACA---UAUAUGCUAGACAGCAGCAGCAUCCAUU--UG ....((((....................(.(((.(((.(((((.((((....)))).))----)--)).))).))).).---....((((....))))))))........--.. ( -20.30) >DroYak_CAF1 6164 72 - 1 UAAACAG-------------ACACAG------------------UUGCA---GUUUGCA----U--AUAUGUAUGUGCAGCAUGUGGAAUGCACUGCGGCAGCAUCCCAC--UC .......-------------.....(------------------(((((---((.((((----(--((....)))))))((((.....))))))))))))..........--.. ( -22.20) >consensus UAAACAA_____________ACACAUGUGCA_G__UA__UA___UUGCAU__GUAUACA____U__AUAUAUAUGUGCAACAUGUGGAAUGCACUGCAGCAGCAUCCCAC__UC .....................((((((((........................................))))))))......((((.((((.........)))).)))).... ( -6.13 = -7.38 + 1.25)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:12:48 2006