
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 22,057,546 – 22,057,746 |
| Length | 200 |
| Max. P | 0.999240 |

| Location | 22,057,546 – 22,057,666 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.83 |
| Mean single sequence MFE | -42.74 |
| Consensus MFE | -37.54 |
| Energy contribution | -38.22 |
| Covariance contribution | 0.68 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.901427 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
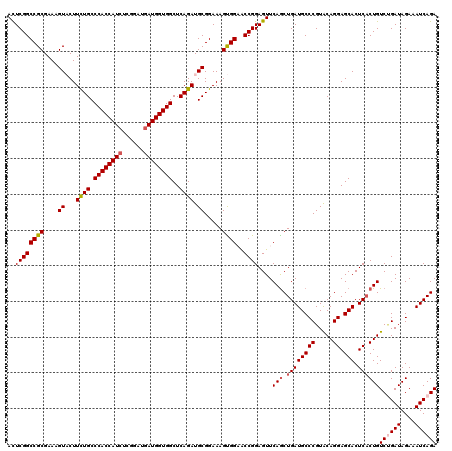
>3R_DroMel_CAF1 22057546 120 + 27905053 ACUCGGCCGCGAAAGUACUUUUGCCCACCAUCUCGGAUGAUGGUGGCUCAGAUGCGGAAAGUGGAGCCGGAGUUCAACUGAUGCCCGUACAGGAGCACUCACUGUCAGAUAGAAAUCAGA ((((((((((....))((((((((((((((((......)))))))).......)).)))))).).))).))))....(((((((((.....)).)).....((((...))))..))))). ( -37.71) >DroSec_CAF1 49619 120 + 1 ACUCGGCCGCGAAAGUACUUCUGCCCACCAUCUCGGAUGAUGGUGGCUCAGAUGCGGAAAGUGGAACCGGAGUUCAGCUGAUGCCCGUACAGGAGCACUCACUGUCUGAUAGAAAUCAGA ((((((((((....(((..((((.((((((((......))))))))..))))))).....))))..)).)))).(((.((((((((.....)).))).))))))((((((....)))))) ( -43.80) >DroSim_CAF1 42303 120 + 1 ACUCGGCCGCGAAAGUACUUCUGCCCACCAUCUCGGAUGAUGGUGGCUCAGAUGCGGAAAGUGGAACCGGAGUUCAGCUGAUGCCCGUACAGGAGCACUCCCUGUCCGAUAGAAAUCAGA ((((((((((....(((..((((.((((((((......))))))))..))))))).....))))..)).))))....(((((...((.(((((.......))))).))......))))). ( -41.20) >DroEre_CAF1 44072 120 + 1 AUUCGGCCGCGAAAGUCCUUCUGCCCACCAUCUCCGAUGAUGGUGGCUCAGAUGCGGAAAGCGGCACCGGAGUUCAGCUGAUGCCCGUGCAGGAGCACUCACUGUCUGAUAGAAAUCAGA .(((((((((.....(((.((((.((((((((......))))))))..))))...)))..)))))..))))...(((.((((((((.....)).))).))))))((((((....)))))) ( -48.50) >DroYak_CAF1 51864 120 + 1 AUUCGGCCGCGAAAGUCCUUCUGCCCACCAUAUCCGAUGAUGGUGGCUCAGAUGCGGAAAGUGGUACCGGAGUUCAGCUGAUGCCCGUGCAGGAGCACUCACUGUCUGAUAGAAAUCAGA .(((((((((.....(((.((((.(((((((........)))))))..))))...)))..))))..)))))...(((.((((((((.....)).))).))))))((((((....)))))) ( -42.50) >consensus ACUCGGCCGCGAAAGUACUUCUGCCCACCAUCUCGGAUGAUGGUGGCUCAGAUGCGGAAAGUGGAACCGGAGUUCAGCUGAUGCCCGUACAGGAGCACUCACUGUCUGAUAGAAAUCAGA ..((((((((....((...((((.((((((((......))))))))..)))).)).....))))..))))....(((.((((((((.....)).))).))))))((((((....)))))) (-37.54 = -38.22 + 0.68)


| Location | 22,057,546 – 22,057,666 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.83 |
| Mean single sequence MFE | -43.23 |
| Consensus MFE | -38.82 |
| Energy contribution | -39.34 |
| Covariance contribution | 0.52 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.66 |
| SVM RNA-class probability | 0.970678 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 22057546 120 - 27905053 UCUGAUUUCUAUCUGACAGUGAGUGCUCCUGUACGGGCAUCAGUUGAACUCCGGCUCCACUUUCCGCAUCUGAGCCACCAUCAUCCGAGAUGGUGGGCAAAAGUACUUUCGCGGCCGAGU ...((.(((...((((..((..((((....))))..)).))))..))).))(((((..................((((((((......))))))))((.(((....))).)))))))... ( -35.20) >DroSec_CAF1 49619 120 - 1 UCUGAUUUCUAUCAGACAGUGAGUGCUCCUGUACGGGCAUCAGCUGAACUCCGGUUCCACUUUCCGCAUCUGAGCCACCAUCAUCCGAGAUGGUGGGCAGAAGUACUUUCGCGGCCGAGU ((((((....))))))((((..((((((......))))))..)))).((((.((.........((((.((((..((((((((......)))))))).)))).(......))))))))))) ( -45.40) >DroSim_CAF1 42303 120 - 1 UCUGAUUUCUAUCGGACAGGGAGUGCUCCUGUACGGGCAUCAGCUGAACUCCGGUUCCACUUUCCGCAUCUGAGCCACCAUCAUCCGAGAUGGUGGGCAGAAGUACUUUCGCGGCCGAGU ...........((((...(..((((((.((((...(((.((((.((.....(((.........))))).))))))).((((((((...)))))))))))).))))))..)....)))).. ( -44.30) >DroEre_CAF1 44072 120 - 1 UCUGAUUUCUAUCAGACAGUGAGUGCUCCUGCACGGGCAUCAGCUGAACUCCGGUGCCGCUUUCCGCAUCUGAGCCACCAUCAUCGGAGAUGGUGGGCAGAAGGACUUUCGCGGCCGAAU ((((((....))))))((((..((((((......))))))..)))).......(.(((((..(((...((((..((((((((......)))))))).)))).))).....)))))).... ( -49.40) >DroYak_CAF1 51864 120 - 1 UCUGAUUUCUAUCAGACAGUGAGUGCUCCUGCACGGGCAUCAGCUGAACUCCGGUACCACUUUCCGCAUCUGAGCCACCAUCAUCGGAUAUGGUGGGCAGAAGGACUUUCGCGGCCGAAU ((((((....))))))((((..((((((......))))))..)))).....((((.......(((...((((..(((((((........))))))).)))).)))........))))... ( -41.86) >consensus UCUGAUUUCUAUCAGACAGUGAGUGCUCCUGUACGGGCAUCAGCUGAACUCCGGUUCCACUUUCCGCAUCUGAGCCACCAUCAUCCGAGAUGGUGGGCAGAAGUACUUUCGCGGCCGAGU ((((((....))))))((((..((((((......))))))..)))).....(((.........((((.((((..((((((((......)))))))).)))).(......))))))))... (-38.82 = -39.34 + 0.52)



| Location | 22,057,586 – 22,057,706 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.83 |
| Mean single sequence MFE | -34.65 |
| Consensus MFE | -29.06 |
| Energy contribution | -29.78 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.46 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.713538 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 22057586 120 - 27905053 UUUAUUUCACCACUGGCUGUUUGCAGAGUAAGCAUAAUGUUCUGAUUUCUAUCUGACAGUGAGUGCUCCUGUACGGGCAUCAGUUGAACUCCGGCUCCACUUUCCGCAUCUGAGCCACCA .....((((..(((((.(((((((((((((..(((..((((..(((....))).)))))))..)))).)))))..)))))))))))))....(((((..............))))).... ( -30.04) >DroSec_CAF1 49659 120 - 1 UUGAUCUCACCACUCGCUGUUUGCAGAGUCAGCAUAAUGUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGUACGGGCAUCAGCUGAACUCCGGUUCCACUUUCCGCAUCUGAGCCACCA ..........(((((((((((((.((((((((.(......))))).))))..)))))))))))))..........(((.((((.((.....(((.........))))).))))))).... ( -35.50) >DroSim_CAF1 42343 120 - 1 UUGAUCUCACCACUGGCUGUUUGCAGAGUCAGCAUAAUGUUCUGAUUUCUAUCGGACAGGGAGUGCUCCUGUACGGGCAUCAGCUGAACUCCGGUUCCACUUUCCGCAUCUGAGCCACCA .............(((((...(((.((((((((...((((((.(((....)))(.((((((.....)))))).)))))))..)))).)))).((.........)))))....)))))... ( -33.80) >DroEre_CAF1 44112 120 - 1 UUGAUCUCACCACUGGCUGUUUGCAGAGUCAGCAUAAUAUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGCACGGGCAUCAGCUGAACUCCGGUGCCGCUUUCCGCAUCUGAGCCACCA ((((((((..(((((((((..........)))).......((((((....))))))))))).((((....))))))).))))).....(((.(((((........))))).)))...... ( -37.40) >DroYak_CAF1 51904 120 - 1 UUGAUCUCACCACUGGCCGUUUGCAGAGUCAGCAUAAUAUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGCACGGGCAUCAGCUGAACUCCGGUACCACUUUCCGCAUCUGAGCCACCA .............((((((..(((.((((((((.......((((((....))))))..((..((((....))))..))....)))).)))).((.........)))))..)).))))... ( -36.50) >consensus UUGAUCUCACCACUGGCUGUUUGCAGAGUCAGCAUAAUGUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGUACGGGCAUCAGCUGAACUCCGGUUCCACUUUCCGCAUCUGAGCCACCA .............(((((...(((.((((((((.......((((((....))))))...(((.(((((......)))))))))))).)))).((.........)))))....)))))... (-29.06 = -29.78 + 0.72)


| Location | 22,057,626 – 22,057,746 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.25 |
| Mean single sequence MFE | -41.34 |
| Consensus MFE | -37.18 |
| Energy contribution | -36.58 |
| Covariance contribution | -0.60 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.90 |
| SVM decision value | 3.46 |
| SVM RNA-class probability | 0.999240 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
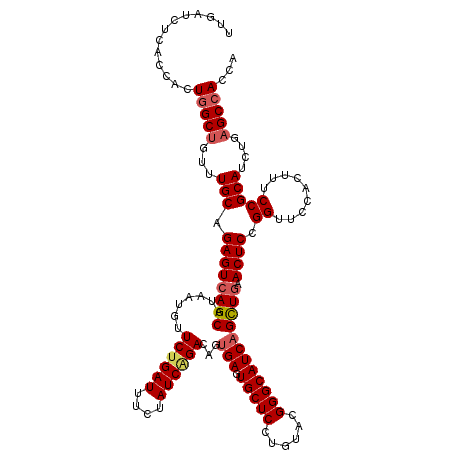
>3R_DroMel_CAF1 22057626 120 + 27905053 AUGCCCGUACAGGAGCACUCACUGUCAGAUAGAAAUCAGAACAUUAUGCUUACUCUGCAAACAGCCAGUGGUGAAAUAAAGAGGAACUACUCCGCCAGACAGCUGCCCCUGUUGGGCACU .((((((.(((((.(((....(((((....................(((.......))).........(((((.......(((......))))))))))))).))).))))))))))).. ( -37.21) >DroSec_CAF1 49699 120 + 1 AUGCCCGUACAGGAGCACUCACUGUCUGAUAGAAAUCAGAACAUUAUGCUGACUCUGCAAACAGCGAGUGGUGAGAUCAAGAGGAACUACUCCGCUAGGCAGCUGCCCCUGUUGGGCACU .((((((.((((((((.(((((..((((((....)))))).((((.(((((..........)))))))))))))).....(((......))).))).(((....)))))))))))))).. ( -47.20) >DroSim_CAF1 42383 120 + 1 AUGCCCGUACAGGAGCACUCCCUGUCCGAUAGAAAUCAGAACAUUAUGCUGACUCUGCAAACAGCCAGUGGUGAGAUCAAGAGGAACUACUCCGCCAGACAGCUGCCCCUGUUGGGCACU .((((((.(((((.(((....(((((.(...((..(((...((((..((((..........)))).)))).)))..))..(((......)))...).))))).))).))))))))))).. ( -38.20) >DroEre_CAF1 44152 120 + 1 AUGCCCGUGCAGGAGCACUCACUGUCUGAUAGAAAUCAGAAUAUUAUGCUGACUCUGCAAACAGCCAGUGGUGAGAUCAAGAGGAACUACUCCGCCAGGCAGCUGCCCCUGUUGGGCACU .((((((.(((((.((.((((((.((((((....)))))).......((((...(((....))).)))))))))).....(((......))).))..(((....)))))))))))))).. ( -43.40) >DroYak_CAF1 51944 120 + 1 AUGCCCGUGCAGGAGCACUCACUGUCUGAUAGAAAUCAGAAUAUUAUGCUGACUCUGCAAACGGCCAGUGGUGAGAUCAAGAGGAACUACUCCGCCAGGCAGCUGCCCUUGUUGGGCACU .((((.(.((.(((((((.(((((((((((....)))))).......((((..........))))))))))))((.((.....)).)).))))))).))))..(((((.....))))).. ( -40.70) >consensus AUGCCCGUACAGGAGCACUCACUGUCUGAUAGAAAUCAGAACAUUAUGCUGACUCUGCAAACAGCCAGUGGUGAGAUCAAGAGGAACUACUCCGCCAGGCAGCUGCCCCUGUUGGGCACU .((((((.(((((.(((....(((((((.............((((((((((..........))))..)))))).......(((......)))...))))))).))).))))))))))).. (-37.18 = -36.58 + -0.60)



| Location | 22,057,626 – 22,057,746 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.25 |
| Mean single sequence MFE | -46.52 |
| Consensus MFE | -39.82 |
| Energy contribution | -40.54 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.91 |
| Structure conservation index | 0.86 |
| SVM decision value | 3.35 |
| SVM RNA-class probability | 0.999060 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 22057626 120 - 27905053 AGUGCCCAACAGGGGCAGCUGUCUGGCGGAGUAGUUCCUCUUUAUUUCACCACUGGCUGUUUGCAGAGUAAGCAUAAUGUUCUGAUUUCUAUCUGACAGUGAGUGCUCCUGUACGGGCAU .((((((.((((((((((((((((((.(((((((.......))))))).))).....(((((((...)))))))....................))))))...)))))))))..)))))) ( -44.70) >DroSec_CAF1 49699 120 - 1 AGUGCCCAACAGGGGCAGCUGCCUAGCGGAGUAGUUCCUCUUGAUCUCACCACUCGCUGUUUGCAGAGUCAGCAUAAUGUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGUACGGGCAU .((((((.(((((((((((((((.....).))))))...............((((((((((((.((((((((.(......))))).))))..))))))))))))))))))))..)))))) ( -50.50) >DroSim_CAF1 42383 120 - 1 AGUGCCCAACAGGGGCAGCUGUCUGGCGGAGUAGUUCCUCUUGAUCUCACCACUGGCUGUUUGCAGAGUCAGCAUAAUGUUCUGAUUUCUAUCGGACAGGGAGUGCUCCUGUACGGGCAU .((((((.(((((((((...(((.((.((((...)))).)).)))(((.((.((((((........)))))).....(((.(((((....))))))))))))))))))))))..)))))) ( -47.00) >DroEre_CAF1 44152 120 - 1 AGUGCCCAACAGGGGCAGCUGCCUGGCGGAGUAGUUCCUCUUGAUCUCACCACUGGCUGUUUGCAGAGUCAGCAUAAUAUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGCACGGGCAU .((((((..((((((.(((((((.....).)))))).)))))).......(((((((((..........)))).......((((((....))))))))))).((((....)))))))))) ( -46.50) >DroYak_CAF1 51944 120 - 1 AGUGCCCAACAAGGGCAGCUGCCUGGCGGAGUAGUUCCUCUUGAUCUCACCACUGGCCGUUUGCAGAGUCAGCAUAAUAUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGCACGGGCAU ..(((((.....)))))..((((((((((((((..((.....)).(((((..((((((.......).)))))........((((((....))))))..))))))))))).)).)))))). ( -43.90) >consensus AGUGCCCAACAGGGGCAGCUGCCUGGCGGAGUAGUUCCUCUUGAUCUCACCACUGGCUGUUUGCAGAGUCAGCAUAAUGUUCUGAUUUCUAUCAGACAGUGAGUGCUCCUGUACGGGCAU .((((((..(((((((.(((....)))((((......))))..........(((.((((((((.((((((((.(......))))).))))..)))))))).))))))))))...)))))) (-39.82 = -40.54 + 0.72)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 13:05:46 2006