
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 21,420,770 – 21,420,980 |
| Length | 210 |
| Max. P | 0.960285 |

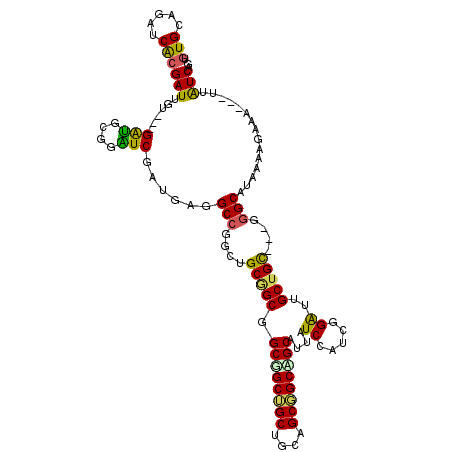
| Location | 21,420,770 – 21,420,878 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 78.29 |
| Mean single sequence MFE | -43.09 |
| Consensus MFE | -27.01 |
| Energy contribution | -25.93 |
| Covariance contribution | -1.08 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.737489 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 21420770 108 - 27905053 GGUGGAGAUCGCGAUUGU---GGUGCGGAUCAAUCAAGCCCGCUGCUGCGGCGGCGGCUGCUGCGGCCGCGUUCUCCAUGGGAUUGCUGC---GUGCAUAAAAGAAA---CUAUCAG .(((((((.((((...((---((.((...........)))))).((((((((((...)))))))))))))).))))))).....(((...---..))).........---....... ( -43.20) >DroVir_CAF1 16059 111 - 1 UGUGCAGAUCGCGAUUGU---GAUGCGGAUCAAUUAGGCCGGCAGCGGCAGCGGCUGCUGCGGCGGCAGCAUUAUCAAUGGGAUUGCUGU---GGGCGGUAAAAAAAUAUUUGUCAG ((.(((((((.(.(((((---(((((...........((((((((((((....))))))))..)))).)))))).)))).)))((((((.---...)))))).......))))))). ( -38.70) >DroGri_CAF1 15243 114 - 1 UGUGCAGCUCACGAUUGU---GAUGCGGAUCGAUUAGGCCAGCGGCGGCUGCGGCUGCGGCAGCGGCAGCAUUAUCGAUCGGAUUGCUGUGAUGGGCAUUAAAGAAAUAUUUAUCAG ...(((((...(((((((---(((((((.((.....)))).((.((.(((((....))))).)).)).)))))).))))))....)))))((((((.(((.....))).)))))).. ( -42.80) >DroMoj_CAF1 15964 111 - 1 UGUGCAGAUCACGAUUGU---GAUGCGGAUCGAUGAGGCCGGCUGCGGCGGCUGCUGCGGCAGCGGCAGCAUUAUCAAUCGGAUUGCUGU---GGGCAUUAAAGAAAUAUUUGUCAG ...((((.((.(((((((---(((((....(.....)((((((((((((....))))))))..)))).)))))).))))))))))))...---.((((.............)))).. ( -41.12) >DroAna_CAF1 108489 108 - 1 GGUGCAAGUCGCGAUUGU---GGCGCGGGUCGAUGAGGCCCGCUGCAGCGGCGGCGGCAGCGGCUGCUGCGUUCUCCAUCGGGUUGCUGC---GGGCAUAGAAAAAA---UUGUCAG .((((..((.((((((((---((.(((((((.....))))))).((((((((.((....)).)))))))).....))))..)))))).))---..))))........---....... ( -52.00) >DroPer_CAF1 15972 111 - 1 UGUGCAGAUCAGGAUGGUGAUGGUGUGGGUCGAUGAGGCCGGCGGCGGCGGCUGCUGCUGCAGCGGCAGCGUUAUCCAUGGGAUUGCUGC---GGGCAAAAAAGAAG---GUAUCGG ..(((((.(((((((..((.((.(((.((((.....)))).))).))((.(((((....))))).))..))..)))).))).....))))---).............---....... ( -40.70) >consensus UGUGCAGAUCACGAUUGU___GAUGCGGAUCGAUGAGGCCGGCUGCGGCGGCGGCUGCUGCAGCGGCAGCAUUAUCCAUCGGAUUGCUGC___GGGCAUAAAAGAAA___UUAUCAG .(((.....)))(((......(((....)))......(((....(((((.((((((((....))))))))....((.....))..)))))....)))...............))).. (-27.01 = -25.93 + -1.08)


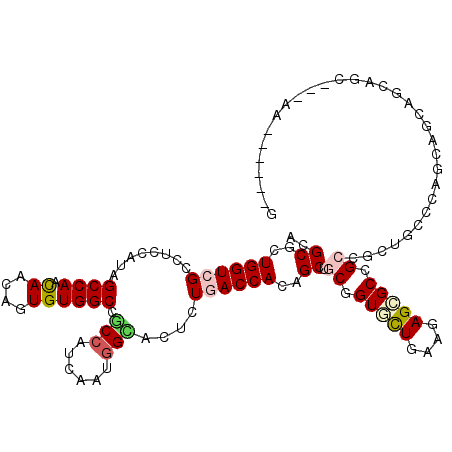
| Location | 21,420,878 – 21,420,980 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 70.85 |
| Mean single sequence MFE | -43.78 |
| Consensus MFE | -19.75 |
| Energy contribution | -20.95 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.88 |
| Structure conservation index | 0.45 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.846295 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 21420878 102 + 27905053 AGGCGCUGGUGGCCUCGAUAGCCAAUAAUAGUGUGGCCGCCAUCGGCGGUGGCCUGACCACAGCGGCAGUCUUGAAGAGUGCCG------CUCAGCAGUCCCAGC---AG------G ....(((((((((.......))))......(((.(((((((......)))))))....)))(((((((.((.....)).)))))------))........)))))---..------. ( -45.60) >DroPse_CAF1 16155 114 + 1 ACGCCCUGGUCGCUUCCAUAGCCAACAACAGUGUGGCGGCCAUCAAUGGCACUCUGACCACGGCUGCCGUGCUAAAGAGCGCCGCCGCUGCCCAGCAGCAGCAGC---AAUCCCAGG ....(((((..((((...(((((.(((....)))(((((((.....((((.....).))).)))))))).))))..))))((.((.(((((...))))).)).))---....))))) ( -44.10) >DroWil_CAF1 120605 114 + 1 AUGCGCUGGUCGCCUCCAUAGCCAAUAGCAAUGUGGCUGCCAUCAAUGGAACUCUGACCACAGCGGCGGUAUUGAAAAGUGCUGCUGCAGUGCAGCAACAGCAGCAACAAUCCC--- .(((((((((((..((((((((((.((....))))))).......)))))....)))))...(((((((((((....)))))))))))))))))((....))............--- ( -44.11) >DroMoj_CAF1 16075 99 + 1 AUGCGCUGGUCGCCUCCAUAGCCAACAGCAAUGUGGCCGCCAUCAAUGGCACUCUGACCACGGCCGCGGUGCUGAAGAGCGCUGCCGCUGCCCAGGC------------------GG ...((((((((.........((((.((....)))))).((((....)))).....)))).((((.((((((((....)))))))).))))....)))------------------). ( -43.60) >DroAna_CAF1 108597 102 + 1 AGGCUCUGGUGGCCUCCAUGGCCAACAAUGGUGUGGCCACCAUCGGCAGUGGACUGACCACAGCUGCGGUCCUCAAAAGCGCCG------CCCAACAGUCACAGG---CG------G .(((..((((((((.((((........))))...))))))))..(((.((((.....)))).(((...(....)...)))))))------)).....((.....)---).------. ( -39.90) >DroPer_CAF1 16083 114 + 1 ACGCCCUGGUCGCUUCCAUAGCCAACAACAGUGUGGCGGCCAUCAAUGGCACUCUGACCACGGCUGCCGUGCUCAAGAGCGCCGCCGCUGCCCAGCAGCAGCAGC---AAUCCCAGG ....(((((..(((................(.((((((((((....)))).((((((.(((((...))))).)).)))))))))))(((((......))))))))---....))))) ( -45.40) >consensus ACGCGCUGGUCGCCUCCAUAGCCAACAACAGUGUGGCCGCCAUCAAUGGCACUCUGACCACAGCUGCGGUGCUGAAGAGCGCCGCCGCUGCCCAGCAGCAGCAGC___AA______G ..((..((((((........((((.((....)))))).(((......)))....))))))..)).((.(((((....))))).))................................ (-19.75 = -20.95 + 1.20)

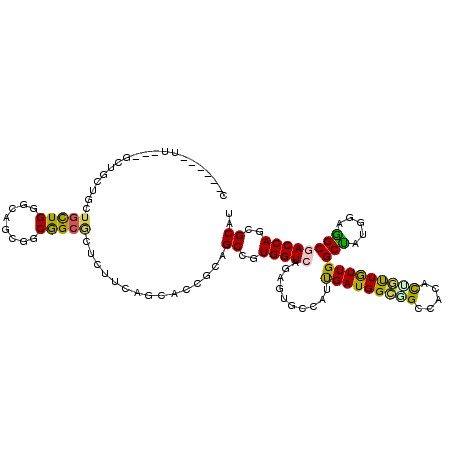
| Location | 21,420,878 – 21,420,980 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 70.85 |
| Mean single sequence MFE | -50.15 |
| Consensus MFE | -19.50 |
| Energy contribution | -18.50 |
| Covariance contribution | -1.00 |
| Combinations/Pair | 1.50 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.39 |
| SVM decision value | 1.51 |
| SVM RNA-class probability | 0.960285 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 21420878 102 - 27905053 C------CU---GCUGGGACUGCUGAG------CGGCACUCUUCAAGACUGCCGCUGUGGUCAGGCCACCGCCGAUGGCGGCCACACUAUUAUUGGCUAUCGAGGCCACCAGCGCCU .------..---((((((((..(..((------(((((.((.....)).))))))))..))).((((..((((...))))((((.........))))......)))).))))).... ( -48.70) >DroPse_CAF1 16155 114 - 1 CCUGGGAUU---GCUGCUGCUGCUGGGCAGCGGCGGCGCUCUUUAGCACGGCAGCCGUGGUCAGAGUGCCAUUGAUGGCCGCCACACUGUUGUUGGCUAUGGAAGCGACCAGGGCGU (((((....---(((((((((((...)))))))))))(((((.((((.(((((((.(((((....(.((((....))))))))))...))))))))))).)).)))..))))).... ( -58.80) >DroWil_CAF1 120605 114 - 1 ---GGGAUUGUUGCUGCUGUUGCUGCACUGCAGCAGCACUUUUCAAUACCGCCGCUGUGGUCAGAGUUCCAUUGAUGGCAGCCACAUUGCUAUUGGCUAUGGAGGCGACCAGCGCAU ---((.((((....(((((((((......))))))))).....)))).))((.(((..((((.(..((((((..(((((((.....)))))))..)...))))).)))))))))).. ( -43.00) >DroMoj_CAF1 16075 99 - 1 CC------------------GCCUGGGCAGCGGCAGCGCUCUUCAGCACCGCGGCCGUGGUCAGAGUGCCAUUGAUGGCGGCCACAUUGCUGUUGGCUAUGGAGGCGACCAGCGCAU ..------------------((((((.(((((((((.(((....)))...(.((((((.(((((.......))))).)))))).).)))))))))(((.....)))..)))).)).. ( -43.80) >DroAna_CAF1 108597 102 - 1 C------CG---CCUGUGACUGUUGGG------CGGCGCUUUUGAGGACCGCAGCUGUGGUCAGUCCACUGCCGAUGGUGGCCACACCAUUGUUGGCCAUGGAGGCCACCAGAGCCU (------((---((((.......))))------))).(((((.(..((((((....))))))..((((..(((((..((((.....))))..)))))..)))).....).))))).. ( -44.60) >DroPer_CAF1 16083 114 - 1 CCUGGGAUU---GCUGCUGCUGCUGGGCAGCGGCGGCGCUCUUGAGCACGGCAGCCGUGGUCAGAGUGCCAUUGAUGGCCGCCACACUGUUGUUGGCUAUGGAAGCGACCAGGGCGU (((((..((---(((((((((((...))))))))((((((((.((.(((((...))))).)))))))))).((.(((((((.((......)).))))))).)))))))))))).... ( -62.00) >consensus C______UU___GCUGCUGCUGCUGGGCAGCGGCGGCGCUCUUCAGCACCGCAGCCGUGGUCAGAGUGCCAUUGAUGGCGGCCACACUGUUGUUGGCUAUGGAGGCGACCAGCGCAU ....................(((((........)))))...............((..(((((..........(((((((((.....)))))))))(((.....))))))))..)).. (-19.50 = -18.50 + -1.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:59:47 2006