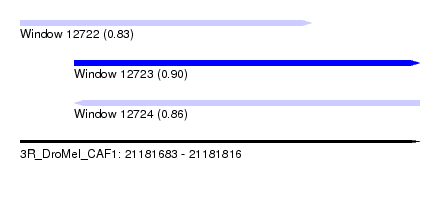
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 21,181,683 – 21,181,816 |
| Length | 133 |
| Max. P | 0.903874 |

| Location | 21,181,683 – 21,181,780 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | forward |
| Mean pairwise identity | 76.33 |
| Mean single sequence MFE | -38.33 |
| Consensus MFE | -26.52 |
| Energy contribution | -26.38 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.827224 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
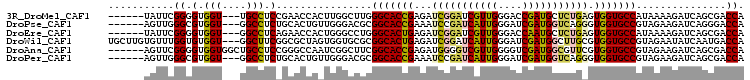
>3R_DroMel_CAF1 21181683 97 + 27905053 ------UAUUCGGGGUGGU---UGCCUCCGAACCACUUGGCUUGGGCACCGAGAUCGGAUCGUUGGGACCGAUGCUCUGAGUGGUGCCAUAAAAGAUCAGCGACCA ------.........((((---(((...(((.((....)).)))(((((((...(((((.(((((....))))).))))).)))))))...........))))))) ( -38.00) >DroPse_CAF1 7881 97 + 1 ------AGUUGGGCGUGGU---GGCCUCUGCACUGUUGGGACGCGGCACCGAAAUCCGAUCAUUGGGAUCGAUGGUCAGGGUGGUGCCGUAGAAGAUCAGGGACCA ------.((.((((.....---.))))..)).....(((..(((((((((...((((((((((((....)))))))).)))))))))))).(.....)..)..))) ( -37.40) >DroEre_CAF1 8315 97 + 1 ------UAUUCGGGGUGGU---GGCCUCAGAACCACUGGGCCUGGGCACUGAGAUCGGAUCGUUGGGACCAAUGCUCUGAGUGGUGCCAUAAAAGAUCAGCGACCA ------.......(((.((---((((.(((.....)))))))..(((((((...(((((.(((((....))))).))))).)))))))...........)).))). ( -39.90) >DroWil_CAF1 10065 103 + 1 UGCUUGUGUUUGGUGUGGU---GGCUUCGGCGCUAGUGGUGCGCGGCACUGAGAUCGGAUCAUUGGGAUCGAUGGCUUGCGUGGUGCCGUAGAAUAUCAAUGACCA ........((..((((.((---(((....))((.....)).))).))))..))...((.(((((((..((.(((((.........))))).))...))))))))). ( -34.30) >DroAna_CAF1 8037 100 + 1 ------AGUUCGGGGUGGUGGCUGCCUCCGGGCCAAUCGGCUUCGGCACCGAGAUGGGGUCGUUGGGGUCGAUGGCGUUCGUGGUGCCGUAGAAGAUCAGCGACCA ------.((((((((..(...)..)).))))))((.(((((....))..)))..)).(((((((((..((.((((((.......)))))).))...))))))))). ( -42.40) >DroPer_CAF1 7930 97 + 1 ------AGUUGGGCGUGGU---GGCCUCUGCACUGUUGGGACGCGGCACCGAAAUCCGAUCAUUGGGAUCGAUGGUCAGGGUGGUGCCGUAGAAGAUCAGCGACCA ------.((.((((.....---.))))..))..((((((...((((((((...((((((((((((....)))))))).))))))))))))......)))))).... ( -38.00) >consensus ______AGUUCGGGGUGGU___GGCCUCCGCACCACUGGGCCGCGGCACCGAGAUCGGAUCAUUGGGAUCGAUGGUCUGAGUGGUGCCGUAGAAGAUCAGCGACCA ...........((.(.(((....))).)................(((((((...(((((((((((....))))))))))).)))))))...............)). (-26.52 = -26.38 + -0.13)
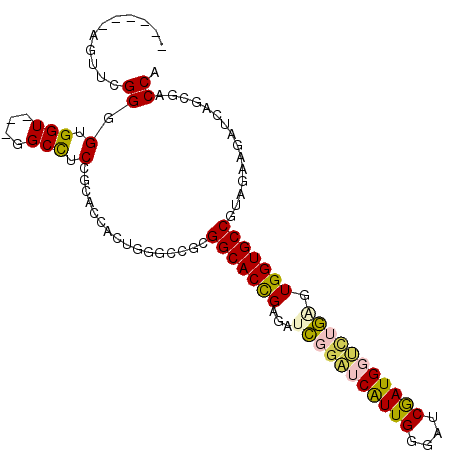
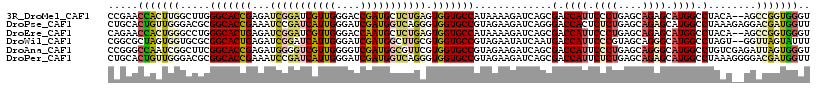
| Location | 21,181,701 – 21,181,816 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 76.90 |
| Mean single sequence MFE | -44.80 |
| Consensus MFE | -34.00 |
| Energy contribution | -34.37 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.44 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.903874 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
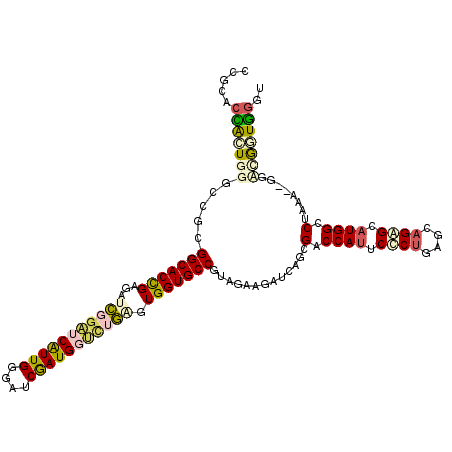
>3R_DroMel_CAF1 21181701 115 + 27905053 CCGAACCACUUGGCUUGGGCACCGAGAUCGGAUCGUUGGGACCGAUGCUCUGAGUGGUGCCAUAAAAGAUCAGCGACCAUUCCCUGAGCAGAGCAUGGCCUACA--AGCCGGUGGGU .....(((((.(((((((((((((...(((((.(((((....))))).))))).)))))))..........((.(.((((.(.((....)).).))))))).))--))))))))).. ( -48.10) >DroPse_CAF1 7899 117 + 1 CUGCACUGUUGGGACGCGGCACCGAAAUCCGAUCAUUGGGAUCGAUGGUCAGGGUGGUGCCGUAGAAGAUCAGGGACCACUCUCUGAGCAGAGCAUGGCCUAAAGAGGACGAUGGUU ((((...........((((((((...((((((((((((....)))))))).))))))))))))......(((((((....)))))))))))..(((.((((....))).).)))... ( -47.10) >DroEre_CAF1 8333 115 + 1 CAGAACCACUGGGCCUGGGCACUGAGAUCGGAUCGUUGGGACCAAUGCUCUGAGUGGUGCCAUAAAAGAUCAGCGACCAUUCCCUGAGCAGAGCAUGGCCUACA--AGCCGGUGGGU .....(((((((((((((((((((...(((((.(((((....))))).))))).)))))))........((((.((....)).)))).))).)).((.....))--..))))))).. ( -44.90) >DroWil_CAF1 10089 115 + 1 CGGCGCUAGUGGUGCGCGGCACUGAGAUCGGAUCAUUGGGAUCGAUGGCUUGCGUGGUGCCGUAGAAUAUCAAUGACCAUUCCCGUAGCAGGGCAUGGCCUAGU--GGUUAGUAUUU ...(((((((((((.(((((((((....(((..(((((....)))))..)))..)))))))))....)))))....((((.(((......))).)))).)))))--).......... ( -38.00) >DroAna_CAF1 8058 117 + 1 CCGGGCCAAUCGGCUUCGGCACCGAGAUGGGGUCGUUGGGGUCGAUGGCGUUCGUGGUGCCGUAGAAGAUCAGCGACCAUUCCCUGAGCAGGGCAUGGCCUGUCGAGAUUAGUGGGU .((((((..(((((....))..))).(((.(((((((((..((.((((((.......)))))).))...)))))))))...((((....)))))))))))))............... ( -46.10) >DroPer_CAF1 7948 117 + 1 CUGCACUGUUGGGACGCGGCACCGAAAUCCGAUCAUUGGGAUCGAUGGUCAGGGUGGUGCCGUAGAAGAUCAGCGACCAUUCUCUGAGCAGAGCAUGGCCUAAAGGGGACGAUGGUU .(((.(((((.(((.((((((((...((((((((((((....)))))))).)))))))))))).(((............)))))).))))).))).((((....(....)...)))) ( -44.60) >consensus CCGCACCACUGGGCCGCGGCACCGAGAUCGGAUCAUUGGGAUCGAUGGUCUGAGUGGUGCCGUAGAAGAUCAGCGACCAUUCCCUGAGCAGAGCAUGGCCUAAA__GGACGGUGGGU .....(((((((.....(((((((...(((((((((((....))))))))))).))))))).............(.((((.((((....)))).)))).)........))))))).. (-34.00 = -34.37 + 0.37)

| Location | 21,181,701 – 21,181,816 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 76.90 |
| Mean single sequence MFE | -38.12 |
| Consensus MFE | -24.71 |
| Energy contribution | -24.49 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.82 |
| SVM RNA-class probability | 0.859097 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
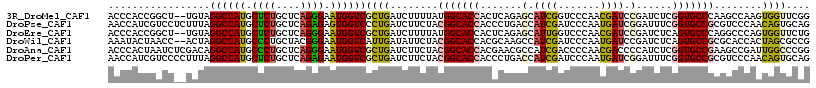
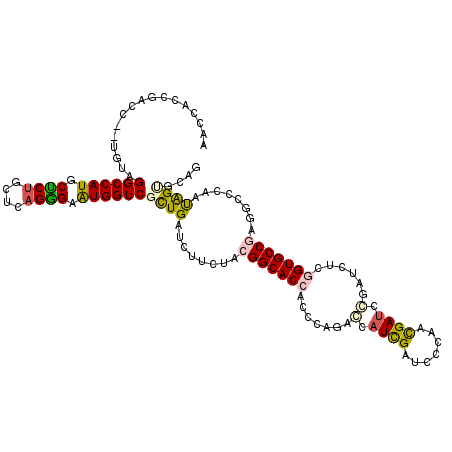
>3R_DroMel_CAF1 21181701 115 - 27905053 ACCCACCGGCU--UGUAGGCCAUGCUCUGCUCAGGGAAUGGUCGCUGAUCUUUUAUGGCACCACUCAGAGCAUCGGUCCCAACGAUCCGAUCUCGGUGCCCAAGCCAAGUGGUUCGG ..((((.((((--((..((((((.((((....)))).)))))).............((((((.....(((.(((((((.....)).))))))))))))))))))))..))))..... ( -46.40) >DroPse_CAF1 7899 117 - 1 AACCAUCGUCCUCUUUAGGCCAUGCUCUGCUCAGAGAGUGGUCCCUGAUCUUCUACGGCACCACCCUGACCAUCGAUCCCAAUGAUCGGAUUUCGGUGCCGCGUCCCAACAGUGCAG .......(..((..(((((((((.((((....)))).))))..))))).......(((((((...........(((((.....)))))(....)))))))).........))..).. ( -32.50) >DroEre_CAF1 8333 115 - 1 ACCCACCGGCU--UGUAGGCCAUGCUCUGCUCAGGGAAUGGUCGCUGAUCUUUUAUGGCACCACUCAGAGCAUUGGUCCCAACGAUCCGAUCUCAGUGCCCAGGCCCAGUGGUUCUG ..((((.((((--((..((((((.((((....)))).)))))).............(((((......(((.(((((((.....)).)))))))).)))))))))))..))))..... ( -40.10) >DroWil_CAF1 10089 115 - 1 AAAUACUAACC--ACUAGGCCAUGCCCUGCUACGGGAAUGGUCAUUGAUAUUCUACGGCACCACGCAAGCCAUCGAUCCCAAUGAUCCGAUCUCAGUGCCGCGCACCACUAGCGCCG ...........--....((((((.(((......))).)))))).............(((((..(....)..(((((((.....))).))))....)))))((((.......)))).. ( -32.80) >DroAna_CAF1 8058 117 - 1 ACCCACUAAUCUCGACAGGCCAUGCCCUGCUCAGGGAAUGGUCGCUGAUCUUCUACGGCACCACGAACGCCAUCGACCCCAACGACCCCAUCUCGGUGCCGAAGCCGAUUGGCCCGG ..((.............((((((.((((....)))).))))))((..(((..((.(((((((.(((......)))........((.......))))))))).))..)))..))..)) ( -42.00) >DroPer_CAF1 7948 117 - 1 AACCAUCGUCCCCUUUAGGCCAUGCUCUGCUCAGAGAAUGGUCGCUGAUCUUCUACGGCACCACCCUGACCAUCGAUCCCAAUGAUCGGAUUUCGGUGCCGCGUCCCAACAGUGCAG .................((((((.((((....)))).))))))((((........(((((((...........(((((.....)))))(....))))))))........)))).... ( -34.89) >consensus AACCACCGACC__UGUAGGCCAUGCUCUGCUCAGGGAAUGGUCGCUGAUCUUCUACGGCACCACCCAGACCAUCGAUCCCAACGAUCCGAUCUCGGUGCCGAGGCCCAAUAGUGCAG .................((((((.((((....)))).))))))((((........(((((((.......(.((((.......)))).)......)))))))........)))).... (-24.71 = -24.49 + -0.22)
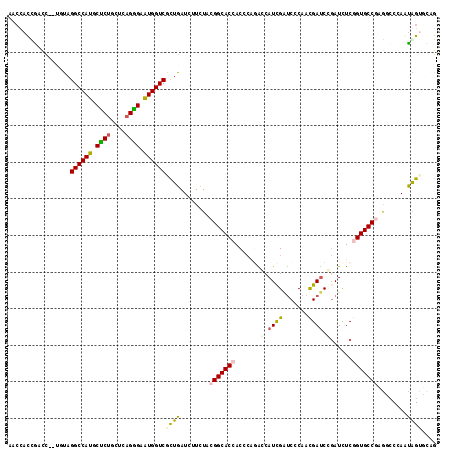
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:57:34 2006