
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 21,072,215 – 21,072,410 |
| Length | 195 |
| Max. P | 0.998624 |

| Location | 21,072,215 – 21,072,335 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.94 |
| Mean single sequence MFE | -48.07 |
| Consensus MFE | -46.99 |
| Energy contribution | -46.63 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.98 |
| SVM decision value | 2.01 |
| SVM RNA-class probability | 0.985414 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
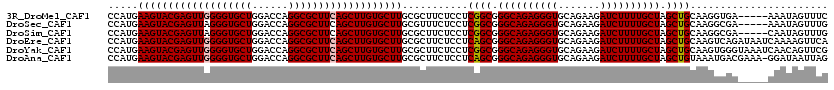
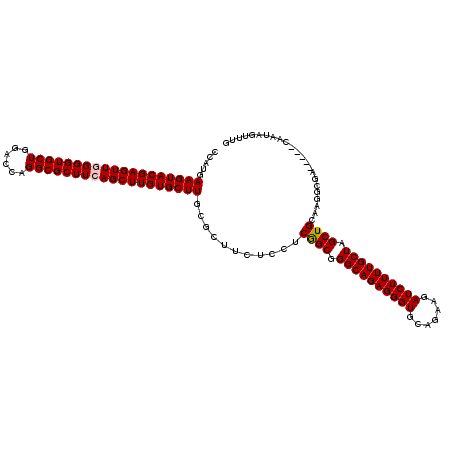
>3R_DroMel_CAF1 21072215 120 - 27905053 CUGAAGCGCCUGGUCCAGCACCCCAACUCGUACUUCAUGGACGUGAAGUGCCCCGGCUGCUACAGGAUCACCACCGUCUUCAGCCACGCCCAAGGCGUCGUGGUCUGCGCUGGAUGCGCU ....(((((...(((((((..........(((((((((....))))))))).(((((((.....((.......)).....)))))(((((...)))))...)).....)))))))))))) ( -47.60) >DroSec_CAF1 25427 120 - 1 CUGAAGCGCCUGGUCCAGCACCCUAACUCGUACUUCAUGGACGUGAAGUGCCCCGGCUGCUACAGGAUCACCACCGUCUUCAGCCACGCCCAGGGCGUCGUGGUCUGCGCCGGAUGCGCA .....(((((((((.(((.((((......(((((((((....)))))))))...(((((.....((.......)).....)))))(((((...))))).).)))))).)))))..)))). ( -47.60) >DroSim_CAF1 24742 120 - 1 CUGAAGCGCCUGGUCCAGCACCCUAACUCGUACUUCAUGGACGUGAAGUGCCCCGGCUGCUACAGGAUCACCACCGUCUUCAGCCACGCCCAGGGCGUCGUGGUCUGCGCCGGAUGCGCA .....(((((((((.(((.((((......(((((((((....)))))))))...(((((.....((.......)).....)))))(((((...))))).).)))))).)))))..)))). ( -47.60) >DroEre_CAF1 26260 120 - 1 CUGAAGCGCCUGGUCCAGCACCCCAACUCGUACUUCAUGGACGUGAAGUGCCCCGGCUGCUACAGGAUCACCACCGUCUUCAGCCACGCCCAGGGCGUCGUGGUCUGCGCCGGAUGCGCA .....(((((((((.(((.(((.(.....(((((((((....)))))))))...(((((.....((.......)).....)))))(((((...))))).).)))))).)))))..)))). ( -48.10) >DroYak_CAF1 25609 120 - 1 CUGAAGCGCCUGGUCCAGCACCCCAACUCGUACUUCAUGGACGUGAAGUGCCCCGGCUGCUACAGGAUCACCACCGUCUUCAGCCACGCCCAGGGCGUCGUGGUCUGCGCCGGAUGCGCA .....(((((((((.(((.(((.(.....(((((((((....)))))))))...(((((.....((.......)).....)))))(((((...))))).).)))))).)))))..)))). ( -48.10) >DroAna_CAF1 27059 120 - 1 CUGAAGCGCCUGGUCCAGCACCCCAACUCGUACUUCAUGGACGUGAAGUGCCCCGGCUGCUACAGGAUCACCACCGUCUUCAGCCACGCCCAGGGCGUUGUGGUCUGCGCCGGUUGCGCC .....(((((((((.(((.(((.((((..(((((((((....)))))))))...(((((.....((.......)).....)))))..(((...))))))).)))))).)))))..)))). ( -49.40) >consensus CUGAAGCGCCUGGUCCAGCACCCCAACUCGUACUUCAUGGACGUGAAGUGCCCCGGCUGCUACAGGAUCACCACCGUCUUCAGCCACGCCCAGGGCGUCGUGGUCUGCGCCGGAUGCGCA .....(((((((((.(((.(((.(.....(((((((((....)))))))))...(((((.....((.......)).....)))))(((((...))))).).)))))).)))))..)))). (-46.99 = -46.63 + -0.36)

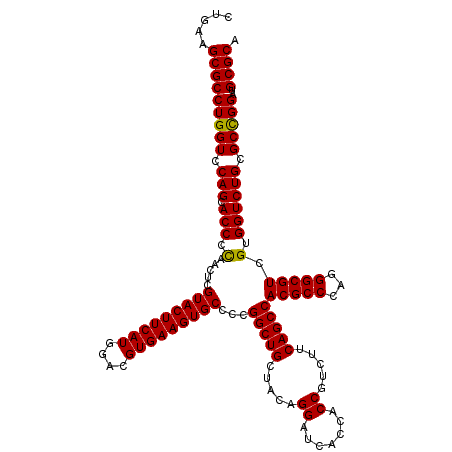
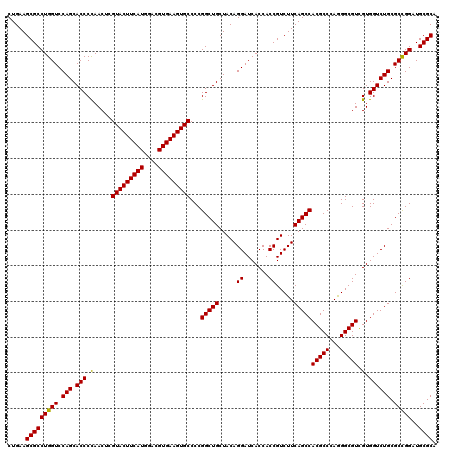
| Location | 21,072,255 – 21,072,375 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.83 |
| Mean single sequence MFE | -52.78 |
| Consensus MFE | -52.10 |
| Energy contribution | -52.30 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.99 |
| SVM decision value | 3.16 |
| SVM RNA-class probability | 0.998624 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 21072255 120 + 27905053 AAGACGGUGGUGAUCCUGUAGCAGCCGGGGCACUUCACGUCCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGA ..((((..((((.(((((.......)))))))))...))))....(((((((((((((((((((......)))))))))))))))))))(((((((((((....))).))).)))))... ( -54.40) >DroSec_CAF1 25467 120 + 1 AAGACGGUGGUGAUCCUGUAGCAGCCGGGGCACUUCACGUCCAUGAAGUACGAGUUAGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGUUUCUCCUCGGCGGGCAGAGGGUGCAGA .........((.(((((.(.((.(((((((......((((.....(((((((((((..((((((......))))))..)))))))))))))))....)))))))..))).)))))))... ( -48.80) >DroSim_CAF1 24782 120 + 1 AAGACGGUGGUGAUCCUGUAGCAGCCGGGGCACUUCACGUCCAUGAAGUACGAGUUAGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGA ..((((..((((.(((((.......)))))))))...))))....(((((((((((..((((((......))))))..)))))))))))(((((((((((....))).))).)))))... ( -50.30) >DroEre_CAF1 26300 120 + 1 AAGACGGUGGUGAUCCUGUAGCAGCCGGGGCACUUCACGUCCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCAGCGGGCAGAGGGUGCAGA ..((((..((((.(((((.......)))))))))...))))....(((((((((((((((((((......)))))))))))))))))))(((((((((((....))).))).)))))... ( -54.40) >DroYak_CAF1 25649 120 + 1 AAGACGGUGGUGAUCCUGUAGCAGCCGGGGCACUUCACGUCCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGA ..((((..((((.(((((.......)))))))))...))))....(((((((((((((((((((......)))))))))))))))))))(((((((((((....))).))).)))))... ( -54.40) >DroAna_CAF1 27099 120 + 1 AAGACGGUGGUGAUCCUGUAGCAGCCGGGGCACUUCACGUCCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCAGCGGGCAGAGGGUGCAGA ..((((..((((.(((((.......)))))))))...))))....(((((((((((((((((((......)))))))))))))))))))(((((((((((....))).))).)))))... ( -54.40) >consensus AAGACGGUGGUGAUCCUGUAGCAGCCGGGGCACUUCACGUCCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGA ..((((..((((.(((((.......)))))))))...))))....(((((((((((((((((((......)))))))))))))))))))(((((((((((....))).))).)))))... (-52.10 = -52.30 + 0.20)

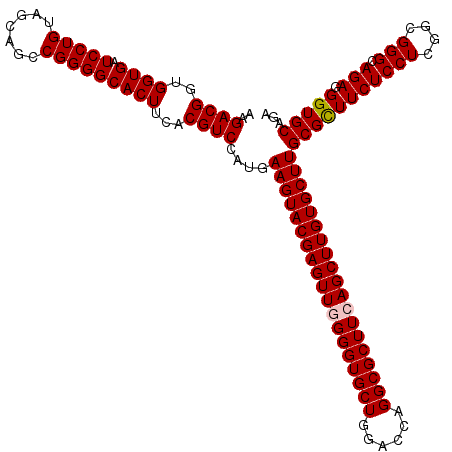
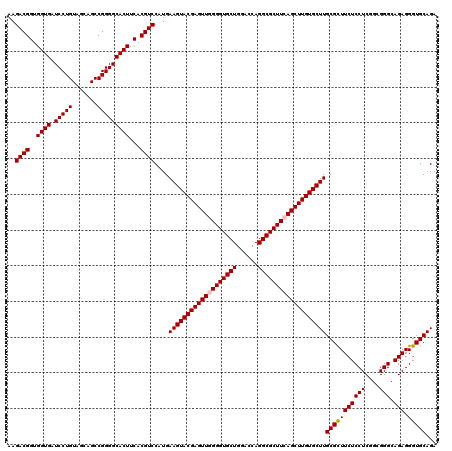
| Location | 21,072,295 – 21,072,410 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.52 |
| Mean single sequence MFE | -47.60 |
| Consensus MFE | -43.35 |
| Energy contribution | -43.47 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.89 |
| SVM RNA-class probability | 0.997608 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 21072295 115 + 27905053 CCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGAAGAUCUUUUGCUAGCUGCAAGGUGA-----AAAUAGUUUC .....(((((((((((((((((((......)))))))))))))))))))....(((.(((((((.((((((((((.......)))))))))).))))..))).))-----)......... ( -50.00) >DroSec_CAF1 25507 115 + 1 CCAUGAAGUACGAGUUAGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGUUUCUCCUCGGCGGGCAGAGGGUGCAGAAGAUCUUUUGCUAGCUGCAAGGCGA-----AAAUAGUUUG .....(((((((((((..((((((......))))))..)))))))))))((.((((.(((((((.((((((((((.......)))))))))).))))..))).))-----))...))... ( -47.20) >DroSim_CAF1 24822 115 + 1 CCAUGAAGUACGAGUUAGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGAAGAUCUUUUGCUAGCUGCAAGGCGA-----CAAUAGUUUG .....(((((((((((..((((((......))))))..))))))))))).(((((.....((((.((((((((((.......)))))))))).))))..))))).-----.......... ( -45.20) >DroEre_CAF1 26340 120 + 1 CCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCAGCGGGCAGAGGGUGCAGAAGAUCUUUUGCUAGCUGCAAGUCAGAUAAUCAAAAGUUCA .....(((((((((((((((((((......)))))))))))))))))))(((((((((((....))).))).))))).((..(((((((((.....)))))..))))..))......... ( -46.90) >DroYak_CAF1 25689 120 + 1 CCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGAAGAUCUUUUGCUAGCUGCAAGUGGGUAAAUCAACAGUUCG ((...(((((((((((((((((((......))))))))))))))))))).(((((.....((((.((((((((((.......)))))))))).)))).)))))))............... ( -48.60) >DroAna_CAF1 27139 119 + 1 CCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCAGCGGGCAGAGGGUGCAGAAGAUCUUUUGCUAGCUGUAAAUGACGAAA-GGAUAAUUAG ((...(((((((((((((((((((......)))))))))))))))))))....(((((..((((.((((((((((.......)))))))))).)))).....)).))).-))........ ( -47.70) >consensus CCAUGAAGUACGAGUUGGGGUGCUGGACCAGGCGCUUCAGCUUGUGCUUGCGCUUCUCCUCGGCGGGCAGAGGGUGCAGAAGAUCUUUUGCUAGCUGCAAGGCGA_____CAAUAGUUUG .....(((((((((((((((((((......)))))))))))))))))))...........((((.((((((((((.......)))))))))).))))....................... (-43.35 = -43.47 + 0.11)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:56:49 2006