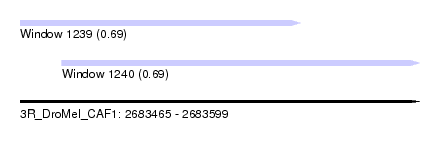
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 2,683,465 – 2,683,599 |
| Length | 134 |
| Max. P | 0.693512 |

| Location | 2,683,465 – 2,683,559 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.05 |
| Mean single sequence MFE | -35.07 |
| Consensus MFE | -25.68 |
| Energy contribution | -26.52 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.16 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.693512 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
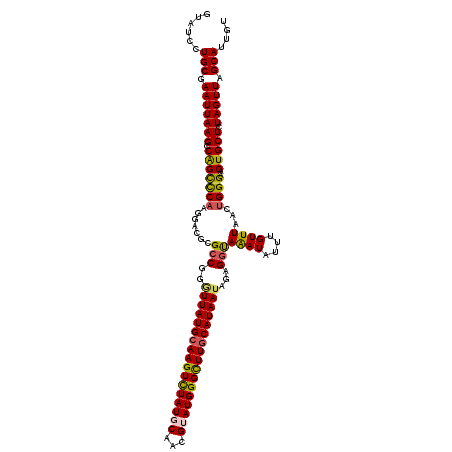
>3R_DroMel_CAF1 2683465 94 + 27905053 --CUCGGGAUA------------------------AGCGCGUAUCCUGCGAAUUAAGCCAGCCCAAGGACGAGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGUUUGCAUAAUAGAGGU --(.(((((((------------------------......))))))).)......(((.((((..((.....))))))(((((((..((((((...))))))..))))))).....))) ( -31.20) >DroSec_CAF1 42959 94 + 1 --CUCGGGAUA------------------------AGCGCGUAUCCUGCGAAUUAAGCCAGUCCAAGGACGCGCCGGAUUAUGCAAGUUUAUGCAACGUAUGGGUUUGCAUAAUAGAGGU --(.(((((((------------------------......))))))).)......((..(((....)))))(((..((((((((((..(((((...)))))..))))))))))...))) ( -31.30) >DroSim_CAF1 36123 94 + 1 --CUCGGGAUA------------------------AGCGCGUAUCCUGCGAAUUAAGCCAGCCCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGUUUACAUAAUAGAGGU --(((......------------------------.(((((..((((((...........))...)))))))))...((((((.((..((((((...))))))..)).)))))).))).. ( -26.80) >DroEre_CAF1 36858 94 + 1 --CUCGGGAUC------------------------UGCGCGUAUCCUGCGAAUUAAGCCAGCCCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGAAUGGGCUUGCAUAAUAGAGGU --(((.....(------------------------((.(((..((((((...........))...))))..))))))(((((((((((((((.......))))))))))))))).))).. ( -37.30) >DroYak_CAF1 43476 94 + 1 --CUCGGGAUC------------------------UGUGCCUAUCCUGCGAAUUAAGACAGCUCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGCUUGCAUAAAUGAGGU --(((((....------------------------(((((((...((((.......).)))....))).)))))))))((((((((((((((((...))))))))))))))))....... ( -33.20) >DroAna_CAF1 30111 120 + 1 GGCUCGGGAUCGGGCUCGGGCUCUGGUUCUCGGCCAGGACAUAUCCUGCGAAUUAAGCCGGUCCAAGGACGCGCCCGGUUAUGCAAGUCUAUGCAACGGAUGGGCUUGCAUAAUAAAGAC ((((((....))))))(((((.(.......(((((((((....)))))........))))(((....)))).)))))(((((((((((((((.(...).)))))))))))))))...... ( -50.60) >consensus __CUCGGGAUA________________________AGCGCGUAUCCUGCGAAUUAAGCCAGCCCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGCUUGCAUAAUAGAGGU ..(((...............................(((((..((((((...........))...)))))))))...(((((((((((((((((...))))))))))))))))).))).. (-25.68 = -26.52 + 0.84)

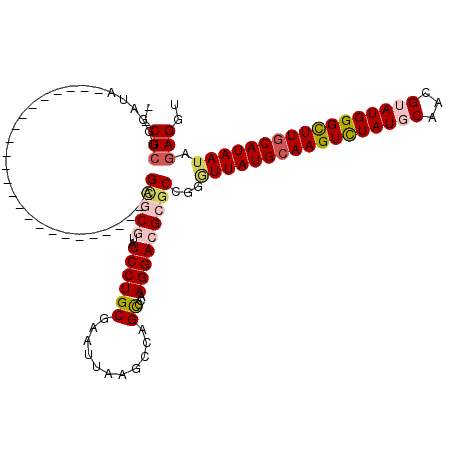
| Location | 2,683,479 – 2,683,599 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.39 |
| Mean single sequence MFE | -36.90 |
| Consensus MFE | -30.76 |
| Energy contribution | -30.48 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.33 |
| SVM RNA-class probability | 0.693365 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 2683479 120 + 27905053 GUAUCCUGCGAAUUAAGCCAGCCCAAGGACGAGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGUUUGCAUAAUAGAGGUAAAUGUUUGUUUAACUGGGAACUGCUCUAGUUAGCAUUGU ......(((.((((((((...((((.(((((((((..(((((((((..((((((...))))))..)))))))))...)))......))))))...))))....))).))))).))).... ( -38.00) >DroSec_CAF1 42973 120 + 1 GUAUCCUGCGAAUUAAGCCAGUCCAAGGACGCGCCGGAUUAUGCAAGUUUAUGCAACGUAUGGGUUUGCAUAAUAGAGGUAAAUGUUUGUUUAACUGGGAACUGCUCUAGUUAGCAUUGU ......(((....(((((..(((....)))..(((..((((((((((..(((((...)))))..))))))))))...)))....)))))..((((((((......))))))))))).... ( -35.00) >DroSim_CAF1 36137 120 + 1 GUAUCCUGCGAAUUAAGCCAGCCCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGUUUACAUAAUAGAGGUAAAUGUUUGUUUAACUGGGAACUGCUCUAGUUAGCAUUGU (..((((((...........))...))))..)(((..((((((.((..((((((...))))))..)).))))))...))).(((((.....((((((((......))))))))))))).. ( -30.60) >DroEre_CAF1 36872 120 + 1 GUAUCCUGCGAAUUAAGCCAGCCCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGAAUGGGCUUGCAUAAUAGAGGUAAAUAUUUGUUUAACUGGGAACUGCUCUAGUUAGCAUUGU (..((((((...........))...))))..)(((..(((((((((((((((.......)))))))))))))))...))).......((((.(((((((......))))))))))).... ( -38.50) >DroYak_CAF1 43490 120 + 1 CUAUCCUGCGAAUUAAGACAGCUCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGCUUGCAUAAAUGAGGUAGAUAUUUGUUUAACUGGGAACUGCUCUAGUUAGCAUUGU ...((((((...........))...)))).(((((...((((((((((((((((...))))))))))))))))....)))...........((((((((......))))))))))..... ( -37.80) >DroAna_CAF1 30151 120 + 1 AUAUCCUGCGAAUUAAGCCGGUCCAAGGACGCGCCCGGUUAUGCAAGUCUAUGCAACGGAUGGGCUUGCAUAAUAAAGACAAAUAUUUGUUUAACGGGGAACUGCUCUAGUUAGCAUUGU ......(((.(((((((((((((....))).))(((((((((((((((((((.(...).)))))))))))))))..((((((....))))))..)))).....))).))))).))).... ( -41.50) >consensus GUAUCCUGCGAAUUAAGCCAGCCCAAGGACGCGCCGGGUUAUGCAAGUCUAUGCAACGUAUGGGCUUGCAUAAUAGAGGUAAAUAUUUGUUUAACUGGGAACUGCUCUAGUUAGCAUUGU ......(((.(((((((.(((((((.......(((..(((((((((((((((((...)))))))))))))))))...)))((((....))))...))))..))))).))))).))).... (-30.76 = -30.48 + -0.27)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:51:53 2006