
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 20,701,137 – 20,701,294 |
| Length | 157 |
| Max. P | 0.972098 |

| Location | 20,701,137 – 20,701,254 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.37 |
| Mean single sequence MFE | -56.70 |
| Consensus MFE | -36.94 |
| Energy contribution | -37.83 |
| Covariance contribution | 0.90 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.24 |
| SVM RNA-class probability | 0.934744 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20701137 117 + 27905053 AGGCCGUGGGGCGGAGUGCCGCUGGUGGGUGAGCUGAGGUGGUUGGUGAUCACAGAGGCGGCCGCCACCACCACCUCUGUGGUUGUAGCCGUGGCCGCCGCAGCAGCUGCCUGUAUG ..((((.(.(((.....))).)))))((((.(((((.(((((((((((((((((((((.((.........)).)))))))))))...))))..))))))....)))))))))..... ( -55.20) >DroPse_CAF1 2696 117 + 1 AGACCAUGGGGUGGCGUUCCGCUGGUGGGGGAGCUCAGGUGAUUUGUGAUCACUGAGGCGGCAGCCACGACCACCUCGGUUGUGGUCGCGGUGGCAGCCGCAGCAGCCGCUUGUAUC ........((((((((((((.(.....).)))))...(((((((...)))))))(..(((((.(((((((((((.......))))))...))))).)))))..).)))))))..... ( -53.10) >DroWil_CAF1 2859 106 + 1 AGACUAUGCGGUGGUGUGCCACUAGUGGGUGAGCUCAGGUGAUUGGUUAUCACCGAGGCGGCAGCCACCACCACUUCGGUAGUGGUUGCAGUGGCAGCAGCUGCAG----------- ......((((((.((.(((((((.........(((..((((((.....))))))..))).((((((((.(((.....))).))))))))))))))))).)))))).----------- ( -52.70) >DroYak_CAF1 3005 117 + 1 AGGCCAUGUGGCGGAGUGCCGCUGGUGGGUGAGCUGAGGUGGUUGGUGAUCACAGAGGCGGCCGCCACCACCACCUCGGUGGUUGUGGCCGUGGCCGCCGCAGCUGCAGCCUGAAUG ..((((.(((((.....)))))))))((.(((((((.(((((((.((....))....((((((((.((((((.....)))))).))))))))))))))).))))).)).))...... ( -65.00) >DroAna_CAF1 2633 117 + 1 AGUCCAUGCGGCGGAGUGCCACUUGUCGGCGAGCUAAGGUGAUUCGUGAUCACCGAGGCAGCAGCUGCAACCACCUCCGUGGUAGUGGCUGUUGCCGCAGCCGCUGCAGCCGUUAUG ....((((((((((....))...((.(((((.(((..(((((((...)))))))(.(((((((((..(.(((((....))))).)..))))))))).))))))))))))))).)))) ( -61.10) >DroPer_CAF1 2706 117 + 1 AGACCAUGGGGUGGCGUUCCGCUGGUGGGGGAGCUCAGGUGAUUUGUGAUCACUGAGGCGGCAGCCACGACCACCUCGGUUGUGGUCGCGGUGGCAGCCGCAGCAGCCGCUUGUAUC ........((((((((((((.(.....).)))))...(((((((...)))))))(..(((((.(((((((((((.......))))))...))))).)))))..).)))))))..... ( -53.10) >consensus AGACCAUGGGGCGGAGUGCCGCUGGUGGGUGAGCUCAGGUGAUUGGUGAUCACAGAGGCGGCAGCCACCACCACCUCGGUGGUGGUCGCCGUGGCAGCCGCAGCAGCAGCCUGUAUG ...((((..(((((....))))).))))....(((..((((((.....))))))..))).((.(((((((((.....))))))))).)).(((((.((....)).)))))....... (-36.94 = -37.83 + 0.90)



| Location | 20,701,137 – 20,701,254 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 79.37 |
| Mean single sequence MFE | -49.36 |
| Consensus MFE | -29.92 |
| Energy contribution | -31.25 |
| Covariance contribution | 1.33 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.92 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.972098 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20701137 117 - 27905053 CAUACAGGCAGCUGCUGCGGCGGCCACGGCUACAACCACAGAGGUGGUGGUGGCGGCCGCCUCUGUGAUCACCAACCACCUCAGCUCACCCACCAGCGGCACUCCGCCCCACGGCCU .....((((.(((((((.(((((((...(((((.(((((....))))).))))))))))))...((((.(.............).))))....)))))))....((.....)))))) ( -49.42) >DroPse_CAF1 2696 117 - 1 GAUACAAGCGGCUGCUGCGGCUGCCACCGCGACCACAACCGAGGUGGUCGUGGCUGCCGCCUCAGUGAUCACAAAUCACCUGAGCUCCCCCACCAGCGGAACGCCACCCCAUGGUCU .......(((.((((((((((.(((...((((((((.......))))))))))).)))))(((((((((.....)))).)))))..........)))))..))).(((....))).. ( -47.10) >DroWil_CAF1 2859 106 - 1 -----------CUGCAGCUGCUGCCACUGCAACCACUACCGAAGUGGUGGUGGCUGCCGCCUCGGUGAUAACCAAUCACCUGAGCUCACCCACUAGUGGCACACCACCGCAUAGUCU -----------..(((((..(..(((((..............)))))..)..))))).((.((((((((.....)))))).))))......((((((((.......)))).)))).. ( -41.24) >DroYak_CAF1 3005 117 - 1 CAUUCAGGCUGCAGCUGCGGCGGCCACGGCCACAACCACCGAGGUGGUGGUGGCGGCCGCCUCUGUGAUCACCAACCACCUCAGCUCACCCACCAGCGGCACUCCGCCACAUGGCCU ......((.((.(((((.(((((((...(((((.(((((....))))).))))))))))))...(((.........)))..))))))).)).((((.(((.....))).).)))... ( -52.60) >DroAna_CAF1 2633 117 - 1 CAUAACGGCUGCAGCGGCUGCGGCAACAGCCACUACCACGGAGGUGGUUGCAGCUGCUGCCUCGGUGAUCACGAAUCACCUUAGCUCGCCGACAAGUGGCACUCCGCCGCAUGGACU .....((((.((((((((((((((....)))((((((.....)))))).)))))))))))...((((((.....)))))).......)))).((.(((((.....))))).)).... ( -58.70) >DroPer_CAF1 2706 117 - 1 GAUACAAGCGGCUGCUGCGGCUGCCACCGCGACCACAACCGAGGUGGUCGUGGCUGCCGCCUCAGUGAUCACAAAUCACCUGAGCUCCCCCACCAGCGGAACGCCACCCCAUGGUCU .......(((.((((((((((.(((...((((((((.......))))))))))).)))))(((((((((.....)))).)))))..........)))))..))).(((....))).. ( -47.10) >consensus CAUACAGGCGGCUGCUGCGGCGGCCACCGCCACCACCACCGAGGUGGUGGUGGCUGCCGCCUCAGUGAUCACCAAUCACCUGAGCUCACCCACCAGCGGCACUCCACCCCAUGGUCU .............((.(((((.(((...(((((((((.....)))))))))))).)))))....(((((.....)))))....))..........((((....)))).......... (-29.92 = -31.25 + 1.33)

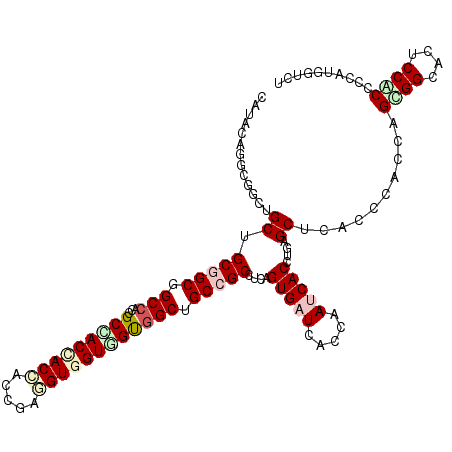

| Location | 20,701,174 – 20,701,294 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.83 |
| Mean single sequence MFE | -54.15 |
| Consensus MFE | -32.03 |
| Energy contribution | -31.87 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.610033 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20701174 120 + 27905053 GGUGGUUGGUGAUCACAGAGGCGGCCGCCACCACCACCUCUGUGGUUGUAGCCGUGGCCGCCGCAGCAGCUGCCUGUAUGGGUGAGGUGAUGGAGUUCAGUGCACUGCCGAUGGAGGAGU (((((((((((((((((((((.((.........)).)))))))))))...))))..))))))......(((.(((.(((.(((..((((.((.....))...))))))).))).)))))) ( -49.60) >DroVir_CAF1 2883 120 + 1 AAUGAUUUGUUAUGGCCGAUGCAGCGGCUACCACCACCUCCGUGGUGGUGGCUGUUGCCGCCGCCGCUGCGGCCGUUAUGGGCGAUGUUAGGGAAUUGAUAGCGCUGCCUAUUGAGGAGU ....((((.((((((((...((((((((.((((((((....))))))))(((....)))...)))))))))))))..(((((((.(((((.........))))).)))))))))).)))) ( -56.00) >DroPse_CAF1 2733 120 + 1 GGUGAUUUGUGAUCACUGAGGCGGCAGCCACGACCACCUCGGUUGUGGUCGCGGUGGCAGCCGCAGCAGCCGCUUGUAUCGGCGAAGUUAUUGAGUUGAGGGCGCUGCCGAUCGAGGAGU (((((((...)))))))..(((.((.(((((((((.....))))))))).(((((....))))).)).))).((((.(((((((..((..((.....))..))..))))))))))).... ( -53.40) >DroYak_CAF1 3042 120 + 1 GGUGGUUGGUGAUCACAGAGGCGGCCGCCACCACCACCUCGGUGGUUGUGGCCGUGGCCGCCGCAGCUGCAGCCUGAAUGGGUGAGGUGAUGGAGUUGAGGGCACUGCCGAUGGAGGAGU ((((((.((((..(.(....).)..)))))))))).((((((..((...(((....)))(((.(((((.((((((.(.....).))))..)).)))))..)))))..)))).))...... ( -54.50) >DroAna_CAF1 2670 120 + 1 GGUGAUUCGUGAUCACCGAGGCAGCAGCUGCAACCACCUCCGUGGUAGUGGCUGUUGCCGCAGCCGCUGCAGCCGUUAUGGGGGAUGUGAUCGAGUUGAGGGCGCUGCCAAUGGAGGAGU (((((((...)))))))..(((((((((..(.(((((....))))).)..)))))))))(((((.(((.(((((((((((.....))))).)).))))..))))))))............ ( -56.50) >DroPer_CAF1 2743 120 + 1 GGUGAUUUGUGAUCACUGAGGCGGCAGCCACGACCACCUCGGUUGUGGUCGCGGUGGCAGCCGCAGCAGCCGCUUGUAUCGGCGAAGUUAUCGAGUUGAGGGCGCUGCCGAUCGAGGAGU (((((((...)))))))..(((.((.(((((((((.....))))))))).(((((....))))).)).))).((((.(((((((..((..((.....))..))..))))))))))).... ( -54.90) >consensus GGUGAUUUGUGAUCACAGAGGCGGCAGCCACCACCACCUCGGUGGUGGUGGCCGUGGCCGCCGCAGCAGCAGCCUGUAUGGGCGAAGUGAUGGAGUUGAGGGCGCUGCCGAUGGAGGAGU .(((((.....)))))...((((((.((((((((.......)))))))).)))(((((.((....)).))))).......((((...(((......)))...)))))))........... (-32.03 = -31.87 + -0.16)



| Location | 20,701,174 – 20,701,294 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.83 |
| Mean single sequence MFE | -46.28 |
| Consensus MFE | -29.98 |
| Energy contribution | -30.78 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.34 |
| Mean z-score | -2.85 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.936915 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20701174 120 - 27905053 ACUCCUCCAUCGGCAGUGCACUGAACUCCAUCACCUCACCCAUACAGGCAGCUGCUGCGGCGGCCACGGCUACAACCACAGAGGUGGUGGUGGCGGCCGCCUCUGUGAUCACCAACCACC ..........(((((((((.(((.....................)))))).)))))).(((((((...(((((.(((((....))))).))))))))))))...(((.........))). ( -43.80) >DroVir_CAF1 2883 120 - 1 ACUCCUCAAUAGGCAGCGCUAUCAAUUCCCUAACAUCGCCCAUAACGGCCGCAGCGGCGGCGGCAACAGCCACCACCACGGAGGUGGUGGUAGCCGCUGCAUCGGCCAUAACAAAUCAUU ...........(((.......................)))......(((((..(((((((((....).(((((((((.....))))))))).))))))))..)))))............. ( -49.80) >DroPse_CAF1 2733 120 - 1 ACUCCUCGAUCGGCAGCGCCCUCAACUCAAUAACUUCGCCGAUACAAGCGGCUGCUGCGGCUGCCACCGCGACCACAACCGAGGUGGUCGUGGCUGCCGCCUCAGUGAUCACAAAUCACC .......(((((((((((((((....((............))....)).))).)))))(((.((..((((((((((.......))))))))))..)).)))....))))).......... ( -45.60) >DroYak_CAF1 3042 120 - 1 ACUCCUCCAUCGGCAGUGCCCUCAACUCCAUCACCUCACCCAUUCAGGCUGCAGCUGCGGCGGCCACGGCCACAACCACCGAGGUGGUGGUGGCGGCCGCCUCUGUGAUCACCAACCACC .....((((..(((.(.(((.(((...((((((((((..........(((((....)))))(((....))).........))))))))))))).)))))))..)).))............ ( -44.70) >DroAna_CAF1 2670 120 - 1 ACUCCUCCAUUGGCAGCGCCCUCAACUCGAUCACAUCCCCCAUAACGGCUGCAGCGGCUGCGGCAACAGCCACUACCACGGAGGUGGUUGCAGCUGCUGCCUCGGUGAUCACGAAUCACC ...........(((...)))........((((((.....((.....))..((((((((((((((....)))((((((.....)))))).)))))))))))....)))))).......... ( -45.60) >DroPer_CAF1 2743 120 - 1 ACUCCUCGAUCGGCAGCGCCCUCAACUCGAUAACUUCGCCGAUACAAGCGGCUGCUGCGGCUGCCACCGCGACCACAACCGAGGUGGUCGUGGCUGCCGCCUCAGUGAUCACAAAUCACC .......(((((((((((((((....(((..........)))....)).))).)))))(((.((..((((((((((.......))))))))))..)).)))....))))).......... ( -48.20) >consensus ACUCCUCCAUCGGCAGCGCCCUCAACUCCAUAACAUCGCCCAUACAGGCGGCAGCUGCGGCGGCCACAGCCACCACCACCGAGGUGGUGGUGGCUGCCGCCUCAGUGAUCACAAAUCACC ...........(((.(((((...........................(((((((...)))))))....(((((((((.....)))))))))))).)).)))...(((((.....))))). (-29.98 = -30.78 + 0.81)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:52:36 2006