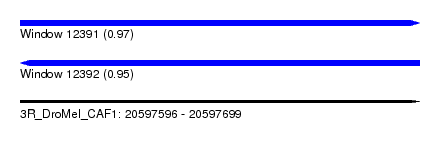
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 20,597,596 – 20,597,699 |
| Length | 103 |
| Max. P | 0.972787 |

| Location | 20,597,596 – 20,597,699 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 79.83 |
| Mean single sequence MFE | -29.53 |
| Consensus MFE | -21.32 |
| Energy contribution | -22.04 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.35 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972787 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
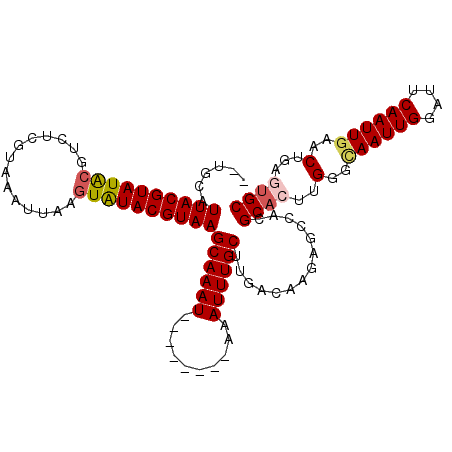
>3R_DroMel_CAF1 20597596 103 + 27905053 GCUCACAGUUCAAUUGAAUCCAAUUGACCAAGUGCGUGGCUCUUGUCAAGCAAAUAC--------AUUUGCUUACGUAUACUUAAUUUACGUGGCGUAUACGUAAUGCAAC .......(.(((((((....))))))).)...(((.((((....)))).))).....--------..((((((((((((((..............)))))))))).)))). ( -27.94) >DroVir_CAF1 25412 101 + 1 GCACUCAGUUUAAUUGAAUCCAAUUGCCCAUGUGCGUGGCUAUUGUCAUGCAAAUUC--------AUUUGCUUACGUAUACUUAAUUUAAGAUAUGUAUACGUAACACA-- (((....(..((((((....))))))..)...((((((((....)))))))).....--------...)))((((((((((..............))))))))))....-- ( -26.94) >DroPse_CAF1 23805 111 + 1 GCAUACAGUUCAAUUGAAGCCAAUUGUCCAAGUGCGUGGCUCUUGUCAAGCAAAUUUAUUUAUUUAUUUGCUUACGUAUACUUAAUGUACGGGCCGUAUACGUAAUGCAAC ((((((.(..((((((....))))))..)..(((..((((((..((.((((((((..........))))))))))(((((.....)))))))))))..))))).))))... ( -34.10) >DroGri_CAF1 27587 99 + 1 GCACUCAGUUCAAUUGAAUUCAAUUACCCAAGUGCGUGGGUCUG-UCAGGCAAAUGA--------AUUUGCUUACGUAUACUUUAUUUACGAGACGUAUACGUAAAGC--- (((...((((((.(((....((...(((((......))))).))-.....))).)))--------))))))((((((((((.((........)).))))))))))...--- ( -24.30) >DroMoj_CAF1 25136 100 + 1 GCACUCGGUUCAAUUGAAUUCAAUUACCCAAGUGCGUGGCUAUGGCCA-GCAAAUUA--------AUUUGCUUACGUACAUUUCAUUUAAGAGAUCCAUACGUAACACG-- (((((.((...(((((....)))))..)).))))).((((....))))-(((((...--------.)))))(((((((.(((((......)))))...)))))))....-- ( -28.90) >DroPer_CAF1 23774 111 + 1 GCAUACAGUUCAAUUGAAACCAAUUGUCCAAGUGCGUGGCUCUUGUCAAGCAAAUUUAUUUAUUUAUUUGCUUACGUAUACUUAAUGUACGAGCCGUAUACGUAAUGCAAC ((((((.(..((((((....))))))..)..(((..((((((..((.((((((((..........))))))))))(((((.....)))))))))))..))))).))))... ( -35.00) >consensus GCACACAGUUCAAUUGAAUCCAAUUGCCCAAGUGCGUGGCUCUUGUCAAGCAAAUUA________AUUUGCUUACGUAUACUUAAUUUACGAGACGUAUACGUAAUGCA__ ((((......((((((....)))))).....)))).((((....)))).((((((..........))))))((((((((((..............))))))))))...... (-21.32 = -22.04 + 0.72)

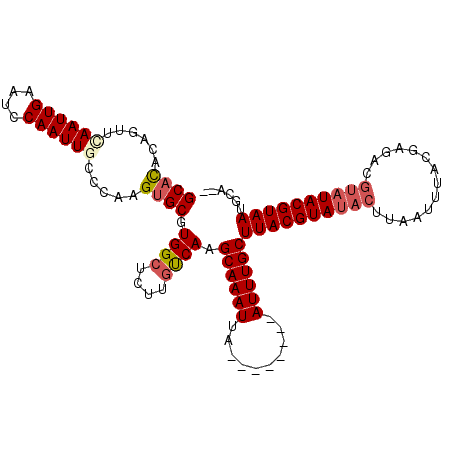
| Location | 20,597,596 – 20,597,699 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 79.83 |
| Mean single sequence MFE | -27.74 |
| Consensus MFE | -19.42 |
| Energy contribution | -19.81 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.42 |
| SVM RNA-class probability | 0.951804 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20597596 103 - 27905053 GUUGCAUUACGUAUACGCCACGUAAAUUAAGUAUACGUAAGCAAAU--------GUAUUUGCUUGACAAGAGCCACGCACUUGGUCAAUUGGAUUCAAUUGAACUGUGAGC .((((.((((((((((..............))))))))))))))..--------......((((..((((.((...)).)))).(((((((....))))))).....)))) ( -27.84) >DroVir_CAF1 25412 101 - 1 --UGUGUUACGUAUACAUAUCUUAAAUUAAGUAUACGUAAGCAAAU--------GAAUUUGCAUGACAAUAGCCACGCACAUGGGCAAUUGGAUUCAAUUAAACUGAGUGC --.(((((((((((((.((........)).))))))))))(((((.--------...)))))...........)))((((.(((..(((((....)))))...))).)))) ( -24.70) >DroPse_CAF1 23805 111 - 1 GUUGCAUUACGUAUACGGCCCGUACAUUAAGUAUACGUAAGCAAAUAAAUAAAUAAAUUUGCUUGACAAGAGCCACGCACUUGGACAAUUGGCUUCAAUUGAACUGUAUGC .((((.((((((((((..............))))))))))))))................((((.....))))...(((..(((.((((((....))))))..)))..))) ( -24.74) >DroGri_CAF1 27587 99 - 1 ---GCUUUACGUAUACGUCUCGUAAAUAAAGUAUACGUAAGCAAAU--------UCAUUUGCCUGA-CAGACCCACGCACUUGGGUAAUUGAAUUCAAUUGAACUGAGUGC ---...((((((((((..............))))))))))(((((.--------...)))))....-.........(((((..(.((((((....))))))..)..))))) ( -27.34) >DroMoj_CAF1 25136 100 - 1 --CGUGUUACGUAUGGAUCUCUUAAAUGAAAUGUACGUAAGCAAAU--------UAAUUUGC-UGGCCAUAGCCACGCACUUGGGUAAUUGAAUUCAAUUGAACCGAGUGC --....(((((((((..((........))..)))))))))(((((.--------...)))))-((((....)))).((((((((.((((((....))))))..)))))))) ( -33.00) >DroPer_CAF1 23774 111 - 1 GUUGCAUUACGUAUACGGCUCGUACAUUAAGUAUACGUAAGCAAAUAAAUAAAUAAAUUUGCUUGACAAGAGCCACGCACUUGGACAAUUGGUUUCAAUUGAACUGUAUGC ..........(((((((((((((((.....))))..((((((((((..........)))))))).))..)))))........(..((((((....))))))..).)))))) ( -28.80) >consensus __UGCAUUACGUAUACGUCUCGUAAAUUAAGUAUACGUAAGCAAAU________AAAUUUGCUUGACAAGAGCCACGCACUUGGGCAAUUGGAUUCAAUUGAACUGAGUGC ......((((((((((..............))))))))))((((((..........))))))..............((((..(..((((((....))))))..)...)))) (-19.42 = -19.81 + 0.39)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:51:53 2006