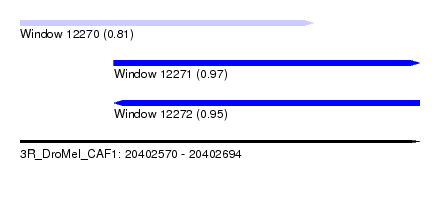
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 20,402,570 – 20,402,694 |
| Length | 124 |
| Max. P | 0.967923 |

| Location | 20,402,570 – 20,402,661 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 76.12 |
| Mean single sequence MFE | -25.67 |
| Consensus MFE | -11.07 |
| Energy contribution | -11.88 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.43 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.813746 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
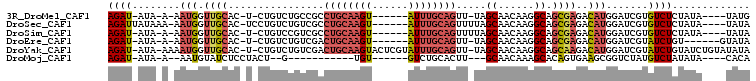
>3R_DroMel_CAF1 20402570 91 + 27905053 AGAU-AUA-A-AAUGGUUGCAC-U-CUGUCUGCCGCCUGCAAGU------AUUUGCAGUU-UAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA----UAUG ..((-(((-.-....(((((.(-(-.(((.(((...((((((..------..))))))..-..)))))))))))))((((((((...))))))))..))----))). ( -29.20) >DroSec_CAF1 25175 95 + 1 AGAUUAUAAA-AAUGGUUGCAC-UCCUGUCUGUCGCCUGCAAGU------AUUUGCAGUUUUAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA----UAUA ..........-.((((..((((-((((((((((.((((((((..------..)))).(((.....))).)))).)).))))).)))..))))..)))).----.... ( -26.40) >DroSim_CAF1 28167 92 + 1 AGAU-AUA-A-AAUGGUUGCAC-U-CUGUCCGUCGCCUGCAAGU------AUUUGCAGUUUUAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA----UAUA ..((-(((-.-....(((((.(-(-.(((..((...((((((..------..)))))).....)).))))))))))((((((((...))))))))..))----))). ( -25.30) >DroEre_CAF1 24692 89 + 1 AGAU-AUA-A-AAUGGUUGCAC-U-CUGUCUGUCGACUGCAAGU------AUUUGCAGUU-UAGCAACAAGGCAGCGAGACAUGGAUCGUAUCUGU------GUAUA ....-...-.-....(((((.(-(-.(((.(((.((((((((..------..))))))))-..)))))))))))))...((((((((...))))))------))... ( -24.70) >DroYak_CAF1 28887 102 + 1 AGAU-AUA-AAAAUGGUUGCAC-U-CUGUCUGUCGACUGCAAGUACUCGUAUUUGCAGUU-UAGCAACAAGGCAGCAAGACAUGGAUCGUAUCUGUAUCUGUAUAUA ..((-(((-......(((((.(-(-.(((.(((.(((((((((((....)))))))))))-..))))))))))))).(((.((((((...)))))).))).))))). ( -29.40) >DroMoj_CAF1 50128 77 + 1 AGAU-AUA-A--AAUGUAUCUCCUACU--G-----------UGU------GUCUGCACUU---GCAACAAAGCACAGUGAAGCGGUCUAUGUCUAUAUA----CACA ((((-(((-.--..(((.((....(((--(-----------(((------...(((....---))).....))))))))).)))...))))))).....----.... ( -19.00) >consensus AGAU_AUA_A_AAUGGUUGCAC_U_CUGUCUGUCGCCUGCAAGU______AUUUGCAGUU_UAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA____UAUA ((((........(((.((((................((((((((......)))))))).....((......)).))))..))).......))))............. (-11.07 = -11.88 + 0.81)
| Location | 20,402,599 – 20,402,694 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 71.40 |
| Mean single sequence MFE | -24.62 |
| Consensus MFE | -6.76 |
| Energy contribution | -8.52 |
| Covariance contribution | 1.75 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.27 |
| SVM decision value | 1.62 |
| SVM RNA-class probability | 0.967923 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
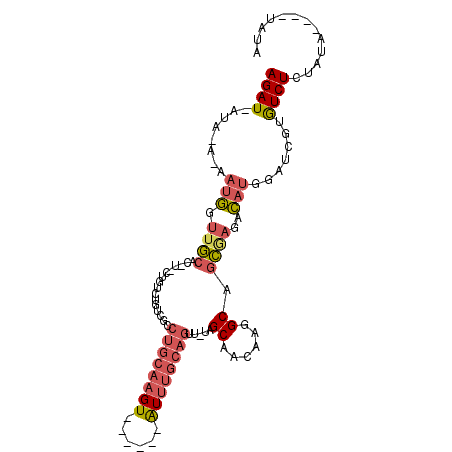
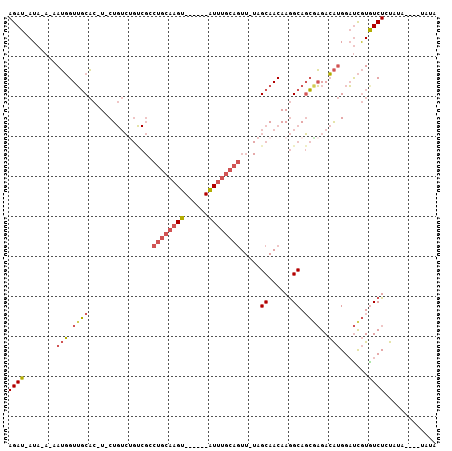
>3R_DroMel_CAF1 20402599 95 + 27905053 GCCUGCAAGU------AUUUGCAGUU-UAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA----UAUG--AAUGUAUAUAUACUGUACAUAUAGACAGAUAAA ((((((((..------..))))))..-..((......)).))((((((((...))))))))((((----((((--....))))))))((((........))))..... ( -24.30) >DroSec_CAF1 25207 97 + 1 GCCUGCAAGU------AUUUGCAGUUUUAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA----UAUAU-UAUGUAUAUAUACUGCAUAUGUAGCCAGAUAAA ..((((((..------..)))))).(((.((.(((..((((.((((((((...))))))))((((----(((((-...)))))))))))))...))).)).))).... ( -29.00) >DroSim_CAF1 28196 98 + 1 GCCUGCAAGU------AUUUGCAGUUUUAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA----UAUAUUUAUGUAUAUAUACUGCAUAUAUAGCCAGAUAAA ..(((...((------((.(((((.....((......))...((((((((...))))))))((((----(((((....)))))))))))))).))))...)))..... ( -27.30) >DroEre_CAF1 24721 83 + 1 GACUGCAAGU------AUUUGCAGUU-UAGCAACAAGGCAGCGAGACAUGGAUCGUAUCUGU------GUAUAU-A-----------AUGCAUAUAUAGCCAGAUAAA ((((((((..------..))))))))-..((......)).((((........))))(((((.------.(((((-(-----------.....))))))..)))))... ( -20.60) >DroYak_CAF1 28917 105 + 1 GACUGCAAGUACUCGUAUUUGCAGUU-UAGCAACAAGGCAGCAAGACAUGGAUCGUAUCUGUAUCUGUAUAUAU-ACUGCAUAUAU-AUGCAUAUAUAGACAGAUAAA (((((((((((....)))))))))))-..((......))....(((.(((...))).))).(((((((.(((((-(.(((((....-))))))))))).))))))).. ( -31.60) >DroMoj_CAF1 50152 80 + 1 -------UGU------GUCUGCACUU---GCAACAAAGCACAGUGAAGCGGUCUAUGUCUAUAUA----CACAU--ACAUGUACACAUUAUAUAUAGAGACA------ -------.((------.(((((((((---((......))).))))..(((...(((((.......----.))))--)..)))............)))).)).------ ( -14.90) >consensus GCCUGCAAGU______AUUUGCAGUU_UAGCAACAAGGCAGCGAGACAUGGAUCGUGUCUCUAUA____UAUAU_AAUGUAUAUAUACUGCAUAUAUAGACAGAUAAA ..((((((((......))))))))...........(((((.(((........))))))))................................................ ( -6.76 = -8.52 + 1.75)

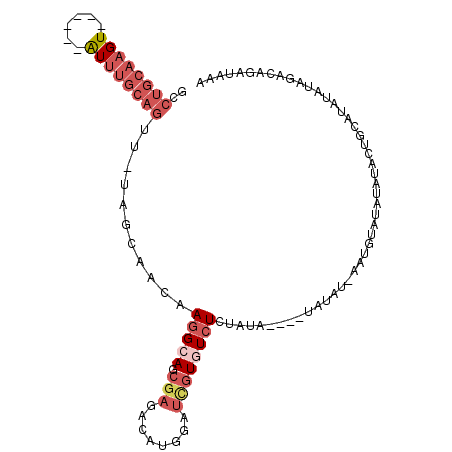
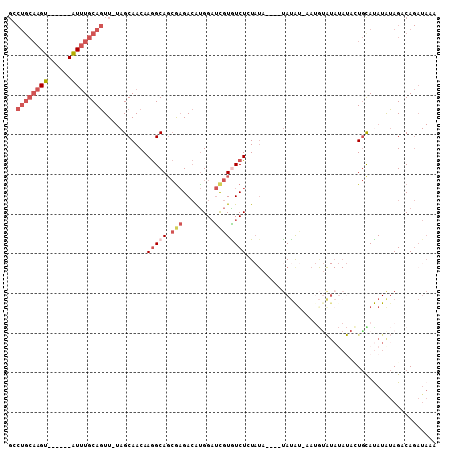
| Location | 20,402,599 – 20,402,694 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 71.40 |
| Mean single sequence MFE | -24.68 |
| Consensus MFE | -4.26 |
| Energy contribution | -5.12 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.17 |
| Mean z-score | -3.29 |
| Structure conservation index | 0.17 |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.954951 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20402599 95 - 27905053 UUUAUCUGUCUAUAUGUACAGUAUAUAUACAUU--CAUA----UAUAGAGACACGAUCCAUGUCUCGCUGCCUUGUUGCUA-AACUGCAAAU------ACUUGCAGGC .....(((((.....).))))(((((((.....--.)))----))))((((((.......)))))).((((....((((..-....))))..------....)))).. ( -21.00) >DroSec_CAF1 25207 97 - 1 UUUAUCUGGCUACAUAUGCAGUAUAUAUACAUA-AUAUA----UAUAGAGACACGAUCCAUGUCUCGCUGCCUUGUUGCUAAAACUGCAAAU------ACUUGCAGGC ......((((.(((...((((((((((((....-.))))----))).((((((.......)))))))))))..))).))))...((((((..------..)))))).. ( -30.50) >DroSim_CAF1 28196 98 - 1 UUUAUCUGGCUAUAUAUGCAGUAUAUAUACAUAAAUAUA----UAUAGAGACACGAUCCAUGUCUCGCUGCCUUGUUGCUAAAACUGCAAAU------ACUUGCAGGC ......((((.(((...((((((((((((......))))----))).((((((.......)))))))))))..))).))))...((((((..------..)))))).. ( -28.80) >DroEre_CAF1 24721 83 - 1 UUUAUCUGGCUAUAUAUGCAU-----------U-AUAUAC------ACAGAUACGAUCCAUGUCUCGCUGCCUUGUUGCUA-AACUGCAAAU------ACUUGCAGUC ..((((((..((((((.....-----------)-))))).------.)))))).............(((((....((((..-....))))..------....))))). ( -19.90) >DroYak_CAF1 28917 105 - 1 UUUAUCUGUCUAUAUAUGCAU-AUAUAUGCAGU-AUAUAUACAGAUACAGAUACGAUCCAUGUCUUGCUGCCUUGUUGCUA-AACUGCAAAUACGAGUACUUGCAGUC ..(((((((.(((((((((((-....))))).)-))))).))))))).(((((.......))))).(((((((((((((..-....)))...))))).....))))). ( -28.50) >DroMoj_CAF1 50152 80 - 1 ------UGUCUCUAUAUAUAAUGUGUACAUGU--AUGUG----UAUAUAGACAUAGACCGCUUCACUGUGCUUUGUUGC---AAGUGCAGAC------ACA------- ------(((((.(((((((((((....)))..--.))))----)))).)))))............(((..(((......---)))..)))..------...------- ( -19.40) >consensus UUUAUCUGGCUAUAUAUGCAGUAUAUAUACAUU_AUAUA____UAUAGAGACACGAUCCAUGUCUCGCUGCCUUGUUGCUA_AACUGCAAAU______ACUUGCAGGC ................((((.............................((((.......))))...........((((.......))))...........))))... ( -4.26 = -5.12 + 0.86)

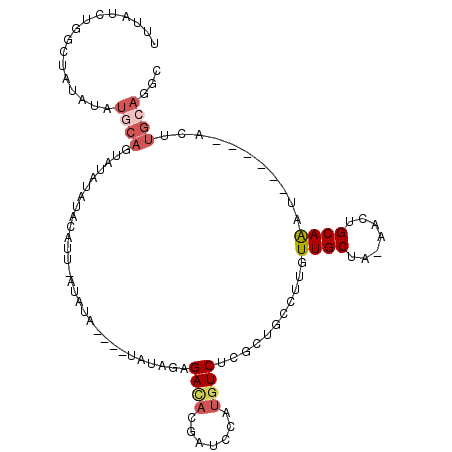
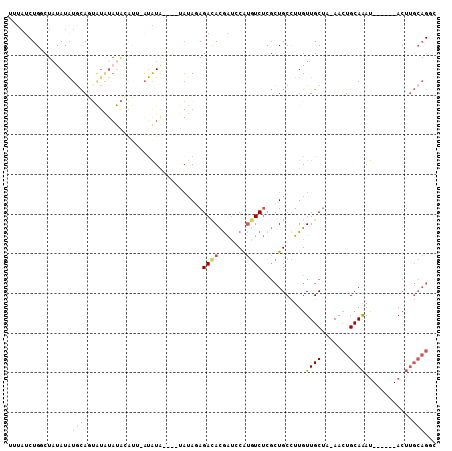
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:49:59 2006