
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 20,338,670 – 20,338,776 |
| Length | 106 |
| Max. P | 0.992588 |

| Location | 20,338,670 – 20,338,776 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 74.79 |
| Mean single sequence MFE | -33.23 |
| Consensus MFE | -20.45 |
| Energy contribution | -21.82 |
| Covariance contribution | 1.36 |
| Combinations/Pair | 1.11 |
| Mean z-score | -4.96 |
| Structure conservation index | 0.62 |
| SVM decision value | 2.34 |
| SVM RNA-class probability | 0.992588 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
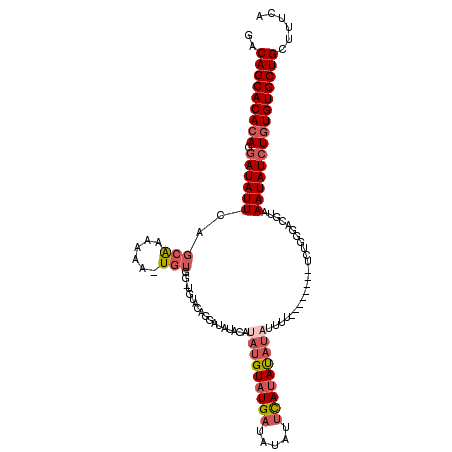
>3R_DroMel_CAF1 20338670 106 + 27905053 GACAGCACACAUGAUAUUCAGCAAAAAA-UGUGG-UGUACACGAUAUACAUAUGUAUGAUAUAUUCAUAUAUAUU-U--------UCUGAGACGUAAAUAUCUGUGUGCUGCUUUCA ..(((((((((.(((((((((.((((..-((((.-....)))).......(((((((((.....)))))))))))-)--------))))).......))))))))))))))...... ( -32.41) >DroVir_CAF1 188546 84 + 1 AACAGCACAAAUGAUAUUCGACGCAAAUUUGAAUUUGAA-------UCCUUUU---UGG-AAAUAUAU----------------------GACGUGAAUAUAUGUGUGCUGCACGAU ..(((((((....((((((.((((((((....)))))..-------(((....---.))-).......----------------------..)))))))))...)))))))...... ( -22.70) >DroSec_CAF1 147317 107 + 1 GACAGCACACAGGAUAUUCAGCAGAAAA-UGUGG-UGUACAGGAUAUACAUAUGUAUGAUAUAUUCAUAUAUAUUUU--------UCUGGGACGUAAAUAUCUGUGUGCUGCUUUCA ..((((((((((.((((((..(((((((-....(-((((.....)))))((((((((((.....)))))))))))))--------)))).)).....))))))))))))))...... ( -35.30) >DroSim_CAF1 156415 107 + 1 GACAGCACACAGGAUAUUCAGCAAAAAA-UGUGG-UGUACAGGAUAUACAUAUGUAUGAUAUAUUCAUAUAUAUUUU--------UCUGGGACGCAAAUAUCUGUGUGCUGCUUUCA ..((((((((((.(((((..(((.....-))).(-(((.(((((.....((((((((((.....))))))))))..)--------))))..)))).)))))))))))))))...... ( -36.90) >DroEre_CAF1 150518 111 + 1 GACAGCACACAUGAUAUUCAGCAAAAAA-UGUGG-UAUUCAGGAUA----CAUGUAUGAUAUAUUCAUACAUAUUUUUUUUUUUUUUUGGGAAGUAAAUAUCUGUGUGCUGCUUUCA ..(((((((((.(((((((..(((((((-....(-((((...))))----)((((((((.....))))))))..........))))))).)).....))))))))))))))...... ( -32.00) >DroYak_CAF1 155797 103 + 1 GACAGCACACAGGAUAUUCAGCAAAAAC-UUUGG-UAUUCAGGAUA----UAUGUAUGAUAUAUUCAUAUAUAUUUU--------UCUGGAACUUGAAUAUCUGUGUGCUGCUUGCA ..((((((((((.(((((((((((....-.))).-..(((((((.(----(((((((((.....))))))))))..)--------))))))..))))))))))))))))))...... ( -40.10) >consensus GACAGCACACAGGAUAUUCAGCAAAAAA_UGUGG_UGUACAGGAUAUACAUAUGUAUGAUAUAUUCAUAUAUAUUUU________UCUGGGACGUAAAUAUCUGUGUGCUGCUUUCA ..(((((((((.((((((..(((......)))..................(((((((((.....))))))))).......................)))))))))))))))...... (-20.45 = -21.82 + 1.36)

| Location | 20,338,670 – 20,338,776 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 74.79 |
| Mean single sequence MFE | -30.60 |
| Consensus MFE | -20.02 |
| Energy contribution | -21.55 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.08 |
| Mean z-score | -5.47 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.979344 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20338670 106 - 27905053 UGAAAGCAGCACACAGAUAUUUACGUCUCAGA--------A-AAUAUAUAUGAAUAUAUCAUACAUAUGUAUAUCGUGUACA-CCACA-UUUUUUGCUGAAUAUCAUGUGUGCUGUC .....((((((((((((((((((.....((((--------(-((((((((((.((.....)).)))))))))...(((....-.))).-.)))))).)))))))).)))))))))). ( -32.40) >DroVir_CAF1 188546 84 - 1 AUCGUGCAGCACACAUAUAUUCACGUC----------------------AUAUAUUU-CCA---AAAAGGA-------UUCAAAUUCAAAUUUGCGUCGAAUAUCAUUUGUGCUGUU .....((((((((.(((((((((((..----------------------.......(-((.---....)))-------..(((((....)))))))).)))))).)).)))))))). ( -21.20) >DroSec_CAF1 147317 107 - 1 UGAAAGCAGCACACAGAUAUUUACGUCCCAGA--------AAAAUAUAUAUGAAUAUAUCAUACAUAUGUAUAUCCUGUACA-CCACA-UUUUCUGCUGAAUAUCCUGUGUGCUGUC .....((((((((((((((((((.....((((--------(((((((.(((((.....))))).))))((((.....)))).-.....-))))))).))))))).))))))))))). ( -34.10) >DroSim_CAF1 156415 107 - 1 UGAAAGCAGCACACAGAUAUUUGCGUCCCAGA--------AAAAUAUAUAUGAAUAUAUCAUACAUAUGUAUAUCCUGUACA-CCACA-UUUUUUGCUGAAUAUCCUGUGUGCUGUC .....((((((((((((((((((((.......--------...((((.(((((.....))))).))))((((.....)))).-.....-.....))).)))))).))))))))))). ( -30.60) >DroEre_CAF1 150518 111 - 1 UGAAAGCAGCACACAGAUAUUUACUUCCCAAAAAAAAAAAAAAAUAUGUAUGAAUAUAUCAUACAUG----UAUCCUGAAUA-CCACA-UUUUUUGCUGAAUAUCAUGUGUGCUGUC .....((((((((((((((((((.....(((((((........((((((((((.....)))))))))----)....((....-.))..-))))))).)))))))).)))))))))). ( -34.00) >DroYak_CAF1 155797 103 - 1 UGCAAGCAGCACACAGAUAUUCAAGUUCCAGA--------AAAAUAUAUAUGAAUAUAUCAUACAUA----UAUCCUGAAUA-CCAAA-GUUUUUGCUGAAUAUCCUGUGUGCUGUC .....(((((((((((((((((..((((..((--------...((((.(((((.....))))).)))----).))..)))).-....(-((....))))))))).))))))))))). ( -31.30) >consensus UGAAAGCAGCACACAGAUAUUUACGUCCCAGA________AAAAUAUAUAUGAAUAUAUCAUACAUAUGUAUAUCCUGUACA_CCACA_UUUUUUGCUGAAUAUCAUGUGUGCUGUC .....(((((((((((((((((..........................(((((.....))))).....((((.....)))).................))))))).)))))))))). (-20.02 = -21.55 + 1.53)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:49:31 2006