
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 20,277,894 – 20,278,061 |
| Length | 167 |
| Max. P | 0.994134 |

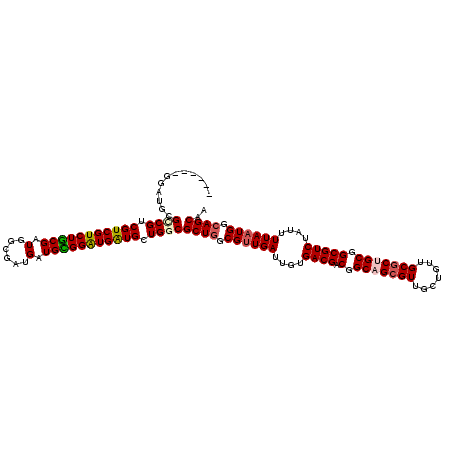
| Location | 20,277,894 – 20,278,008 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.27 |
| Mean single sequence MFE | -54.73 |
| Consensus MFE | -46.45 |
| Energy contribution | -46.78 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.13 |
| Mean z-score | -3.84 |
| Structure conservation index | 0.85 |
| SVM decision value | 2.45 |
| SVM RNA-class probability | 0.994134 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20277894 114 + 27905053 ------UGCUGCGCCGUCGUCGUCUGCGAUGGCGAUGAUGCGGGUGGUGUUGUCGCUGGCGUUGAUUAUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUA ------.(((((((((((((((((.(((..(((((..((((.....))))..)))))..))).)))...))))))))).(((((((.(.....).)))))))...........))))).. ( -56.10) >DroVir_CAF1 115259 120 + 1 AUGAUUGGAUGCGCCGUCGUCGUCUGCGAUGACGAUGAUGUGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGAAGCAA ............((((.(((((((..((.(......).))..))))))).))))(((..((((((....((((.(.(((((((.......))))))).)))))....))))))..))).. ( -48.80) >DroGri_CAF1 111894 120 + 1 AUGCAAGGACGCGCCGUCGUCGUCUGCGAUGGCGAUGAUGUGGAUGAUGUUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAA .(((......((((((.(((((((..((.(......).))..))))))).))))))((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))). ( -55.30) >DroWil_CAF1 95618 114 + 1 ------UACUGCGUCGUCGUCGUCUACGAUGGCGAUGAUGUGGAUGAUGCUGGCGCUGGCGUUGAUUAUGACGACGGCAGCGUUGUUGUUGCACUGCGGCGUCUAUUUUAAUGGCAGCUA ------..(..(((((((((((((...)))))))))))))..)...........((((.((((((....((((.(.((((.((.......)).)))).)))))....)))))).)))).. ( -51.30) >DroMoj_CAF1 117835 120 + 1 AUCUUAGGAAACGCCGUCGUCGUCUGCGAUGACGAUGGUGCGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAA ......(....)((((.(((((((((((..........))))))))))).))))((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).)))).. ( -58.10) >DroAna_CAF1 91031 114 + 1 ------UGCUGCGCCGUCGUCGUCUGCGAUGGCGAUGGUGCGGAUGGUGUUGUCGCUGGCGCUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCAA ------((((((((((((((((((.(((..(((((..(..(.....)..)..)))))..))).)))...))))))))).(((((((.(.....).)))))))...........)))))). ( -58.80) >consensus ______GGAUGCGCCGUCGUCGUCUGCGAUGGCGAUGAUGCGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAA ............((((.(((((((((((.(......).))))))))))).))))((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).)))).. (-46.45 = -46.78 + 0.33)

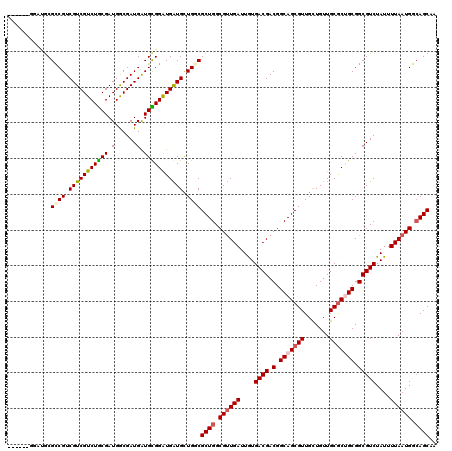
| Location | 20,277,894 – 20,278,008 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.27 |
| Mean single sequence MFE | -35.49 |
| Consensus MFE | -28.46 |
| Energy contribution | -27.77 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.730506 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20277894 114 - 27905053 UAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCAUAAUCAACGCCAGCGACAACACCACCCGCAUCAUCGCCAUCGCAGACGACGACGGCGCAGCA------ ..(((((................(((..((....))..))).(((((((((.............(((...........)))......((....))....)))))))))))))).------ ( -39.00) >DroVir_CAF1 115259 120 - 1 UUGCUUCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCAUCAUCCACAUCAUCGUCAUCGCAGACGACGACGGCGCAUCCAAUCAU .(((.............(((((.((((((.........)))))).).)))).........(((.((....))..........((.(((((......))))).)).))))))......... ( -34.00) >DroGri_CAF1 111894 120 - 1 UUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAACAUCAUCCACAUCAUCGCCAUCGCAGACGACGACGGCGCGUCCUUGCAU ..(((((................)))))((((.....((((.(((((((((........((....))....................((....))....)))))))))))))..)))).. ( -35.39) >DroWil_CAF1 95618 114 - 1 UAGCUGCCAUUAAAAUAGACGCCGCAGUGCAACAACAACGCUGCCGUCGUCAUAAUCAACGCCAGCGCCAGCAUCAUCCACAUCAUCGCCAUCGUAGACGACGACGACGCAGUA------ ..(((((................((((((.........))))))(((((((....((.(((...(((...................)))...))).)).)))))))..))))).------ ( -28.81) >DroMoj_CAF1 117835 120 - 1 UUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCAUCAUCCGCACCAUCGUCAUCGCAGACGACGACGGCGUUUCCUAAGAU ................(((((((((((((.........)))))).((((((.............((....))...............(((......))))))))))))))))........ ( -35.20) >DroAna_CAF1 91031 114 - 1 UUGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCACAAUCAGCGCCAGCGACAACACCAUCCGCACCAUCGCCAUCGCAGACGACGACGGCGCAGCA------ .((((((................(((..((....))..))).(((((((((....((.(((...((((.................))))...))).)).)))))))))))))))------ ( -40.53) >consensus UUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAACAUCAUCCACAUCAUCGCCAUCGCAGACGACGACGGCGCAGCA______ .................(.((((((((((.........))))))(((((((........((....))....................((....)).)))))))..))))).......... (-28.46 = -27.77 + -0.69)



| Location | 20,277,928 – 20,278,047 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 87.88 |
| Mean single sequence MFE | -51.02 |
| Consensus MFE | -41.06 |
| Energy contribution | -41.95 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.80 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.87 |
| SVM RNA-class probability | 0.871150 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
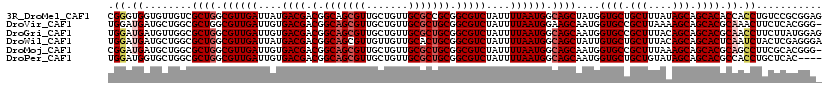
>3R_DroMel_CAF1 20277928 119 + 27905053 CGGGUGGUGUUGUCGCUGGCGUUGAUUAUGACGACGGCAGCGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUAUGGUGCUGCUUUAUAGCAGCACACCACCUGUCCGCGGAG ((((((((......((((.((((((....((((.(.((.((((.......)))).)).)))))....)))))).))))....((((((((....))))))))))))))))((....)). ( -57.40) >DroVir_CAF1 115299 118 + 1 UGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGAAGCAAUGGUGCCGCUUAAAAGCAGCACGCAAACUUCUCACGGG- .(((...(((.(((((((.(((((.......))))).)))))))((((((.....((((((.(((((...........))))))))))).....))))))..)))....)))......- ( -43.50) >DroGri_CAF1 111934 119 + 1 UGGAUGAUGUUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCCGCUUUACAGCAGCACGCAACCUUCUUAUGGAG ........((((.(((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))....((((.(((....))).)))))))))((((....)))) ( -48.50) >DroWil_CAF1 95652 119 + 1 UGGAUGAUGCUGGCGCUGGCGUUGAUUAUGACGACGGCAGCGUUGUUGUUGCACUGCGGCGUCUAUUUUAAUGGCAGCUAUUGUGCUGCUUUACAGCAGCACUCAAUCUACUCGAGGGA (((((((.......((((.((((((....((((.(.((((.((.......)).)))).)))))....)))))).))))....((((((((....)))))))))).)))))......... ( -45.00) >DroMoj_CAF1 117875 118 + 1 CGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCCGCUUUAAAGCAGCACGCAGCCUUCGCACGGG- ((..(((.((((.(((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))....((((.(((....))).)))))))))..)))..))..- ( -54.20) >DroPer_CAF1 94648 115 + 1 UGGAUGGUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCUGCUGUAUAGCAGCACGCCACCUGCUCAC---- ((..(((.(.((((((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))....((((((((....))))))))))))))))..)).---- ( -57.50) >consensus UGGAUGAUGCUGGCGCUGGCGUUGAUUGUGACGACGGCAGCGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCCGCUUUAAAGCAGCACGCAACCUUCUCACGGG_ .((.((........((((.((((((....((((.(.(((((((.......))))))).)))))....)))))).))))....((((.(((....))).)))).)).))........... (-41.06 = -41.95 + 0.89)


| Location | 20,277,928 – 20,278,047 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 87.88 |
| Mean single sequence MFE | -40.99 |
| Consensus MFE | -34.19 |
| Energy contribution | -34.05 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.92 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.44 |
| SVM RNA-class probability | 0.953942 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20277928 119 - 27905053 CUCCGCGGACAGGUGGUGUGCUGCUAUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACGCUGCCGUCGUCAUAAUCAACGCCAGCGACAACACCACCCG ...........((((((((((((((....))))))......((((...........(((((.((((((.........)))))).).))))...........))))....)))))))).. ( -43.75) >DroVir_CAF1 115299 118 - 1 -CCCGUGAGAAGUUUGCGUGCUGCUUUUAAGCGGCACCAUUGCUUCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCAUCAUCCA -...(((((((((.((.((((((((....))))))))))..)))))..........((((..((((((.........)))))))))).))))...........((....))........ ( -39.90) >DroGri_CAF1 111934 119 - 1 CUCCAUAAGAAGGUUGCGUGCUGCUGUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAACAUCAUCCA ........((..((((.((((((.(((...((((((....))))))..........(((((.((((((.........)))))).).)))).......))).)))))))))).))..... ( -40.70) >DroWil_CAF1 95652 119 - 1 UCCCUCGAGUAGAUUGAGUGCUGCUGUAAAGCAGCACAAUAGCUGCCAUUAAAAUAGACGCCGCAGUGCAACAACAACGCUGCCGUCGUCAUAAUCAACGCCAGCGCCAGCAUCAUCCA ...........(..(((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))........((....)).)))).)))..). ( -36.80) >DroMoj_CAF1 117875 118 - 1 -CCCGUGCGAAGGCUGCGUGCUGCUUUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCAUCAUCCG -...((((...(((((.((((((((....))))))))))..((((...........(((((.((((((.........)))))).).))))...........))))))).))))...... ( -42.25) >DroPer_CAF1 94648 115 - 1 ----GUGAGCAGGUGGCGUGCUGCUAUACAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCACCAUCCA ----.......(.((((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))...........)))))))).)........ ( -42.55) >consensus _CCCGUGAGAAGGUUGCGUGCUGCUAUAAAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACGCUGCCGUCGUCACAAUCAACGCCAGCGCCAGCAUCAUCCA ...........(((.((((((((((....))))))))....((((...........(((((.((((((.........)))))).).))))...........))))....)))))..... (-34.19 = -34.05 + -0.14)


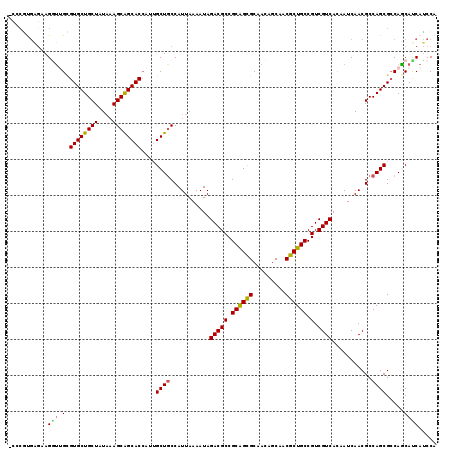
| Location | 20,277,968 – 20,278,061 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 76.13 |
| Mean single sequence MFE | -33.55 |
| Consensus MFE | -28.05 |
| Energy contribution | -28.55 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.75 |
| SVM RNA-class probability | 0.975258 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20277968 93 + 27905053 CGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCUAUGGUGCUGCUUUAUAGCAGCACACCACCUGUCCGCGGAG----GUU---GU------CCUGCU------CCU .(((((((((((((....)))).......)))))))))....((((((((....))))))))...........((((..----...---..------.)))).------... ( -32.81) >DroPse_CAF1 96233 95 + 1 CGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCUGCUGUAUAGCAGCACGCCACCUGCUCAC---------UU---GU-----AUCUACUCUGGGCUCU ((((((((((((((....))))..........))))))))))((((((((....)))))))).......(((((.---------..---((-----....))..)))))... ( -35.10) >DroGri_CAF1 111974 97 + 1 CGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCCGCUUUACAGCAGCACGCAACCUUCUUAUGGAG----GUACAAGUGCU--GUUAA---------CU ((((((((((((((....))))..........))))))))))............((((((((...((((((....))))----))....)))))--)))..---------.. ( -36.30) >DroMoj_CAF1 117915 93 + 1 CGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCCGCUUUAAAGCAGCACGCAGCCUUCGCACGGG-----UUA-----GGUAGGUCUA---------CU ((((((((((((((....))))..........))))))))))((((.(((....))).))))..(((((......)))-----)).-----((((....))---------)) ( -30.20) >DroAna_CAF1 91105 97 + 1 CGUUGCUGUUGCGCCGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCUGCUUUACAGCAGCACACCACCUACCCACAGAGUCUUGUU---GC------CCCGCU------ACU .((.((....((((....))))..........((((((((..((((((((....))))))))...((((......)).)).)))))---))------)..)).------)). ( -31.80) >DroPer_CAF1 94688 95 + 1 CGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCUGCUGUAUAGCAGCACGCCACCUGCUCAC---------UU---GU-----AUCUACUCUGGGCUCU ((((((((((((((....))))..........))))))))))((((((((....)))))))).......(((((.---------..---((-----....))..)))))... ( -35.10) >consensus CGUUGCUGUUGCGCUGCGGCGUCUAUUUUAAUGGCAGCAAUGGUGCUGCUUUAUAGCAGCACGCCACCUGCUCACGGA_____GUU___GU______UCUACU_______CU ((((((((((((((....))))..........))))))))))((((((((....)))))))).................................................. (-28.05 = -28.55 + 0.50)


| Location | 20,277,968 – 20,278,061 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 76.13 |
| Mean single sequence MFE | -30.81 |
| Consensus MFE | -24.23 |
| Energy contribution | -23.98 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.63 |
| SVM RNA-class probability | 0.968760 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20277968 93 - 27905053 AGG------AGCAGG------AC---AAC----CUCCGCGGACAGGUGGUGUGCUGCUAUAAAGCAGCACCAUAGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACG .(.------.(((((------..---..)----))((((((.(.((..((((((((((....))))))))....))..)).((......)).))))))).))..)....... ( -33.10) >DroPse_CAF1 96233 95 - 1 AGAGCCCAGAGUAGAU-----AC---AA---------GUGAGCAGGUGGCGUGCUGCUAUACAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACG ................-----..---..---------((..((.((..((((((((((....))))))))....))..)).........(.(((....))))....)).)). ( -27.20) >DroGri_CAF1 111974 97 - 1 AG---------UUAAC--AGCACUUGUAC----CUCCAUAAGAAGGUUGCGUGCUGCUGUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACG ..---------.....--.((.(((((..----....)))))...(((((((((((((....))))))))....(((((................)))))))))).)).... ( -32.39) >DroMoj_CAF1 117915 93 - 1 AG---------UAGACCUACC-----UAA-----CCCGUGCGAAGGCUGCGUGCUGCUUUAAAGCGGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACG .(---------(((.(((.((-----...-----.....).).)))))))((((((((....))))))))....(((((................)))))((....)).... ( -27.69) >DroAna_CAF1 91105 97 - 1 AGU------AGCGGG------GC---AACAAGACUCUGUGGGUAGGUGGUGUGCUGCUGUAAAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCGGCGCAACAGCAACG .((------.(((((------..---..).....(((((.((((.(..((((((((((....))))))).)))..)))))......)))))..)))).))((....)).... ( -37.30) >DroPer_CAF1 94688 95 - 1 AGAGCCCAGAGUAGAU-----AC---AA---------GUGAGCAGGUGGCGUGCUGCUAUACAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACG ................-----..---..---------((..((.((..((((((((((....))))))))....))..)).........(.(((....))))....)).)). ( -27.20) >consensus AG_______AGUAGA______AC___AAC_____UCCGUGAGCAGGUGGCGUGCUGCUAUAAAGCAGCACCAUUGCUGCCAUUAAAAUAGACGCCGCAGCGCAACAGCAACG ............................................((((((((((((((....))))))))....)))))).........(.(((....)))).......... (-24.23 = -23.98 + -0.25)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:49:02 2006