
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 20,227,344 – 20,227,501 |
| Length | 157 |
| Max. P | 0.999585 |

| Location | 20,227,344 – 20,227,461 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.07 |
| Mean single sequence MFE | -43.28 |
| Consensus MFE | -35.26 |
| Energy contribution | -35.82 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.26 |
| SVM RNA-class probability | 0.658451 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20227344 117 + 27905053 AUACUCAUGGUUUGGCU---CAUCGUAUAUUAUCCACGAUCAUCGUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAGAUUCCACCAGCCAGCCGCGAACCCAAGUGGUCGC ((((..((((......)---))).))))........((((((((((((....))))))...((((.(((.((..((((.((.........)).))))..)).))).))))...)))))). ( -38.60) >DroVir_CAF1 49076 117 + 1 ACGCUCAUGGUUUGGCU---CGUUGUAUAUUAUCCACGAUCAUCGUCGCGUGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCUGCUGCGAACCCAAGUGGACGU ..((.((.....)))).---............(((((.....((((((....))))))...(((((((((.((.((((.((.........)).)))).)).)))))))))..)))))... ( -43.00) >DroPse_CAF1 44409 117 + 1 AUGCUCAUGGUUUGGCU---CAUUAUAUAUUAUCCACGAUCAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUAAGAUUCCACCAGCCGGCUGCGAACCCUAGCGGUCGC ..((.((.....)))).---................(((((.((((((....))))))..((((((((((.((.((((.((.........)).))))))..))))))))))...))))). ( -43.10) >DroGri_CAF1 52531 117 + 1 ACGCUCAUGGUUUGGCU---CAUUGUAUAUUAUCCACGAUCAUCGUCGCGGGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGU ..((.((.....)))).---............(((((.....((((((....))))))...(((((((((.((.((((.((.........)).)))).)).)))))))))..)))))... ( -44.80) >DroWil_CAF1 39845 120 + 1 GUGCUGAUGGUAUUGCUGGAUAUUGUAUAUUAUCCACGAUCAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGC .(((.((((((((((.((((((........)))))))))).))))))))).(((.(.(...(((((((((.((.((((.((.........)).)))).)).)))))))))...).).))) ( -47.10) >DroPer_CAF1 42909 117 + 1 AUGCUCAUGGUUUGGCU---CAUUAUAUAUUAUCCACGAUCAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUAAGAUUCCACCAGCCGGCUGCGAACCCUAGCGGUCGC ..((.((.....)))).---................(((((.((((((....))))))..((((((((((.((.((((.((.........)).))))))..))))))))))...))))). ( -43.10) >consensus AUGCUCAUGGUUUGGCU___CAUUGUAUAUUAUCCACGAUCAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGC ..((.((.....)))).................((((.....((((((....))))))...(((((((((.(..((((.((.........)).))))..).)))))))))..)))).... (-35.26 = -35.82 + 0.56)


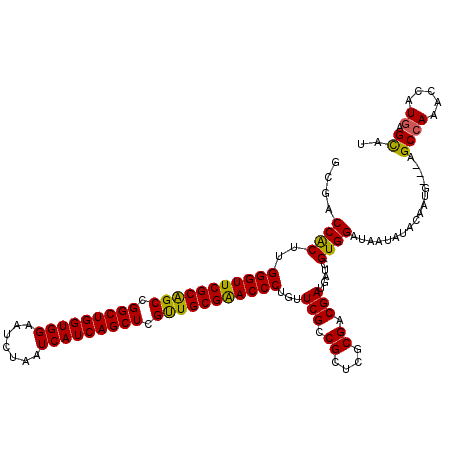
| Location | 20,227,344 – 20,227,461 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.07 |
| Mean single sequence MFE | -50.26 |
| Consensus MFE | -44.16 |
| Energy contribution | -43.55 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.92 |
| Structure conservation index | 0.88 |
| SVM decision value | 3.49 |
| SVM RNA-class probability | 0.999290 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20227344 117 - 27905053 GCGACCACUUGGGUUCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGAUGAUCGUGGAUAAUAUACGAUG---AGCCAAACCAUGAGUAU ((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).)))).(((((......(.((((((.......)))))).---).........))))).. ( -48.26) >DroVir_CAF1 49076 117 - 1 ACGUCCACUUGGGUUCGCAGCAGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCACGCGACGAUGAUCGUGGAUAAUAUACAACG---AGCCAAACCAUGAGCGU ..((((((..(((((((((((..((((((((.......))))))))..)))))))))))...(((.((....)).))).....))))))...........---.((((.....)).)).. ( -47.70) >DroPse_CAF1 44409 117 - 1 GCGACCGCUAGGGUUCGCAGCCGGCUGGUGGAAUCUUAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUGAUCGUGGAUAAUAUAUAAUG---AGCCAAACCAUGAGCAU ((((..((.((((((((((((.(((((((((.......))))))))).)))))))))))))))))).((((((((......))))((...((.....)).---..)).......)))).. ( -50.20) >DroGri_CAF1 52531 117 - 1 ACGUCCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCCCGCGACGAUGAUCGUGGAUAAUAUACAAUG---AGCCAAACCAUGAGCGU ..((((((..(((((((((((((((((((((.......)))))))))))))))))))))...(((.((....)).))).....))))))...........---.((((.....)).)).. ( -52.60) >DroWil_CAF1 39845 120 - 1 GCGUCCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUGAUCGUGGAUAAUAUACAAUAUCCAGCAAUACCAUCAGCAC (((..((...(((((((((((((((((((((.......)))))))))))))))))))))))..))).(((.((((......))))(((((........)))))))).............. ( -52.60) >DroPer_CAF1 42909 117 - 1 GCGACCGCUAGGGUUCGCAGCCGGCUGGUGGAAUCUUAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUGAUCGUGGAUAAUAUAUAAUG---AGCCAAACCAUGAGCAU ((((..((.((((((((((((.(((((((((.......))))))))).)))))))))))))))))).((((((((......))))((...((.....)).---..)).......)))).. ( -50.20) >consensus GCGACCACUUGGGUUCGCAGCCGGCUGGUGGAAUCUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUGAUCGUGGAUAAUAUACAAUG___AGCCAAACCAUGAGCAU ....((((..(((((((((((.(((((((((.......))))))))).)))))))))))...(((.((....)).))).....)))).................((((.....)).)).. (-44.16 = -43.55 + -0.61)


| Location | 20,227,381 – 20,227,501 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.11 |
| Mean single sequence MFE | -43.60 |
| Consensus MFE | -36.88 |
| Energy contribution | -37.33 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.71 |
| SVM RNA-class probability | 0.828339 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20227381 120 + 27905053 CAUCGUCGCGAGCGGCGAACAGGGUCCGCAGCGAGCUGAUGAUUAGAUUCCACCAGCCAGCCGCGAACCCAAGUGGUCGCUAUAAGGUGUUCUCGCUGCCCAUCUUGAGCAGCUCGAACU ........(((((.((...((((((..((((((((((((...))))....(((((((..(((((........))))).)))....))))..))))))))..)))))).)).))))).... ( -45.00) >DroVir_CAF1 49113 120 + 1 CAUCGUCGCGUGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCUGCUGCGAACCCAAGUGGACGUUAUAAGGAGUUUUCACUACCCAUUUUGAGCAGCUCGAAUU ..((((((....))))))...(((((((((.((.((((.((.........)).)))).)).))))))))).((((((..((......))..))))))......((((((...)))))).. ( -40.60) >DroGri_CAF1 52568 120 + 1 CAUCGUCGCGGGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGUUAUAAGGUGUUAUCACUGCCCAUUUUAAGCAGCUCGAAUU ........(((((.((.....(((((((((.((.((((.((.........)).)))).)).)))))))))((((((.........((((....))))..))))))...)).))))).... ( -42.90) >DroWil_CAF1 39885 120 + 1 CAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGCUAUAAGGUAUUUUCACUGCCCAUUUUCAGUAAUUCGAAUU ..((((((....))))))...(((((((((.((.((((.((.........)).)))).)).))))))))).((((((.(((....)))...))))))....................... ( -40.30) >DroMoj_CAF1 46553 120 + 1 CAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGACGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGUCGCUAUAAGGAGUUGUCACUGCCCAUUUUGAGCAGCUCGAACU ........(((((.((.....(((((((((.((.((((..((......))...)))).)).))))))))).(((((..(((......)))..)))))...........)).))))).... ( -45.10) >DroPer_CAF1 42946 120 + 1 CAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUAAGAUUCCACCAGCCGGCUGCGAACCCUAGCGGUCGCUAUAAGGUGUUGUCGCUGCCCAUCUUGAGCAGCUCGAAUU ..((((((....))))))..((((((((((.((.((((.((.........)).))))))..)))))))))).((((.((((....)))))))).(((((.(.....).)))))....... ( -47.70) >consensus CAUCGUCGCGAGCGGCGAACAGGGUUCGCAACGAGCUGAUGAUUAAAUUCCACCAGCCCGCUGCGAACCCAAGUGGACGCUAUAAGGUGUUGUCACUGCCCAUUUUGAGCAGCUCGAAUU ........(((((.((.....(((((((((.((.((((..((......))...)))).)).))))))))).(((((.((((....))))...)))))...........)).))))).... (-36.88 = -37.33 + 0.45)


| Location | 20,227,381 – 20,227,501 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.11 |
| Mean single sequence MFE | -54.17 |
| Consensus MFE | -47.85 |
| Energy contribution | -46.92 |
| Covariance contribution | -0.94 |
| Combinations/Pair | 1.26 |
| Mean z-score | -4.34 |
| Structure conservation index | 0.88 |
| SVM decision value | 3.75 |
| SVM RNA-class probability | 0.999585 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 20227381 120 - 27905053 AGUUCGAGCUGCUCAAGAUGGGCAGCGAGAACACCUUAUAGCGACCACUUGGGUUCGCGGCUGGCUGGUGGAAUCUAAUCAUCAGCUCGCUGCGGACCCUGUUCGCCGCUCGCGACGAUG .((((..(((((((.....)))))))..))))........((((.((...(((((((((((.(((((((((.......))))))))).))))))))))))).))))((....))...... ( -59.30) >DroVir_CAF1 49113 120 - 1 AAUUCGAGCUGCUCAAAAUGGGUAGUGAAAACUCCUUAUAACGUCCACUUGGGUUCGCAGCAGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCACGCGACGAUG ...(((.(((((.....(((((.(((....))).)))))..((..((...(((((((((((..((((((((.......))))))))..)))))))))))))..))..))).))..))).. ( -48.30) >DroGri_CAF1 52568 120 - 1 AAUUCGAGCUGCUUAAAAUGGGCAGUGAUAACACCUUAUAACGUCCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCCCGCGACGAUG .......(((((((.....)))))))...............((((.....(((((((((((((((((((((.......)))))))))))))))))))))...((((.....)))).)))) ( -52.30) >DroWil_CAF1 39885 120 - 1 AAUUCGAAUUACUGAAAAUGGGCAGUGAAAAUACCUUAUAGCGUCCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUG ...(((..((((((........))))))............(((..((...(((((((((((((((((((((.......)))))))))))))))))))))))..)))((....)).))).. ( -50.30) >DroMoj_CAF1 46553 120 - 1 AGUUCGAGCUGCUCAAAAUGGGCAGUGACAACUCCUUAUAGCGACCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCGUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUG .(((((((((((((.....)))))................((((.((...(((((((((((((((((..((.......))..))))))))))))))))))).)))).))))).))).... ( -57.00) >DroPer_CAF1 42946 120 - 1 AAUUCGAGCUGCUCAAGAUGGGCAGCGACAACACCUUAUAGCGACCGCUAGGGUUCGCAGCCGGCUGGUGGAAUCUUAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUG ...(((.(((((((.....)))))))..............(((((.(((((((((((((((.(((((((((.......))))))))).)))))))))))))...)).).))))..))).. ( -57.80) >consensus AAUUCGAGCUGCUCAAAAUGGGCAGUGAAAACACCUUAUAGCGACCACUUGGGUUCGCAGCGGGCUGGUGGAAUUUAAUCAUCAGCUCGUUGCGAACCCUGUUCGCCGCUCGCGACGAUG ...(((.(((((((.....)))))))........................(((((((((((.(((((((((.......))))))))).)))))))))))...((((.....))))))).. (-47.85 = -46.92 + -0.94)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:48:39 2006