
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 19,947,768 – 19,948,008 |
| Length | 240 |
| Max. P | 0.862180 |

| Location | 19,947,768 – 19,947,888 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.94 |
| Mean single sequence MFE | -51.05 |
| Consensus MFE | -37.27 |
| Energy contribution | -36.80 |
| Covariance contribution | -0.47 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.36 |
| SVM RNA-class probability | 0.702730 |
| Prediction | RNA |
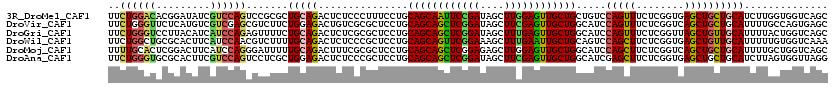
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19947768 120 - 27905053 AGCUGUUUGGUACCCUCGCUGGUCUCCAGCUGGGCCUUGGUGGUGCGCAGGGUCUCGCUGGUGUUCUGGAAUUUCACCUGCAUGUCGUGCAGGGCCAGCUGGUAUUUGUGGAGCGCCGCC .(((.(..((((((...(((((((.(((((.(((((((((.....).)))))))).))))).).............(((((((...))))))))))))).))))))...).)))...... ( -51.40) >DroVir_CAF1 10012 120 - 1 AGCUGUUUGGUGCCCUCGCUGGUAUCCAGCUGAGAUCGUGUGCUGCGCAACGAUUCCGAGCUCUGCUGGAACUUGACCUGCAUGUCGUGCAGGGCCAGCUGGUAUUUGUGGAGCGCCGCC .(((.(..((((((...((((((.((((((.((((((((.(((...))))))))......))).))))))......(((((((...))))))))))))).))))))...).)))...... ( -49.00) >DroGri_CAF1 9988 120 - 1 AGCUGUUUGGUGCCCUCGCUGGUAUCCAGUUGGGUUCUGGUGCUGCGCAACGUUUCGCUACUAUGCUGGAACUUCACCUGCAUAUCGUGUAGCGCCAGUUGAUACUUGUGGAGCGCUGCC .(((.(..(((((((..(((((...))))).)))).((((((((((((...((......))(((((.((.......)).)))))..)))))))))))).....)))...).)))...... ( -44.00) >DroWil_CAF1 9331 120 - 1 AGCUGUUUGGUGCCAUCGCUGGUGUCCAGUUGAGUUUUGGUCGAGCGUAGAGUCUCGGUCGUAUGCUGGAAUUUCACUUGCAUGUCGUGCAAGGCCAGUUGGUACUUGUGGAGGGCAGCC .((((((((((((((..((((((.((((((.......((..(((((.....).))))..))...))))))......(((((((...))))))))))))))))))))......))))))). ( -49.20) >DroMoj_CAF1 10465 120 - 1 AGCUGCUUGGUGCCCUCGCUGGUCUCCAGCUGGGAGCGAGUGCUGCGCAGCGUUUCGCUGCUCUGCUGGAACUUCACCUGCAUGUCGUGCAGGGCCAGCUGGUACUUGUGGAGCGCUGCC .((.((....((((((.(((((...))))).))).)))...)).))((((((((((((((((..(((((.......(((((((...))))))).))))).))))...)))))))))))). ( -60.60) >DroAna_CAF1 8849 120 - 1 AGCUGCUUGGUGCCUUCACUGGUGUCCAACUGGGACUUGGUGGUGCGGAGAGUGUCACUGGUGUGCUGGAACUUCACCUGCAUGUCGUGUAGGGCCAGUUGGUACUUGUGGAGCGCCGCC ........(((((.(((((.((((.((((((((..((.(((((..(.....)..)))))((..(((.((.......)).)))..))....))..)))))))))))).))))))))))... ( -52.10) >consensus AGCUGUUUGGUGCCCUCGCUGGUAUCCAGCUGGGAUCUGGUGCUGCGCAGCGUUUCGCUGCUAUGCUGGAACUUCACCUGCAUGUCGUGCAGGGCCAGCUGGUACUUGUGGAGCGCCGCC .(((.(..((((((...((((((.((((((........((((..((.....))..)))).....))))))......(((((((...))))))))))))).))))))...).)))...... (-37.27 = -36.80 + -0.47)



| Location | 19,947,848 – 19,947,968 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.89 |
| Mean single sequence MFE | -51.17 |
| Consensus MFE | -39.38 |
| Energy contribution | -37.58 |
| Covariance contribution | -1.80 |
| Combinations/Pair | 1.46 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.83 |
| SVM RNA-class probability | 0.862180 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19947848 120 - 27905053 CCCUUUCCUGCAGCAAUUCCGAUAGCUUGGAGUUGCUGCUGUCCAGUUUCUCGGUGAGCUGCUGCAUCUUGGUGGUCAGCAGCUGUUUGGUACCCUCGCUGGUCUCCAGCUGGGCCUUGG .((......(((((((((((((....))))))))))))).((((((((..((((((((((((((.(((.....)))))))))).....((...))))))))).....))))))))...)) ( -51.20) >DroVir_CAF1 10092 120 - 1 CGCGCUCCUGCAGCAGCUCGGAUAGCUUCGAGUUGCUGGCAUCCAGUUUCUCGGUCAGCUGCUGCAUUUUGCCAGUGAGCAGCUGUUUGGUGCCCUCGCUGGUAUCCAGCUGAGAUCGUG (((((((.((((((((((..(((.....((((..(((((...)))))..)))))))))))))))))...((((((((((..(((....)))...)))))))))).......)))..)))) ( -51.60) >DroGri_CAF1 10068 120 - 1 CGCGCUCCUGCAGCAGCUCGGAUAGCUUUGAGUUGCUGGCAUCCAGUUUCUCGGUUAGCUGUUGCAUUUUACUGGUCAGCAGCUGUUUGGUGCCCUCGCUGGUAUCCAGUUGGGUUCUGG .((((....))(((((((((((....))))))))))).))..((((....((((.(((((((((.(((.....)))))))))))).)))).((((..(((((...))))).)))).)))) ( -45.80) >DroWil_CAF1 9411 120 - 1 CCCGCUCCUGCAGCAGUUCGGAAAGCUUUGAAUUGCUGCAGUCCAGCUUCUCGGUGAGCUGUUGCAUUUUUGUGGUCAAAAGCUGUUUGGUGCCAUCGCUGGUGUCCAGUUGAGUUUUGG .((((..(((((((((((((((....)))))))))))))))..((((((......))))))..........))))...((((((..(((((((((....))))).))))...)))))).. ( -44.30) >DroMoj_CAF1 10545 120 - 1 CGCGCUCCUGCAGCAGCUCGGAGAGCUUGGAGUUGCUGGCAUCCAGCUUCUCGGUCAGCUGCUGCAUUUUGCUGGUCAGCAGCUGCUUGGUGCCCUCGCUGGUCUCCAGCUGGGAGCGAG ..(((((((((((((((((.(((((((.(((((.....)).)))))).)))).)..))))))))).....(((((..(.((((.((.....))....)))).)..))))).))))))).. ( -59.90) >DroAna_CAF1 8929 120 - 1 CCCGCUCCUGCAGCAGCUCGGAUAGCUUCGAGUUGCUGGCAUCGAGCUUCUCGGUGAGCUGCUGCAUCUUAGUGGUUAGGAGCUGCUUGGUGCCUUCACUGGUGUCCAACUGGGACUUGG (((((((((.((((((((((((....)))))))))))).(((((((...)))))))(((..(((.....)))..)))))))))...((((((((......)))).))))..)))...... ( -54.20) >consensus CCCGCUCCUGCAGCAGCUCGGAUAGCUUCGAGUUGCUGGCAUCCAGCUUCUCGGUGAGCUGCUGCAUUUUGCUGGUCAGCAGCUGUUUGGUGCCCUCGCUGGUAUCCAGCUGGGAUCUGG ...(((..((((((((((((((....)))))))))))).))...)))..((((((.((((((((.(((.....)))))))))))...(((((((......)))).)))))))))...... (-39.38 = -37.58 + -1.80)

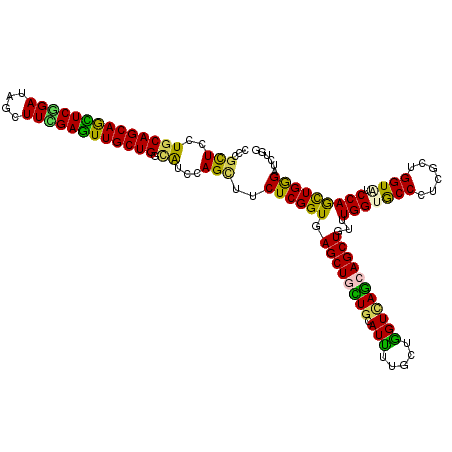

| Location | 19,947,888 – 19,948,008 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.72 |
| Mean single sequence MFE | -43.49 |
| Consensus MFE | -26.12 |
| Energy contribution | -25.77 |
| Covariance contribution | -0.36 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.95 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.783409 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19947888 120 - 27905053 UUCUGGACACGGAUAUCGUCCAGUCCGCGCUGCAGACUCUCCCUUUCCUGCAGCAAUUCCGAUAGCUUGGAGUUGCUGCUGUCCAGUUUCUCGGUGAGCUGCUGCAUCUUGGUGGUCAGC ..(((..(((((((........))))..((.((((.(((.((.......(((((((((((((....)))))))))))))......(.....))).))))))).))......)))..))). ( -46.50) >DroVir_CAF1 10132 120 - 1 UUCUGGGUUCUCAUGUCGUCGAGCGUCUUCUGGAGACUGUCGCGCUCCUGCAGCAGCUCGGAUAGCUUCGAGUUGCUGGCAUCCAGUUUCUCGGUCAGCUGCUGCAUUUUGCCAGUGAGC ..((((..............((((((...(.(....).)..)))))).((((((((((..(((.....((((..(((((...)))))..))))))))))))))))).....))))..... ( -46.00) >DroGri_CAF1 10108 120 - 1 UUCUGGGUCCUUACAUCAUCCAGAGUUUUCUGCAGACUCUCGCGCUCCUGCAGCAGCUCGGAUAGCUUUGAGUUGCUGGCAUCCAGUUUCUCGGUUAGCUGUUGCAUUUUACUGGUCAGC ((((((((.........))))))))......((.((((..........((((((((((.....((((..(((..(((((...)))))..))))))))))))))))).......)))).)) ( -36.03) >DroWil_CAF1 9451 120 - 1 UUCUGGCUGCGCACUUCAUCCAACGUCUUUUGCAGACUCUCCCGCUCCUGCAGCAGUUCGGAAAGCUUUGAAUUGCUGCAGUCCAGCUUCUCGGUGAGCUGUUGCAUUUUUGUGGUCAAA ...((((..(.....(((((....((((.....))))......(((.(((((((((((((((....)))))))))))))))...))).....)))))((....))......)..)))).. ( -41.10) >DroMoj_CAF1 10585 120 - 1 UUUUGCACUCGGACUUCAUCCAGGGAUUUUUGCAGACUUUCGCGCUCCUGCAGCAGCUCGGAGAGCUUGGAGUUGCUGGCAUCCAGCUUCUCGGUCAGCUGCUGCAUUUUGCUGGUCAGC ...((((((((((.....))).))).....))))((((...(((....(((((((((((.(((((((.(((((.....)).)))))).)))).)..))))))))))...))).))))... ( -44.00) >DroAna_CAF1 8969 120 - 1 UUCUGGGUGCGCACUUCGUCCAGUCCUCGCUGGAGACUCUCCCGCUCCUGCAGCAGCUCGGAUAGCUUCGAGUUGCUGGCAUCGAGCUUCUCGGUGAGCUGCUGCAUCUUAGUGGUUAGG ....(((.(((....((.((((((....))))))))......))).))).((((((((((((....)))))))))))).(((((((...)))))))(((..(((.....)))..)))... ( -47.30) >consensus UUCUGGGUGCGCACUUCAUCCAGCGUUUUCUGCAGACUCUCCCGCUCCUGCAGCAGCUCGGAUAGCUUCGAGUUGCUGGCAUCCAGCUUCUCGGUGAGCUGCUGCAUUUUGCUGGUCAGC ..((((((.........)))))).......(((((...............((((((((((((....)))))))))))).....(((((........)))))))))).............. (-26.12 = -25.77 + -0.36)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:46:50 2006