| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 19,869,749 – 19,869,862 |
| Length | 113 |
| Max. P | 0.888529 |

| Location | 19,869,749 – 19,869,862 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.17 |
| Mean single sequence MFE | -47.37 |
| Consensus MFE | -22.30 |
| Energy contribution | -24.38 |
| Covariance contribution | 2.08 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.47 |
| SVM decision value | 0.95 |
| SVM RNA-class probability | 0.888529 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19869749 113 + 27905053 GCUGCAGCGGCCGCGGAUGCUGCGGCAGCCGUCACACCGGGCACCACAGU---UCCGGGCACGCCCCAUGCUACAUUCGGCGGCACCAUGCGGUAUCCAGCCGCUGAAAAU----UGUAU ....(((((((...((((((((((...((((((...(((((.((....))---)))))(((.......))).......))))))....)))))))))).))))))).....----..... ( -50.40) >DroVir_CAF1 737 112 + 1 GCGGCAGCAGCCGCGCUGGCA---GCGGCAGACACGCCUGGCACCACAGUGGCGCCUGGCACUCCCCAUGCCAUAUUUGGCGGCAUCACACGGUACCCGGCAGCUGCAAAG----GACA- ...(((((.(((((.......---)))))......(((.((.(((...(((..((((((......))).((((....)))))))..)))..))).)).))).)))))....----....- ( -49.80) >DroPse_CAF1 356 110 + 1 GCUGCUGCGGCCGCAGACG---AGGCCGCCGACAUGCCUGGGACAACAGU---ACCGGGUACACCCCAUGCCACAUUCGGUGGCACCAUGCGAUACCCGGCAGCUGGAAAG----AUCAU ((((((((((((.......---.))))))((.(((((((((.((....))---.))))).........((((((.....)))))).))))))......)))))).......----..... ( -45.30) >DroWil_CAF1 642 117 + 1 GCUGCUGCAGCCGCUGUGGCAGCAGCGGCAGUCACACCUGGCACUACUGU---ACCGGGCACUCCCCAUGCCACAUUCGGCGGCACCAUGCGAUAUCCAGCAGCUAAAUAAAAAAAACAU (((((((..((((((((....)))))))).(((...(((((.((....))---.)))))..........(((......)))))).............)))))))................ ( -45.10) >DroAna_CAF1 432 107 + 1 GCUGCUGCGGCGGCGGUGGCUGCG---GCCGUUACGCCCGGCACCACAGU---ACCUGGCACGCCCCAUGCCACAUUUGGUGGCACCAUACGGUAUCCGGCAGCUGCAAAU----AA--- ((.(((((((((((((((.(((.(---((......)))))))))).(((.---..))))).))))...((((((.....)))))).....(((...)))))))).))....----..--- ( -48.30) >DroPer_CAF1 356 110 + 1 GCUGCUGCGGCCGCAGACG---AGGCCGCCGACAUGCCUGGGACAACAGU---ACCGGGUACACCCCAUGCCACAUUCGGUGGCACCAUGCGAUACCCGGCAGCUGGAAAG----AUCAU ((((((((((((.......---.))))))((.(((((((((.((....))---.))))).........((((((.....)))))).))))))......)))))).......----..... ( -45.30) >consensus GCUGCUGCGGCCGCGGAGGC__CGGCCGCCGACACGCCUGGCACCACAGU___ACCGGGCACACCCCAUGCCACAUUCGGCGGCACCAUGCGAUACCCGGCAGCUGAAAAG____AACAU (((((((((((.((..........)).))))...(((.(((..((...........(((....)))...(((......)))))..))).))).....)))))))................ (-22.30 = -24.38 + 2.08)

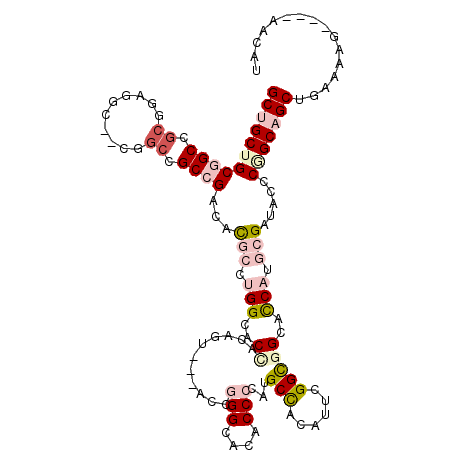

| Location | 19,869,749 – 19,869,862 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.17 |
| Mean single sequence MFE | -51.18 |
| Consensus MFE | -28.37 |
| Energy contribution | -27.73 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.35 |
| SVM RNA-class probability | 0.698782 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19869749 113 - 27905053 AUACA----AUUUUCAGCGGCUGGAUACCGCAUGGUGCCGCCGAAUGUAGCAUGGGGCGUGCCCGGA---ACUGUGGUGCCCGGUGUGACGGCUGCCGCAGCAUCCGCGGCCGCUGCAGC .((((----...(((.(((((.....(((....)))))))).)))))))((.((.((((.(((((((---.(((((((..(((......)))..)))))))..)))).))))))).)))) ( -53.00) >DroVir_CAF1 737 112 - 1 -UGUC----CUUUGCAGCUGCCGGGUACCGUGUGAUGCCGCCAAAUAUGGCAUGGGGAGUGCCAGGCGCCACUGUGGUGCCAGGCGUGUCUGCCGC---UGCCAGCGCGGCUGCUGCCGC -....----....(((((((((.((((.((.(((((((.(((.....((((((.....))))))((((((.....)))))).)))))))).)))).---)))).).))))))))...... ( -54.60) >DroPse_CAF1 356 110 - 1 AUGAU----CUUUCCAGCUGCCGGGUAUCGCAUGGUGCCACCGAAUGUGGCAUGGGGUGUACCCGGU---ACUGUUGUCCCAGGCAUGUCGGCGGCCU---CGUCUGCGGCCGCAGCAGC .....----.....(((.(((((((((.(((.(.(((((((.....))))))).).)))))))))))---)))).........((.(((..((((((.---.......)))))).))))) ( -49.60) >DroWil_CAF1 642 117 - 1 AUGUUUUUUUUAUUUAGCUGCUGGAUAUCGCAUGGUGCCGCCGAAUGUGGCAUGGGGAGUGCCCGGU---ACAGUAGUGCCAGGUGUGACUGCCGCUGCUGCCACAGCGGCUGCAGCAGC ................(((((((....((((((.(((((((.....)))))))(((.....)))(((---((....)))))..))))))..(((((((......)))))))..))))))) ( -52.50) >DroAna_CAF1 432 107 - 1 ---UU----AUUUGCAGCUGCCGGAUACCGUAUGGUGCCACCAAAUGUGGCAUGGGGCGUGCCAGGU---ACUGUGGUGCCGGGCGUAACGGC---CGCAGCCACCGCCGCCGCAGCAGC ---..----....((.((((((((...)))....(((((((.....)))))))((((((.(((.(((---((....))))).))).....(((---....)))..)))).))))))).)) ( -47.80) >DroPer_CAF1 356 110 - 1 AUGAU----CUUUCCAGCUGCCGGGUAUCGCAUGGUGCCACCGAAUGUGGCAUGGGGUGUACCCGGU---ACUGUUGUCCCAGGCAUGUCGGCGGCCU---CGUCUGCGGCCGCAGCAGC .....----.....(((.(((((((((.(((.(.(((((((.....))))))).).)))))))))))---)))).........((.(((..((((((.---.......)))))).))))) ( -49.60) >consensus AUGUU____CUUUCCAGCUGCCGGAUACCGCAUGGUGCCACCGAAUGUGGCAUGGGGAGUGCCCGGU___ACUGUGGUGCCAGGCGUGACGGCCGCCG__GCCACCGCGGCCGCAGCAGC ................(((((.((...((((.(.(((((((.....))))))).)(..(((((.((....((....)).)).)))))..)................)))))))))))... (-28.37 = -27.73 + -0.64)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:46:27 2006