
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 19,799,887 – 19,800,094 |
| Length | 207 |
| Max. P | 0.948133 |

| Location | 19,799,887 – 19,799,981 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | forward |
| Mean pairwise identity | 82.25 |
| Mean single sequence MFE | -48.70 |
| Consensus MFE | -36.29 |
| Energy contribution | -37.80 |
| Covariance contribution | 1.51 |
| Combinations/Pair | 1.26 |
| Mean z-score | -3.61 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948133 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19799887 94 + 27905053 AGC--UGCCCGAUACCGGGCGCCGGAUCGGGUUC-ACUGACGAGCCUGGGCCUGAAGGAGCUGCUCGAGCUCCACAGGCAAAGGCUUGAGCAAUCGA .((--((((((((.((((...)))))))))))..-.....(((((((..(((((..((((((.....)))))).)))))..))))))))))...... ( -52.30) >DroGri_CAF1 35154 91 + 1 GGUGCCG---AAUCCG---CGGCUGGUGACGUUGCUGCGUCCGGUAUGUGUCUCAAGGAGCUGCUCGAGCUUCACCGGCAGCGACUUGAGCAAUCGA .((((((---....((---((((((....))..))))))..)))))).((.((((((..(((((.((........)))))))..))))))))..... ( -41.10) >DroSec_CAF1 30280 94 + 1 AGC--UGCCCGAUUCCGGGCGCCGGAUCGGGUUC-GCUGACGAGCCUGGGCCUGAAGGAGCUGCUCGAGCUCCACAGGCAAAGGCUUGAGCAAUCGA (((--.(((((((.((((...)))))))))))..-)))..(((((((..(((((..((((((.....)))))).)))))..)))))))......... ( -52.30) >DroSim_CAF1 30333 94 + 1 AGC--UGCCCGAUUCCGGGCGCCGGAUCGGGUUC-GCUGACGAGCCUGGGCCUGAAGGAGCUGCUCGAGCUCCACAGGCAAAGGCUUGAGCAAUCGA (((--.(((((((.((((...)))))))))))..-)))..(((((((..(((((..((((((.....)))))).)))))..)))))))......... ( -52.30) >DroEre_CAF1 30850 94 + 1 AGC--UGCCCGCUUCCGGGCGCCGAAUCCGCUUC-GCUGGCGGGCCUGAACCUGAAGGAGCUGCUCGAGCUCCACAGGCAAAGGCUCGAGCAAUCGA ...--.(((((....)))))(((((..(((((..-...)))))((((...((((..((((((.....)))))).))))...))))))).))...... ( -44.50) >DroYak_CAF1 30858 94 + 1 AGC--UGCCCCAUUCCGGGCGCCGGAUCGGGUUC-GCUAGCGGGCCUGGGCCUGAAGGAGCUGCUCGAGCUCCACAGGCAAAGGCUCGAGCAAUCGA .((--((((((..(((((...)))))..)))...-)).)))((((((..(((((..((((((.....)))))).)))))..))))))((....)).. ( -49.70) >consensus AGC__UGCCCGAUUCCGGGCGCCGGAUCGGGUUC_GCUGACGAGCCUGGGCCUGAAGGAGCUGCUCGAGCUCCACAGGCAAAGGCUUGAGCAAUCGA (((...(((((((.(((.....))))))))))...)))..(((((((..(((((..((((((.....)))))).)))))..)))))))......... (-36.29 = -37.80 + 1.51)


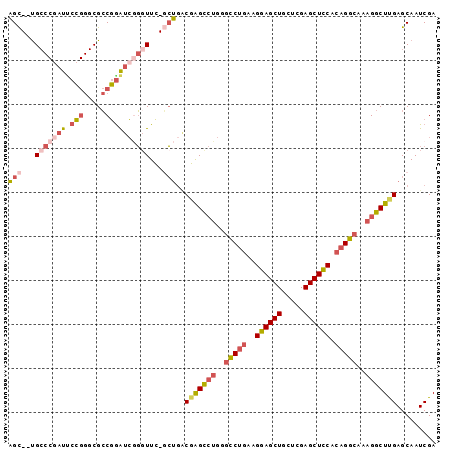
| Location | 19,799,887 – 19,799,981 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 97 |
| Reading direction | reverse |
| Mean pairwise identity | 82.25 |
| Mean single sequence MFE | -43.35 |
| Consensus MFE | -27.90 |
| Energy contribution | -28.98 |
| Covariance contribution | 1.09 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.56 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.806106 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19799887 94 - 27905053 UCGAUUGCUCAAGCCUUUGCCUGUGGAGCUCGAGCAGCUCCUUCAGGCCCAGGCUCGUCAGU-GAACCCGAUCCGGCGCCCGGUAUCGGGCA--GCU ((.((((..(.(((((..(((((.((((((.....))))))..)))))..))))).).))))-)).((((((((((...)))).))))))..--... ( -44.30) >DroGri_CAF1 35154 91 - 1 UCGAUUGCUCAAGUCGCUGCCGGUGAAGCUCGAGCAGCUCCUUGAGACACAUACCGGACGCAGCAACGUCACCAGCCG---CGGAUU---CGGCACC .....((((((((..((((((((......))).)))))..)))))).)).......((((......))))....((((---......---))))... ( -31.10) >DroSec_CAF1 30280 94 - 1 UCGAUUGCUCAAGCCUUUGCCUGUGGAGCUCGAGCAGCUCCUUCAGGCCCAGGCUCGUCAGC-GAACCCGAUCCGGCGCCCGGAAUCGGGCA--GCU ....((((((.(((((..(((((.((((((.....))))))..)))))..))))).)..)))-)).((((((((((...)))).))))))..--... ( -46.00) >DroSim_CAF1 30333 94 - 1 UCGAUUGCUCAAGCCUUUGCCUGUGGAGCUCGAGCAGCUCCUUCAGGCCCAGGCUCGUCAGC-GAACCCGAUCCGGCGCCCGGAAUCGGGCA--GCU ....((((((.(((((..(((((.((((((.....))))))..)))))..))))).)..)))-)).((((((((((...)))).))))))..--... ( -46.00) >DroEre_CAF1 30850 94 - 1 UCGAUUGCUCGAGCCUUUGCCUGUGGAGCUCGAGCAGCUCCUUCAGGUUCAGGCCCGCCAGC-GAAGCGGAUUCGGCGCCCGGAAGCGGGCA--GCU ......((.(((((((..(((((.((((((.....))))))..)))))..))))((((....-...))))..)))))(((((....))))).--... ( -47.10) >DroYak_CAF1 30858 94 - 1 UCGAUUGCUCGAGCCUUUGCCUGUGGAGCUCGAGCAGCUCCUUCAGGCCCAGGCCCGCUAGC-GAACCCGAUCCGGCGCCCGGAAUGGGGCA--GCU ....(((((((.((((..(((((.((((((.....))))))..)))))..)))).))..)))-)).((((.(((((...))))).))))...--... ( -45.60) >consensus UCGAUUGCUCAAGCCUUUGCCUGUGGAGCUCGAGCAGCUCCUUCAGGCCCAGGCCCGUCAGC_GAACCCGAUCCGGCGCCCGGAAUCGGGCA__GCU ..(..(((((..((((..(((((.((((((.....))))))..)))))..)))).................((((.....))))...)))))...). (-27.90 = -28.98 + 1.09)

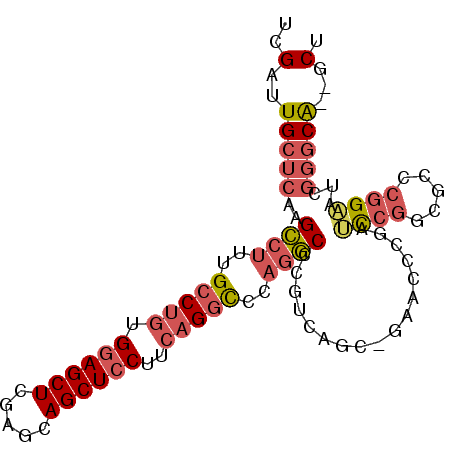
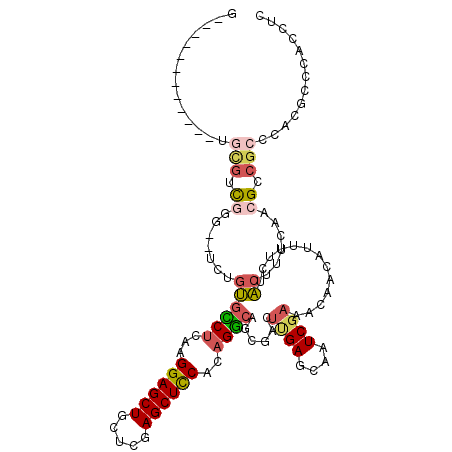
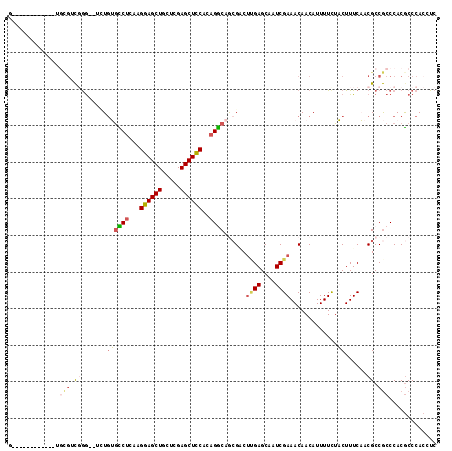
| Location | 19,799,918 – 19,800,021 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 77.34 |
| Mean single sequence MFE | -35.35 |
| Consensus MFE | -14.27 |
| Energy contribution | -15.80 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.37 |
| Structure conservation index | 0.40 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.733441 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
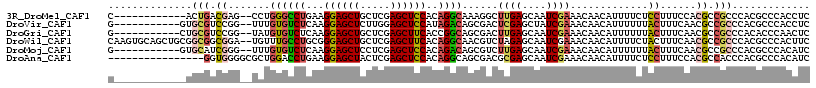
>3R_DroMel_CAF1 19799918 103 + 27905053 C------------ACUGACGAG--CCUGGGCCUGAAGGAGCUGCUCGAGCUCCACAGGCAAAGGCUUGAGCAAUCGAAACAACAUUUUCUCCUUUCCACGCCGCCCACGCCCACCUC .------------.....((((--(((..(((((..((((((.....)))))).)))))..))))))).......((((......))))............................ ( -32.70) >DroVir_CAF1 33751 104 + 1 G-----------GUGCGUCCGG--UUUGUGUCUCAAGGAGCUCUUGGAGCUCCAUAGACAGCGACUCGAGCUAUCGAAACAACAUUUUUUACUUUCAACGCCGCCCACGCCCACCUC (-----------(.((((.(((--(((((((((...(((((((...)))))))..))))).))).((((....))))......................))))...))))))..... ( -32.80) >DroGri_CAF1 35181 104 + 1 G-----------CUGCGUCCGG--UAUGUGUCUCAAGGAGCUGCUCGAGCUUCACCGGCAGCGACUUGAGCAAUCGAAACAACAUUUUUUACUUUCAACGCCGCCCACACCCAACUC (-----------(.((((..((--((.((((((((((..(((((.((........)))))))..))))))...........))))....))))....)))).))............. ( -30.00) >DroWil_CAF1 34014 115 + 1 CAAGUGCAGCUGCGGCGGCGGA--UGUUUGCCUGCGGGAGCUGCUCGAGCUUCACAGGCAACGUCUAGAGCAAUCGAAACAACAUUUUCUACUUUCAACGCCGCCCACGCCCACUUC .(((((..((((.(((((((((--((((.(((((..((((((.....)))))).)))))))))))..((....))((((......)))).........))))))))).)).))))). ( -47.70) >DroMoj_CAF1 36831 104 + 1 G-----------GUGCAUCGGG--UUUGUGUCUCAAGGAGCUCCUCGAGCUCCACAGACAGCGUCUUGAGCAAUCGAAACAACAUUUUUUACUUUCAACGCCGCCCACGCCCACAUC (-----------(.((...(((--(...(((((...(((((((...)))))))..)))))((((...((....))((((.............)))).)))).))))..))))..... ( -31.32) >DroAna_CAF1 32231 101 + 1 ----------------GGUGGGGCGCUGGACCUGAAGGAGCUACUCGAGCUCCACAGGCAGCGACGCGAGCAAUCGAAACAACAUUUUCUCCUUUCCACGCCACCCACGCCCACAUC ----------------(((((((((.(((.((((..((((((.....)))))).))))..(((....((....))((((......)))).........)))...)))))))).)))) ( -37.60) >consensus G____________UGCGUCGGG__UCUGUGCCUCAAGGAGCUGCUCGAGCUCCACAGGCAGCGACUUGAGCAAUCGAAACAACAUUUUCUACUUUCAACGCCGCCCACGCCCACCUC ..............(((.((.......((((((...((((((.....))))))..))))......((((....)))).............))......)).)))............. (-14.27 = -15.80 + 1.53)
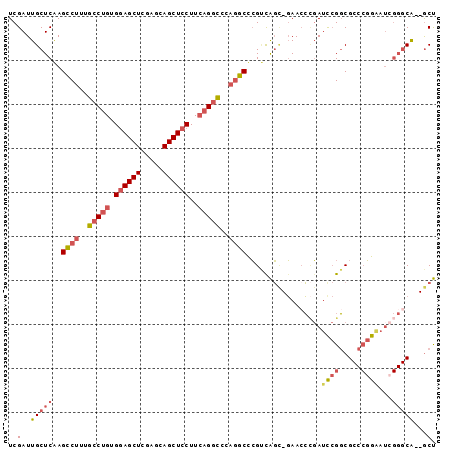
| Location | 19,799,918 – 19,800,021 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 77.34 |
| Mean single sequence MFE | -41.66 |
| Consensus MFE | -21.60 |
| Energy contribution | -22.02 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.790284 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19799918 103 - 27905053 GAGGUGGGCGUGGGCGGCGUGGAAAGGAGAAAAUGUUGUUUCGAUUGCUCAAGCCUUUGCCUGUGGAGCUCGAGCAGCUCCUUCAGGCCCAGG--CUCGUCAGU------------G ..(..((((.((((((((.(((..((..((((......))))..))..))).))).....(((.((((((.....))))))..)))))))).)--)))..)...------------. ( -42.90) >DroVir_CAF1 33751 104 - 1 GAGGUGGGCGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUUUCGAUAGCUCGAGUCGCUGUCUAUGGAGCUCCAAGAGCUCCUUGAGACACAAA--CCGGACGCAC-----------C ..((((.(..((((((((.((((...........(((((....))))))))))))))(((((..(((((((...)))))))...)))))....--)))..).)))-----------) ( -37.20) >DroGri_CAF1 35181 104 - 1 GAGUUGGGUGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUUUCGAUUGCUCAAGUCGCUGCCGGUGAAGCUCGAGCAGCUCCUUGAGACACAUA--CCGGACGCAG-----------C ..(((.(((((((((((((((..........))))))))).....((((((((..((((((((......))).)))))..)))))).))))))--)).)))....-----------. ( -40.20) >DroWil_CAF1 34014 115 - 1 GAAGUGGGCGUGGGCGGCGUUGAAAGUAGAAAAUGUUGUUUCGAUUGCUCUAGACGUUGCCUGUGAAGCUCGAGCAGCUCCCGCAGGCAAACA--UCCGCCGCCGCAGCUGCACUUG .((((((((.(((((((((((...((((((((......))))...))))...))).(((((((((.((((.....))))..)))))))))...--...)))))).))))).))))). ( -46.30) >DroMoj_CAF1 36831 104 - 1 GAUGUGGGCGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUUUCGAUUGCUCAAGACGCUGUCUGUGGAGCUCGAGGAGCUCCUUGAGACACAAA--CCCGAUGCAC-----------C (...((((...((((((((((...(((((...............)))))...))))))))))(((...(((((((....))))))).)))...--))))...)..-----------. ( -36.96) >DroAna_CAF1 32231 101 - 1 GAUGUGGGCGUGGGUGGCGUGGAAAGGAGAAAAUGUUGUUUCGAUUGCUCGCGUCGCUGCCUGUGGAGCUCGAGUAGCUCCUUCAGGUCCAGCGCCCCACC---------------- ..((.(((((((((((((((((..((..((((......))))..))..))))))))).(((((.((((((.....))))))..)))))))).)))))))..---------------- ( -46.40) >consensus GAGGUGGGCGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUUUCGAUUGCUCAAGUCGCUGCCUGUGGAGCUCGAGCAGCUCCUUCAGACACAAA__CCCGACGCA____________C ....((((((.((((((((((..........))))))))))....)))))).......((((..((((((.....))))))...))))............................. (-21.60 = -22.02 + 0.42)

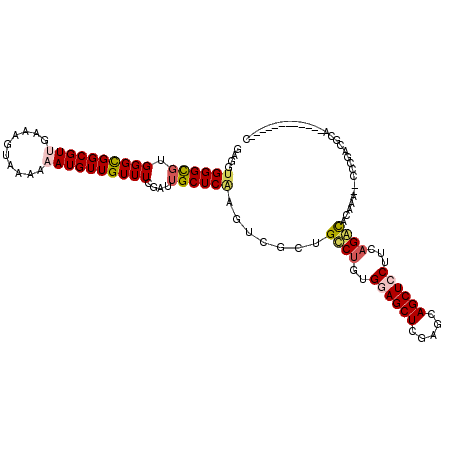
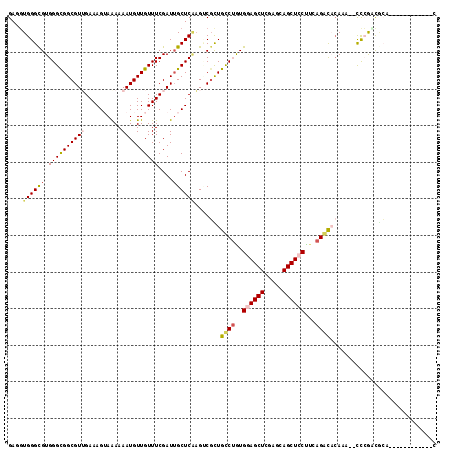
| Location | 19,799,981 – 19,800,094 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.23 |
| Mean single sequence MFE | -39.93 |
| Consensus MFE | -22.82 |
| Energy contribution | -23.85 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.584397 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19799981 113 - 27905053 --CGCCU-----CGUUGCUCGUCUCGCCGACUGUCAGCGAGGAGGGCGUCGGAGAGUGGCACAGUGAGAUGGAGCCACAGGAGGUGGGCGUGGGCGGCGUGGAAAGGAGAAAAUGUUGUU --(((((-----((.(((((..(((((.(.....).)))))..))))).).....(((((.((......))..)))))..))))))......((((((((............)))))))) ( -41.80) >DroVir_CAF1 33815 110 - 1 --CUGGU-----AGUUGCUUGACUCGCCCACUGUUAGCGA---GGGCGUGGGUGAGACACAGAGUGACAUGGAGCCACAGGAGGUGGGCGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUU --...((-----.((..(((..(((((((((.(((.....---.)))))))))))).....)))..))......((((.....))))))...(((((((((..........))))))))) ( -35.20) >DroPse_CAF1 33412 110 - 1 --CAGCU-----CGGUGCUCGUCUCUCCCACUGUCAGCGA---GGGUGUGGGCGAGUGGCAGAGCGAGAUGGAGCCGCAGGAUGUGGGCGUGGGCGGUGAUGAAAGGAGAAAAUGUUGUU --(((((-----(.((.((((.((((.((((((((.(((.---...))).))).))))).)))))))))).))(((((...(((....)))..)))))................)))).. ( -37.30) >DroGri_CAF1 35245 98 - 1 --CUCGU-----UGUUGCUCGUCUCGCCCACUGUCAGCGA---CGGCGUGGGUGAGACACAGAGC------G------AGGAGUUGGGUGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUU --(((((-----(.(((((((((((((((((.(((.....---.))))))))))))))...))))------)------).))..))))....(((((((((..........))))))))) ( -39.40) >DroWil_CAF1 34089 110 - 1 --CAGCU-----CAUUGGUCGUCUCGCCCACUGUCAAUGA---GGGAGUGGGUGAUACGCACAGUGAGAUGGAACCACAGGAAGUGGGCGUGGGCGGCGUUGAAAGUAGAAAAUGUUGUU --((.((-----(((((..(((.(((((((((.((.....---)).))))))))).)))..))))))).))...((((.....)))).....(((((((((..........))))))))) ( -43.90) >DroMoj_CAF1 36895 111 - 1 CGCCGGUUAGCGAGUUGCUUGUGUCGCCCACUGUCAGCGA---UGGCGUGGGUGAGACACAGAG------GGAGCCACAGGAUGUGGGCGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUU (((((.(((((...((.((((((((((((((.((((....---))))))))))..)))))).))------.)).((((.....)))))).))).)))))..................... ( -42.00) >consensus __CAGCU_____CGUUGCUCGUCUCGCCCACUGUCAGCGA___GGGCGUGGGUGAGACACAGAGUGAGAUGGAGCCACAGGAGGUGGGCGUGGGCGGCGUUGAAAGUAAAAAAUGUUGUU .............(((((((..(((((((((.(((.........))))))))))))..................((((.....))))....)))))))...................... (-22.82 = -23.85 + 1.03)

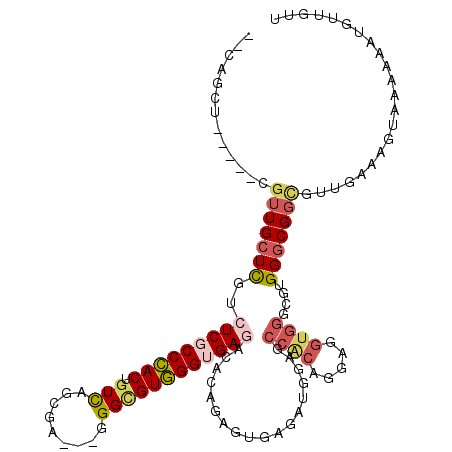
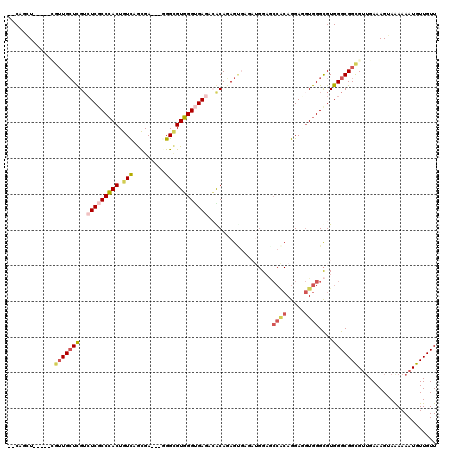
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:45:55 2006