
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 18,970,749 – 18,970,904 |
| Length | 155 |
| Max. P | 0.981512 |

| Location | 18,970,749 – 18,970,864 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.49 |
| Mean single sequence MFE | -38.88 |
| Consensus MFE | -24.07 |
| Energy contribution | -25.85 |
| Covariance contribution | 1.78 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.624718 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 18970749 115 + 27905053 GCUUUAUGAUCACU--GGGCUAGCUGCG---GCUGCUGGCAAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAU ((((..........--))))....((.(---(((((((((((.....)))))....(((..((((..((((((.(.....).))))))..))))..)))..........))))))).)). ( -38.30) >DroSec_CAF1 81832 91 + 1 -----------------------------UUGCUGCUGGCAAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUAAGCGGCCACAU -----------------------------..(((((((((((.....)))))....(((..((((..((((((.(.....).))))))..))))..)))..........))))))..... ( -31.50) >DroSim_CAF1 83632 118 + 1 GCUUUAUGAUCACU--GGGCUAGCUGUGGCUGCUGCUGGCAAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAU ((((..........--))))....((((((((((..((((((.....))))))...(((..((((..((((((.(.....).))))))..))))..)))..........)))))))))). ( -43.00) >DroEre_CAF1 82407 115 + 1 GCUUUAUGAUCACU--GAGCUAGCUGUGGCU---GCUGGCAAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAU ((((..........--))))....(((((((---((((((((.....)))))....(((..((((..((((((.(.....).))))))..))))..)))..........)))))))))). ( -43.00) >DroYak_CAF1 84942 118 + 1 GCUUUAUGAUCACU--GAGCUAGCUGUGGCUGCUGCUGGCAAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAU ((((..........--))))....((((((((((..((((((.....))))))...(((..((((..((((((.(.....).))))))..))))..)))..........)))))))))). ( -43.80) >DroAna_CAF1 84479 96 + 1 GCUUUAUGGCCGCUCAGAGCUAG------------CUGGCAAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGCUU--GGGCUCUACCAUUAGAGAGA----------GCCACAU (((...(((((.....).)))).------------..)))((((((((.(((((((.(....))))))))...))))))))--.((((((..(....)..)))----------))).... ( -33.70) >consensus GCUUUAUGAUCACU__GAGCUAGCUGUG_CUGCUGCUGGCAAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAU ...............................(((((((((((.....)))))....(((..((((..((((((.(.....).))))))..))))..)))..........))))))..... (-24.07 = -25.85 + 1.78)



| Location | 18,970,784 – 18,970,904 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.27 |
| Mean single sequence MFE | -37.17 |
| Consensus MFE | -31.04 |
| Energy contribution | -31.60 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.90 |
| SVM RNA-class probability | 0.878552 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 18970784 120 + 27905053 AAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAUAAUUAACAAAUAAUAAUGGCCAUAACGCGUGCUUUGGCCC .....(((.(((((((.(....)))))))).)))((((.(((((((((((((............))).))))(((((....((((.....))))..)))))....)))))).)))).... ( -39.00) >DroSec_CAF1 81843 120 + 1 AAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUAAGCGGCCACAUAAUUAACAAAUAAUAAUGGCCAUAACGCGUGCUUUGGCCC .....(((.(((((((.(....)))))))).)))((((.((((((.(((((....)))))............(((((....((((.....))))..)))))....)))))).)))).... ( -34.60) >DroSim_CAF1 83670 120 + 1 AAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAUAAUUAACAAAUAAUAAUGGCCAUAACGCGUGCUUUGGCCC .....(((.(((((((.(....)))))))).)))((((.(((((((((((((............))).))))(((((....((((.....))))..)))))....)))))).)))).... ( -39.00) >DroEre_CAF1 82442 119 + 1 AAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAUAAUUAACAAAUAAUAAUGGCCAUAACGCGUGCUUUGGC-C .....(((.(((((((.(....)))))))).)))((((.(((((((((((((............))).))))(((((....((((.....))))..)))))....)))))).))))..-. ( -39.00) >DroYak_CAF1 84980 119 + 1 AAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAUAAUUAACAAAUAAUAAUGGCCAUAACGCGUGCUUUGGC-C .....(((.(((((((.(....)))))))).)))((((.(((((((((((((............))).))))(((((....((((.....))))..)))))....)))))).))))..-. ( -39.00) >DroAna_CAF1 84507 107 + 1 AAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGCUU--GGGCUCUACCAUUAGAGAGA----------GCCACAUAAUUAACAAAUAAUAAUGGCCAUAACGCGUGCUUUGGC-C .........((((((((....((((.(((((((....)).(--(((((((..(....)..)))----------))).))..................))))).))))...))))))))-. ( -32.40) >consensus AAGUUUGUUGCCAAAGUCUUUUGUUUUGGCUGCACAAGUCGCGCGGCUCUGGCAUUAGAGAGAACCAUGAGCGGCCACAUAAUUAACAAAUAAUAAUGGCCAUAACGCGUGCUUUGGC_C .....(((.(((((((.(....)))))))).)))((((.((((((.(((((....)))))............(((((....((((.....))))..)))))....)))))).)))).... (-31.04 = -31.60 + 0.56)



| Location | 18,970,784 – 18,970,904 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.27 |
| Mean single sequence MFE | -38.85 |
| Consensus MFE | -32.15 |
| Energy contribution | -33.65 |
| Covariance contribution | 1.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.16 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.89 |
| SVM RNA-class probability | 0.981512 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 18970784 120 - 27905053 GGGCCAAAGCACGCGUUAUGGCCAUUAUUAUUUGUUAAUUAUGUGGCCGCUCAUGGUUCUCUCUAAUGCCAGAGCCGCGCGACUUGUGCAGCCAAAACAAAAGACUUUGGCAACAAACUU ..((((((((.(((((...((((((.((((.....))))...))))))((((.((((..........)))))))).)))))..((((.........))))..).)))))))......... ( -40.30) >DroSec_CAF1 81843 120 - 1 GGGCCAAAGCACGCGUUAUGGCCAUUAUUAUUUGUUAAUUAUGUGGCCGCUUAUGGUUCUCUCUAAUGCCAGAGCCGCGCGACUUGUGCAGCCAAAACAAAAGACUUUGGCAACAAACUU ..((((((((.(((((...((((((.((((.....))))...))))))......((((((..(....)..)))))))))))..((((.........))))..).)))))))......... ( -37.90) >DroSim_CAF1 83670 120 - 1 GGGCCAAAGCACGCGUUAUGGCCAUUAUUAUUUGUUAAUUAUGUGGCCGCUCAUGGUUCUCUCUAAUGCCAGAGCCGCGCGACUUGUGCAGCCAAAACAAAAGACUUUGGCAACAAACUU ..((((((((.(((((...((((((.((((.....))))...))))))((((.((((..........)))))))).)))))..((((.........))))..).)))))))......... ( -40.30) >DroEre_CAF1 82442 119 - 1 G-GCCAAAGCACGCGUUAUGGCCAUUAUUAUUUGUUAAUUAUGUGGCCGCUCAUGGUUCUCUCUAAUGCCAGAGCCGCGCGACUUGUGCAGCCAAAACAAAAGACUUUGGCAACAAACUU .-((((((((.(((((...((((((.((((.....))))...))))))((((.((((..........)))))))).)))))..((((.........))))..).)))))))......... ( -40.30) >DroYak_CAF1 84980 119 - 1 G-GCCAAAGCACGCGUUAUGGCCAUUAUUAUUUGUUAAUUAUGUGGCCGCUCAUGGUUCUCUCUAAUGCCAGAGCCGCGCGACUUGUGCAGCCAAAACAAAAGACUUUGGCAACAAACUU .-((((((((.(((((...((((((.((((.....))))...))))))((((.((((..........)))))))).)))))..((((.........))))..).)))))))......... ( -40.30) >DroAna_CAF1 84507 107 - 1 G-GCCAAAGCACGCGUUAUGGCCAUUAUUAUUUGUUAAUUAUGUGGC----------UCUCUCUAAUGGUAGAGCCC--AAGCUUGUGCAGCCAAAACAAAAGACUUUGGCAACAAACUU (-((((.(((....))).))))).......((((((.....((.(((----------(((..(....)..)))))))--).((....)).((((((.........))))))))))))... ( -34.00) >consensus G_GCCAAAGCACGCGUUAUGGCCAUUAUUAUUUGUUAAUUAUGUGGCCGCUCAUGGUUCUCUCUAAUGCCAGAGCCGCGCGACUUGUGCAGCCAAAACAAAAGACUUUGGCAACAAACUU ..((((((((.(((((...((((((.((((.....))))...))))))......((((((..(....)..)))))))))))..((((.........))))..).)))))))......... (-32.15 = -33.65 + 1.50)

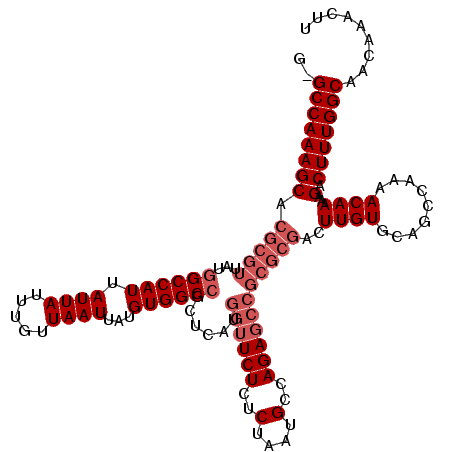

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:40:08 2006