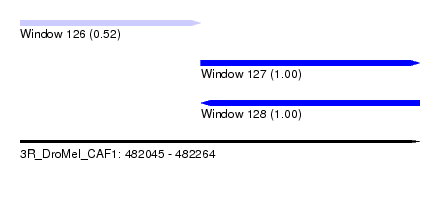
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 482,045 – 482,264 |
| Length | 219 |
| Max. P | 0.998489 |

| Location | 482,045 – 482,144 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 71.60 |
| Mean single sequence MFE | -49.02 |
| Consensus MFE | -20.27 |
| Energy contribution | -22.92 |
| Covariance contribution | 2.64 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.74 |
| Structure conservation index | 0.41 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.520021 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 482045 99 + 27905053 GCUAACGAAGGAGGGCCAG-----ACAUCGC-------------GCACGGAGGACG---AACUGGAGGAUCUUCAGCAGUUCUUUCUUGACCUGUUGGAGGCCGUACUCGCUGGCACGGU ((((.(((.......((((-----...((((-------------........).))---).)))).((.((((((((((.((......)).))))))))))))....))).))))..... ( -31.10) >DroPse_CAF1 10160 117 + 1 GCUAGUGAAGCC---ACAGCGUCCGCUGAGCUCAACGGCCGGGGAGUCGGAGGAGGAGCAGAUGCAGGAUCUGCAGCAGUUUUUCCUUGACCUCCUCGAGGCUGUGCUGGCGGGCACUGU (((.....))).---((((.(((((((.(((...(((((((((((((((.(((((((((...(((((...)))))...)))))))))))).))))))..)))))))))))))))).)))) ( -58.40) >DroGri_CAF1 6162 117 + 1 GCUGAUGCAGCC---AGAGCGUCCGCUGAGCACGAGCGCCGGGGAAACGGAGGAGGAGCAAAUGGCUGAUUUGCAACAAUUCUUUCUCGAUCUGCUCGAGGCCGUGCUGGCGGACACUGU ((((...)))).---..((.(((((((.((((((.((..((((.(....((((((((((((((.....))))))......))))))))....).))))..))))))))))))))).)).. ( -48.10) >DroMoj_CAF1 6869 117 + 1 GCUGGUUAAGCC---ACAGCGUCCGCUGAGCACCAGUGCCGGAGAGUCUGAGGAGGAGCAGAUGGCGGACUUGCAGCAGUUCUUCUUAGACCUACUUGAGGCCGUGCUGGCGGACACUGU (((.....))).---((((.(((((((.(((((..(.(((.(((.(((((((.((((((.(...(((....)))..).)))))))))))))...)))..)))))))))))))))).)))) ( -49.90) >DroAna_CAF1 11488 120 + 1 UCUGGCCAAGGAGGGCUUGCGUCCACUUAGCACCACCGCCGGGGAAUCUGAGGAGGAACAGCUUGAGGACUUGCAGCAGUUCUUUCUGGAUCUCCUGGAAGCCGUGCUUGCCGGCACAGU .((((((.(((.((((....)))).)))(((((.....(((((..((((.(((((((((.((((((....))).))).)))))))))))))..))))).....)))))....))).))). ( -48.20) >DroPer_CAF1 10437 117 + 1 GCUAGUGAAGCC---ACAGCGUCCGCUGAGCUCAACGGCCGGGGAGUCGGAGGAGGAGCAGAUGCAGGAUCUGCAGCAGUUUUUCCUUGACCUCCUCGAGGCUGUGCUGGCGGGCACUGU (((.....))).---((((.(((((((.(((...(((((((((((((((.(((((((((...(((((...)))))...)))))))))))).))))))..)))))))))))))))).)))) ( -58.40) >consensus GCUAGUGAAGCC___ACAGCGUCCGCUGAGCACAACCGCCGGGGAGUCGGAGGAGGAGCAGAUGGAGGAUCUGCAGCAGUUCUUCCUUGACCUCCUCGAGGCCGUGCUGGCGGGCACUGU (((.....))).....(((.(((((((.(((......(((.(((.((((.(((((((((...((.........))...)))))))))))))...)))..)))...)))))))))).))). (-20.27 = -22.92 + 2.64)

| Location | 482,144 – 482,264 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.14 |
| Mean single sequence MFE | -53.82 |
| Consensus MFE | -20.79 |
| Energy contribution | -21.60 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.38 |
| Mean z-score | -3.96 |
| Structure conservation index | 0.39 |
| SVM decision value | 3.11 |
| SVM RNA-class probability | 0.998471 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 482144 120 + 27905053 AGUGCAGCUGAAAUCGCCGACCUCCCAACGUGCUGGCCUAGCUUCAGAUACCGUUUCCGGGUCGGAGCAGGUGUCCAGACACAUUGUAAGUGCGGCUGAUUUCAUUGCUAGUAACCUGGU ...((((.(((((((((((..((..(((.((((((((((.(((((.(((.(((....))))))))))))))...)))).))).)))..))..)))).)))))))))))............ ( -47.50) >DroPse_CAF1 10277 114 + 1 AGAGCAGCUGAAAUCGCCGAGCACCCAACGUGCUGGCUCCGGUCCGAGCAC------CAGCUCGGAGCAGGUGUCCAAGCACAUUGUGUGCGCGGCCGAUUUCAUAGCUCGCAAUCUGGU .((((...(((((((((((.((((.(((.((((((((.(((.(((((((..------..))))))).).)).).)).))))).))).)))).))).))))))))..)))).......... ( -59.60) >DroGri_CAF1 6279 120 + 1 GGAGCAGCUGCAAUCGCCGCGCACACGACGCGCUGACGGCCUCUCGGAUGCGACCACCAGUUCCGAGCAGGUCUCCAAGCACAUUGUGUGCGCAGCGGAUUUCAUAGCGCGCAAUCUGGU ...((.((((.((((((.((((((((((.(.(((...(((((((((((.((........)))))))).)))))....))).).)))))))))).)).))))...))))..))........ ( -52.60) >DroWil_CAF1 9042 114 + 1 GGAGCAAUUGAAAUCCCCAACCACACAACGUGCUGACGCAAG------UUCCGCAUCGACUUCGGAGCAGGUGUCCAAGCACAUUGUCUGUGCGGCGGAUUUCAUUGCCCGCAAUCUGGU ((.(((((.(((((((((...((.((((.((((((((((..(------(((((.(......)))))))..)))))..))))).)))).))...)).)))))))))))))).......... ( -46.80) >DroAna_CAF1 11608 120 + 1 AGAGCAGCUAAAGUCUCCUACCACACUACGUGCUGGCGCCACUUCCGAUGCCGUUUCCGGGUCGGAACAGGUGUCCAGACACAUUGUGUGUGCGGCGGACUUUAUUGCUCGCAACCUGGU .((((((.((((((((.(..((((((...(((((((((((..(((((((.(((....))))))))))..)))).)))).)))...))))).)..).)))))))))))))).......... ( -56.80) >DroPer_CAF1 10554 114 + 1 AGAGCAGCUGAAAUCGCCGAGCACCCAACGUGCUGGCUCCGGUCCGAGCAC------CAGCUCGGAGCAGGUGUCCAAGCACAUUGUGUGCGCGGCCGAUUUCAUAGCUCGCAAUCUGGU .((((...(((((((((((.((((.(((.((((((((.(((.(((((((..------..))))))).).)).).)).))))).))).)))).))).))))))))..)))).......... ( -59.60) >consensus AGAGCAGCUGAAAUCGCCGACCACACAACGUGCUGGCGCCGGUUCGGAUACCG__UCCAGCUCGGAGCAGGUGUCCAAGCACAUUGUGUGCGCGGCGGAUUUCAUAGCUCGCAAUCUGGU ...((((((((((((((((..(((.(((.((((((((.((.............................)).)))..))))).))).)))..))).))))))))..))).))........ (-20.79 = -21.60 + 0.81)


| Location | 482,144 – 482,264 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.14 |
| Mean single sequence MFE | -57.03 |
| Consensus MFE | -28.98 |
| Energy contribution | -28.79 |
| Covariance contribution | -0.19 |
| Combinations/Pair | 1.34 |
| Mean z-score | -4.48 |
| Structure conservation index | 0.51 |
| SVM decision value | 3.12 |
| SVM RNA-class probability | 0.998489 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 482144 120 - 27905053 ACCAGGUUACUAGCAAUGAAAUCAGCCGCACUUACAAUGUGUCUGGACACCUGCUCCGACCCGGAAACGGUAUCUGAAGCUAGGCCAGCACGUUGGGAGGUCGGCGAUUUCAGCUGCACU ............(((.(((((((.((((..((..(((((((.((((...(((((((.((.(((....)))..)).).))).))))))))))))))..))..)))))))))))..)))... ( -51.50) >DroPse_CAF1 10277 114 - 1 ACCAGAUUGCGAGCUAUGAAAUCGGCCGCGCACACAAUGUGCUUGGACACCUGCUCCGAGCUG------GUGCUCGGACCGGAGCCAGCACGUUGGGUGCUCGGCGAUUUCAGCUGCUCU ..........((((..((((((((.(((.((((.(((((((((.((.(.((.(.(((((((..------..)))))))).)).)))))))))))).)))).)))))))))))...)))). ( -63.10) >DroGri_CAF1 6279 120 - 1 ACCAGAUUGCGCGCUAUGAAAUCCGCUGCGCACACAAUGUGCUUGGAGACCUGCUCGGAACUGGUGGUCGCAUCCGAGAGGCCGUCAGCGCGUCGUGUGCGCGGCGAUUGCAGCUGCUCC ........(((.(((....((((.(((((((((((.(((((((.(..(.(((.((((((....((....)).))))))))).)..)))))))).)))))))))))))))..))))))... ( -56.30) >DroWil_CAF1 9042 114 - 1 ACCAGAUUGCGGGCAAUGAAAUCCGCCGCACAGACAAUGUGCUUGGACACCUGCUCCGAAGUCGAUGCGGAA------CUUGCGUCAGCACGUUGUGUGGUUGGGGAUUUCAAUUGCUCC ..........((((((((((((((.(((..((.(((((((((((((((....).)))))....((((((...------..)))))).))))))))).))..)))))))))).))))))). ( -49.70) >DroAna_CAF1 11608 120 - 1 ACCAGGUUGCGAGCAAUAAAGUCCGCCGCACACACAAUGUGUCUGGACACCUGUUCCGACCCGGAAACGGCAUCGGAAGUGGCGCCAGCACGUAGUGUGGUAGGAGACUUUAGCUGCUCU ..........(((((.(((((((..((...(((((.(((((.((((.(.((..((((((.(((....)))..))))))..)).)))))))))).)))))...)).)))))))..))))). ( -58.50) >DroPer_CAF1 10554 114 - 1 ACCAGAUUGCGAGCUAUGAAAUCGGCCGCGCACACAAUGUGCUUGGACACCUGCUCCGAGCUG------GUGCUCGGACCGGAGCCAGCACGUUGGGUGCUCGGCGAUUUCAGCUGCUCU ..........((((..((((((((.(((.((((.(((((((((.((.(.((.(.(((((((..------..)))))))).)).)))))))))))).)))).)))))))))))...)))). ( -63.10) >consensus ACCAGAUUGCGAGCAAUGAAAUCCGCCGCACACACAAUGUGCUUGGACACCUGCUCCGAACUGGA__CGGUAUCCGAACCGGCGCCAGCACGUUGGGUGCUCGGCGAUUUCAGCUGCUCU ..........((((..(((((((.((((..(((.((((((((.(((......................................))))))))))).)))..)))))))))))...)))). (-28.98 = -28.79 + -0.19)

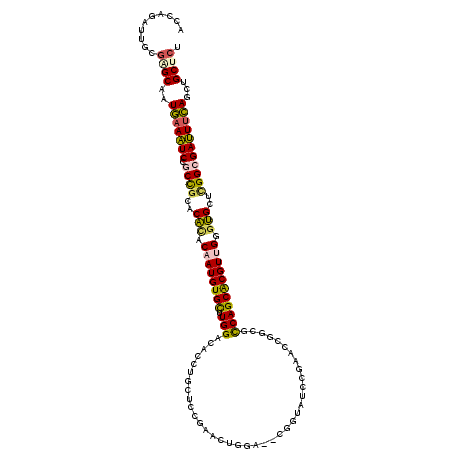
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:32:18 2006