
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 18,667,800 – 18,667,921 |
| Length | 121 |
| Max. P | 0.579488 |

| Location | 18,667,800 – 18,667,902 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 81.56 |
| Mean single sequence MFE | -21.38 |
| Consensus MFE | -13.95 |
| Energy contribution | -12.70 |
| Covariance contribution | -1.25 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.535641 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
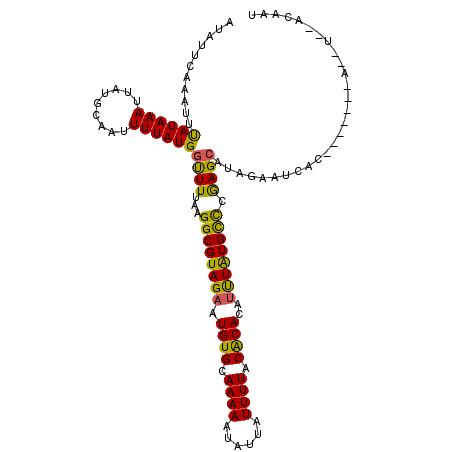
>3R_DroMel_CAF1 18667800 102 - 27905053 AUAUUCAAAUUUAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACAUUUGUGCUCGAGCAUGGAAUCAC-------------ACAAU ....................((((((((((((.(.....).)))))))).))))....................((..(((....)))..)).....-------------..... ( -15.30) >DroVir_CAF1 231487 110 - 1 AUAUUCAAAUUUAUAAAUUAUGAAAUUUUAUGGCUUUGAGGCGUAGUAUGUGCAAAAAUAUUAUUUUACACACGUCUAUGCCCAAGCAUAUUUUCAU-----GCAUUUGCGCAGU .............((((((((((((...((((.(((.(.(((((((..((((.((((......)))).))))...))))))))))))))).))))))-----).)))))...... ( -25.80) >DroGri_CAF1 97239 110 - 1 AUAUUCAAAUUUAUAAAUUAUGAAAUUUUAUGCCUUUGAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACGUCUAUGCCCAAGCAUAUUUUCAU-----GCAUUUGCUGAGU ........(((((((((((((((((...(((((..(((.((((((((.((((.((((......)))).))))..)))))))))))))))).))))))-----).))))).))))) ( -31.00) >DroEre_CAF1 100715 102 - 1 AUAUUCAAAUUUAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACAUUUGUGCCCGAGCACGGCAUCGC-------------CCAAA ..............((((((((......))))))))...((((..(((((((...(((......)))...)))))))(((((.......))))))))-------------).... ( -17.60) >DroYak_CAF1 98194 113 - 1 AUAUUCAAAUUUAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACAUUUGUGCUCGAGCCGAGCAUCGGACAUCGCAUUU--GCAAU ....................(((((.((((......))))(((..(((((((...(((......)))...)))))))(((((((...))))))).......)))..))--))).. ( -20.30) >DroAna_CAF1 93102 104 - 1 AUAUUCAAAUUCAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGGAAAAAUAUUAUUUUGCGCACGUUUAUGUUCGAGCAUGAAAACAC-------A--U--ACAAU ......(((((((((...)))).)))))(((((((((...((((((((((...........)))))))))).....(((((....)))))))))).)-------)--)--).... ( -18.30) >consensus AUAUUCAAAUUUAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACAUUUAUGCCCGAGCAUAGAAUCAC_______A__U__ACAAU ...........((((((.........))))))((((...((((((((.((((.((((......)))).))))..)))))))).))))............................ (-13.95 = -12.70 + -1.25)

| Location | 18,667,825 – 18,667,921 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 81.02 |
| Mean single sequence MFE | -19.30 |
| Consensus MFE | -13.18 |
| Energy contribution | -13.73 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.38 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.579488 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 18667825 96 - 27905053 --GC--------C---------UGCUUUUAUGUGUGAGAUAUUC--AAAUUUAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACAUUU --..--------.---------........(((((((((((...--.((((....)))).((((((((((((.(.....).)))))))).)))).......)))))))))))..... ( -18.60) >DroVir_CAF1 231520 101 - 1 --GC--------UUUU----GAAGCAUUUCUGUGUGGGAUAUUC--AAAUUUAUAAAUUAUGAAAUUUUAUGGCUUUGAGGCGUAGUAUGUGCAAAAAUAUUAUUUUACACACGUCU --((--------(...----..))).....((((((((((((((--(((.(((((((.........))))))).))))))(((.......)))........)))))))))))..... ( -19.20) >DroGri_CAF1 97272 105 - 1 --GC--------CUUUAUUUUGUGUAUUUGUGUGUGAGAUAUUC--AAAUUUAUAAAUUAUGAAAUUUUAUGCCUUUGAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACGUCU --..--------.................(((((((((((((((--(((((....)))).))))((((((((((.....))))))))))............)))))))))))).... ( -26.80) >DroWil_CAF1 98288 108 - 1 AUACACACCAAAC---------UCUAUUUCUGUUUGAGAUAUUCCAAAAAUUAUAAAAUAUGCAAUUUUAUGGCCUUAAGGCGUUGAAUGUGCAAAAAUAUUAUUUUAUACACGUUC .............---------..((((((.....))))))..........(((((((((((((..((((..(((....)))..))))..)))).......)))))))))....... ( -16.41) >DroAna_CAF1 93129 95 - 1 --GC--------C---------U-CUUUUAUGUGUGAGAUAUUC--AAAUUCAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGGAAAAAUAUUAUUUUGCGCACGUUU --..--------.---------.-......((((..(((((...--...((((((..((((((....(((......))).))))))..)))))).......)))))..))))..... ( -18.12) >DroPer_CAF1 112785 96 - 1 --GC--------A---------CCCCUUUAUGUGUGAGAUAUUC--AAAUUUAUAAAUUAUGCAAUUUUAUGGUUUUAAGGCGUAGAAUGUGCAAAAAUAUUAUUUUAUACACGUUU --..--------.---------........(((((((((((...--.((((....)))).((((((((((((.(.....).)))))))).)))).......)))))))))))..... ( -16.70) >consensus __GC________C_________UGCAUUUAUGUGUGAGAUAUUC__AAAUUUAUAAAUUAUGCAAUUUUAUGGCUUUAAGGCGUAGAAUGUGCAAAAAUAUUAUUUUACACACGUUU ..............................(((((((((((......((((....)))).((((((((((((.(.....).)))))))).)))).......)))))))))))..... (-13.18 = -13.73 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:36:39 2006