
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 18,308,851 – 18,309,092 |
| Length | 241 |
| Max. P | 0.995437 |

| Location | 18,308,851 – 18,308,950 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 81.08 |
| Mean single sequence MFE | -40.58 |
| Consensus MFE | -28.84 |
| Energy contribution | -29.20 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.669868 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
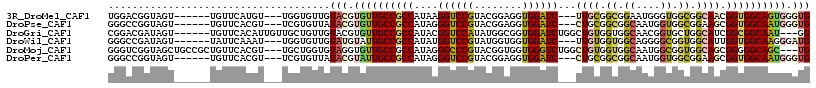
>3R_DroMel_CAF1 18308851 99 - 27905053 CGCCCGGACCCACCCAUGCCGCCACCAGCUAUAUAAAUAUCAUAGGGCGUGGUUUCCCGCUCGGUGACCGUACUCCGGAUCUCCGGUGGCGGUGUGGCU .(((.((......))(((((((((((..................(((((.((...)))))))((.(((((.....))).)).)))))))))))))))). ( -43.20) >DroPse_CAF1 21008 99 - 1 CUGCCAGAACCACCCAUGCCACCUCCGGCCACGUAGAUAUCGAAGGGCGUUGUUUCCCGCUCGGUGACAGUGCUCCUAAUCUCCGGUGGUGGCGUGGCC .............((((((((((.((((...((.......)).(((((((((((.((.....)).)))))))).))).....)))).)))))))))).. ( -40.60) >DroSim_CAF1 21201 99 - 1 CGACCGGACCCACCCAUGCCGCCACCAGCGAUAUAAACAUCAUAGGGCGUGGUGUCCCGCUCGGUGACCGUACUCCGGAUCUCCGGAGGCGGUGUGGCC .....((......))..(((((((((((((......(((((((.....)))))))..)))).))))(((((.((((((....)))))))))))))))). ( -43.30) >DroEre_CAF1 20725 99 - 1 CGCCCGGAGCCGCCCAUGCCGCCACCAGCGACAUACACAUCAUAGGGCGUGGUGUCCCGCUCGGUAACCGUACUUCGGAUCUCCGGAGGCGGUGUGGCC .((((.((((.(((((((((((.....))((........))....))))))).))...)))).)..(((((.((((((....)))))))))))..))). ( -40.20) >DroAna_CAF1 21408 99 - 1 CGACCGGAGCCGCCCAUUCCACCGCCUGCAAUGUACACGUCGUACGGCGUGGUCUCCCUCUCCGUGACUGUACUUCGGAUUUCCGGAGGCGGCGUAGCC .....((.((((((..................(((((.(((..((((.(.((....)).).))))))))))))(((((....)))))))))))....)) ( -35.60) >DroPer_CAF1 21422 99 - 1 CUGCCAGAACCACCCAUGCCACCUCCGGCCACGUAGAUAUCGAAGGGCGUUGUUUCCCGCUCGGUGACAGUGCUCCUAAUCUCCGGUGGUGGCGUGGCC .............((((((((((.((((...((.......)).(((((((((((.((.....)).)))))))).))).....)))).)))))))))).. ( -40.60) >consensus CGACCGGAACCACCCAUGCCACCACCAGCCACAUAAACAUCAUAGGGCGUGGUUUCCCGCUCGGUGACCGUACUCCGGAUCUCCGGAGGCGGCGUGGCC .............((((((((((.((..................(((((........)))))((.(((((.....))).)).)))).)))))))))).. (-28.84 = -29.20 + 0.36)


| Location | 18,308,950 – 18,309,061 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 75.82 |
| Mean single sequence MFE | -47.32 |
| Consensus MFE | -30.08 |
| Energy contribution | -29.78 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.69 |
| Structure conservation index | 0.64 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.901004 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 18308950 111 - 27905053 GUGUUGCCGCCAUAAGGUCCGUACGGAGGUGGAUC---UUGCGGCGGAAUGGGUGGCGGCAACGGUGGCAGUGGGUGGUGCUCGUCCGGCAGCUGCAUGCGAUCGUGAUCAU---UU ..(((((((((((....(((((.((.(((.....)---)).)))))))....)))))))))))(((.((((..(.((.((......)).)).)..).))).)))........---.. ( -49.90) >DroGri_CAF1 24819 111 - 1 GUGUUGCCGCCAUACGGUCCAUAUGGCGGUGGAUCUGGCUGUGGUGGCAACGGUGCUGGCAUCGGCGGCAAU---GGAUUCUCAUCGGGCAACUGUAUACGCUCAUGAUCAU---GA ..((((((((((((.(((((((......)))))))....))))))))))))(.(((((....))))).).((---((....((((.((((..........))))))))))))---.. ( -42.00) >DroWil_CAF1 28454 111 - 1 GUAUUGCCGCCAUAUGGUCCGUAUGGUGGUGGAUC---UUGUGGUGGCAGGGGCGGUGGCAUUGGUGGCAAGGGAUGAUGCUCCUCCGGCAAUUGCAUGUUCUCGUGAUCGA---UG ...(((((((((((.((((((........))))))---.)))))))))))(((((...((((((.(((...((((......))))))).))).))).)))))(((....)))---.. ( -42.70) >DroMoj_CAF1 22157 111 - 1 GUGUUGCCGCCAUAGGGCCCGUACGGUGGUGGGUCUGGCUGUGGUGGCAAUGGCGGUGGCAGCGGGGGCAGC---UGGUGCUCGUCCGGCAGCUGCAUACGCUCGUGAUCUC---GA ..(((((((((((((((((((........))))))...))))))))))))).(.(((.((((((...(((((---((.((......)).)))))))...)))).)).))).)---.. ( -55.70) >DroAna_CAF1 21507 114 - 1 GUAUUUCCGCCGUAAGGACCAUAUGGCGGUGGAUC---CUGUGGCGGUAGUGGAGGUGGUAGUGGGGGCAUUGGAUGGUGUUCGUCCGGCAGCUGCAUGCGAUCAUGAUCAUGGCUG ..(((.(((((...((((((((......)))).))---))..))))).)))((..(((((.((((..((((((((((.....))))))((....))))))..)))).)))))..)). ( -40.80) >DroPer_CAF1 21521 111 - 1 GUAUUGCCGCCAUAGGGUCCGUACGGAGGUGGAUC---CUGCGGCGGCAAUGGUGGCGGAAGCGGUGGCAAUGGGUGGUGCUCGUCCGGCAGCUGCAUGCGAUCGUGAUCGU---UC .((((((((((.(((((((((........))))))---))).))))))))))..(((((..(((((.(((....(..(((((.....)))).)..).))).)))))..))))---). ( -52.80) >consensus GUAUUGCCGCCAUAAGGUCCGUACGGAGGUGGAUC___CUGUGGCGGCAAUGGUGGUGGCAGCGGUGGCAAUGG_UGGUGCUCGUCCGGCAGCUGCAUGCGAUCGUGAUCAU___UA .(((((((((((((.((((((........))))))....)))))))))))))(((((.((..(((.((((........))))...))))).)))))..................... (-30.08 = -29.78 + -0.30)



| Location | 18,308,987 – 18,309,092 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 77.43 |
| Mean single sequence MFE | -48.13 |
| Consensus MFE | -27.52 |
| Energy contribution | -27.50 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.44 |
| Mean z-score | -3.52 |
| Structure conservation index | 0.57 |
| SVM decision value | 2.58 |
| SVM RNA-class probability | 0.995437 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 18308987 105 - 27905053 UGGACGGUAGU------UGUUCAUGU---UGGUGUUGUACGUGUUGCCGCCAUAAGGUCCGUACGGAGGUGGAUC---UUGCGGCGGAAUGGGUGGCGGCAACGGUGGCAGUGGGUG ((((((.....------))))))..(---((.(((..(...((((((((((((....(((((.((.(((.....)---)).)))))))....)))))))))))))..))).)))... ( -41.70) >DroPse_CAF1 21144 105 - 1 GGGCCGGUAGU------UGUUCACGU---UCGUGUUAUACGUGUUGCCGCCAUAGGGUCCGUACGGAGGUGGAUC---CUGCGGCGGCAAUGGUGGCGGAAGCGGUGGCAAUGGGUG ..(((....((------((((..(((---(..((((((....(((((((((.(((((((((........))))))---))).))))))))).))))))..))))..)))))).))). ( -47.50) >DroGri_CAF1 24856 108 - 1 CGGACGAUAGU------UGUUCACAUUGUUGCUGUUGUACGUGUUGCCGCCAUACGGUCCAUAUGGCGGUGGAUCUGGCUGUGGUGGCAACGGUGCUGGCAUCGGCGGCAAU---GG .((((((...)------))))).(((((((((((..((((.(((((((((((((.(((((((......)))))))....)))))))))))))))))......))))))))))---). ( -52.20) >DroWil_CAF1 28491 105 - 1 GGGCCGAUAGU------UAUUCAAAU---UGGUGUUGUAUGUAUUGCCGCCAUAUGGUCCGUAUGGUGGUGGAUC---UUGUGGUGGCAGGGGCGGUGGCAUUGGUGGCAAGGGAUG ..(((((....------...))..((---..((((..(.(((.(((((((((((.((((((........))))))---.)))))))))))..))))..))))..)))))........ ( -41.90) >DroMoj_CAF1 22194 111 - 1 GGGUCGGUAGCUGCCGCUGUUCACGU---UGCUGGUGUAGGUGUUGCCGCCAUAGGGCCCGUACGGUGGUGGGUCUGGCUGUGGUGGCAAUGGCGGUGGCAGCGGGGGCAGC---UG .....(((....)))(((((((.(((---(((((.(((....(((((((((((((((((((........))))))...))))))))))))).))).)))))))).)))))))---.. ( -57.90) >DroPer_CAF1 21558 105 - 1 GGGCCGGUAGU------UGUUCACGU---UCGUGUUAUACGUAUUGCCGCCAUAGGGUCCGUACGGAGGUGGAUC---CUGCGGCGGCAAUGGUGGCGGAAGCGGUGGCAAUGGGUG ..(((.((.((------..(((((..---.((((...))))((((((((((.(((((((((........))))))---))).)))))))))))))((....))))..)).)).))). ( -47.60) >consensus GGGCCGGUAGU______UGUUCACGU___UGGUGUUGUACGUGUUGCCGCCAUAGGGUCCGUACGGAGGUGGAUC___CUGCGGCGGCAAUGGUGGCGGCAGCGGUGGCAAUGGGUG .....................................(((.((((((((((....((((((........))))))...((((.((.((....)).)).)))).)))))))))).))) (-27.52 = -27.50 + -0.02)


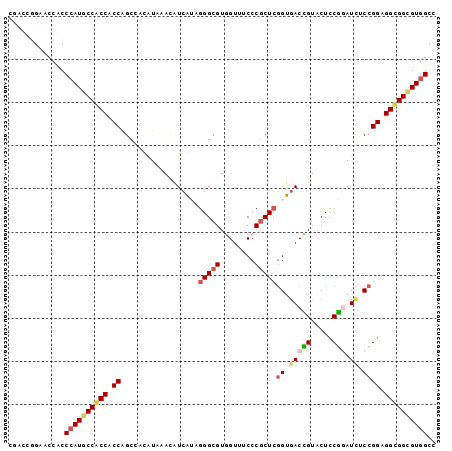
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:49 2006