
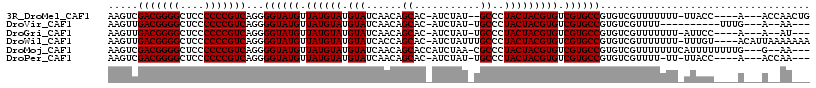
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 18,299,444 – 18,299,550 |
| Length | 106 |
| Max. P | 0.509019 |

| Location | 18,299,444 – 18,299,550 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 86.24 |
| Mean single sequence MFE | -29.53 |
| Consensus MFE | -26.55 |
| Energy contribution | -26.55 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.90 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.509019 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
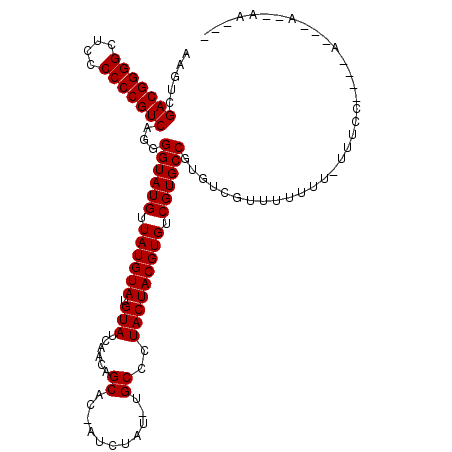
>3R_DroMel_CAF1 18299444 106 + 27905053 AAGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC-AUCUAU--GCCCUACUACGUGUCGUGCCGUGUCGUUUUUUU-UUACC----A---ACCAACUG ....(((((.(((.(..(.(((..((((((((..((((.(((....)))..))-)).)))--)))))...))).)..).))).))))).......-.....----.---........ ( -30.30) >DroVir_CAF1 11533 97 + 1 AAGUUGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC-AUCUAU-UGCCCUACUACGUGUCGUGCCGUGUCGUUUU----------UUUG---A--AA--- ...(((((((((....))))))))).((((((.((((((.(((.(((.((...-..)).)-))...))))))))).))))))..........----------....---.--..--- ( -29.40) >DroGri_CAF1 12751 102 + 1 AAGUUGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC-AUCUAU-UGCCCUACUACGUGUCGUGCCGUGUCGUUUUUUU-AUUCC----A---A--AU--- ...(((((((((....))))))))).((((((.((((((.(((.(((.((...-..)).)-))...))))))))).)))))).............-.....----.---.--..--- ( -29.40) >DroWil_CAF1 17036 111 + 1 AAGUUGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCACCAGCAC-AUCUAUUUGCCCUACUACGUGUCGUGCCGUGUCGUUUUUUU-UUUGU----ACAUUAAAAAAA ...(((((((((....))))))))).((((((.((((((.(((......(((.-.......)))..))))))))).)))))).........((((-((((.----....)))))))) ( -31.20) >DroMoj_CAF1 11850 108 + 1 AAGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCACCAUCUAA-CGCCCUACUACGUGUCGUGCCGUGUCGUUUUUUUCAUUUUUUUUG---G--AA--- .....(((((((....)))))))...((((((.((((((.(((......((.........-.))..))))))))).))))))..........(((((.......))---)--))--- ( -27.40) >DroPer_CAF1 11638 103 + 1 AAGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC-AUCUAU-UGCCCUACUACGUGUCGUGCCGUGUCGUUUU-UU-UUACC----A---ACCAA--- .....(((((((....))))))).((((((((.((((((.(((.(((.((...-..)).)-))...))))))))).))))))(((.......-..-.))).----.---.))..--- ( -29.50) >consensus AAGUCGACGGGGCUCCCCCCGUCAGGGGUAUGUUAUGUAUGUAUCAACAGCAC_AUCUAU_UGCCCUACUACGUGUCGUGCCGUGUCGUUUUUUU_UUUCC____A___A__AA___ .....(((((((....)))))))...((((((.((((((.(((......((...........))..))))))))).))))))................................... (-26.55 = -26.55 + 0.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:46 2006